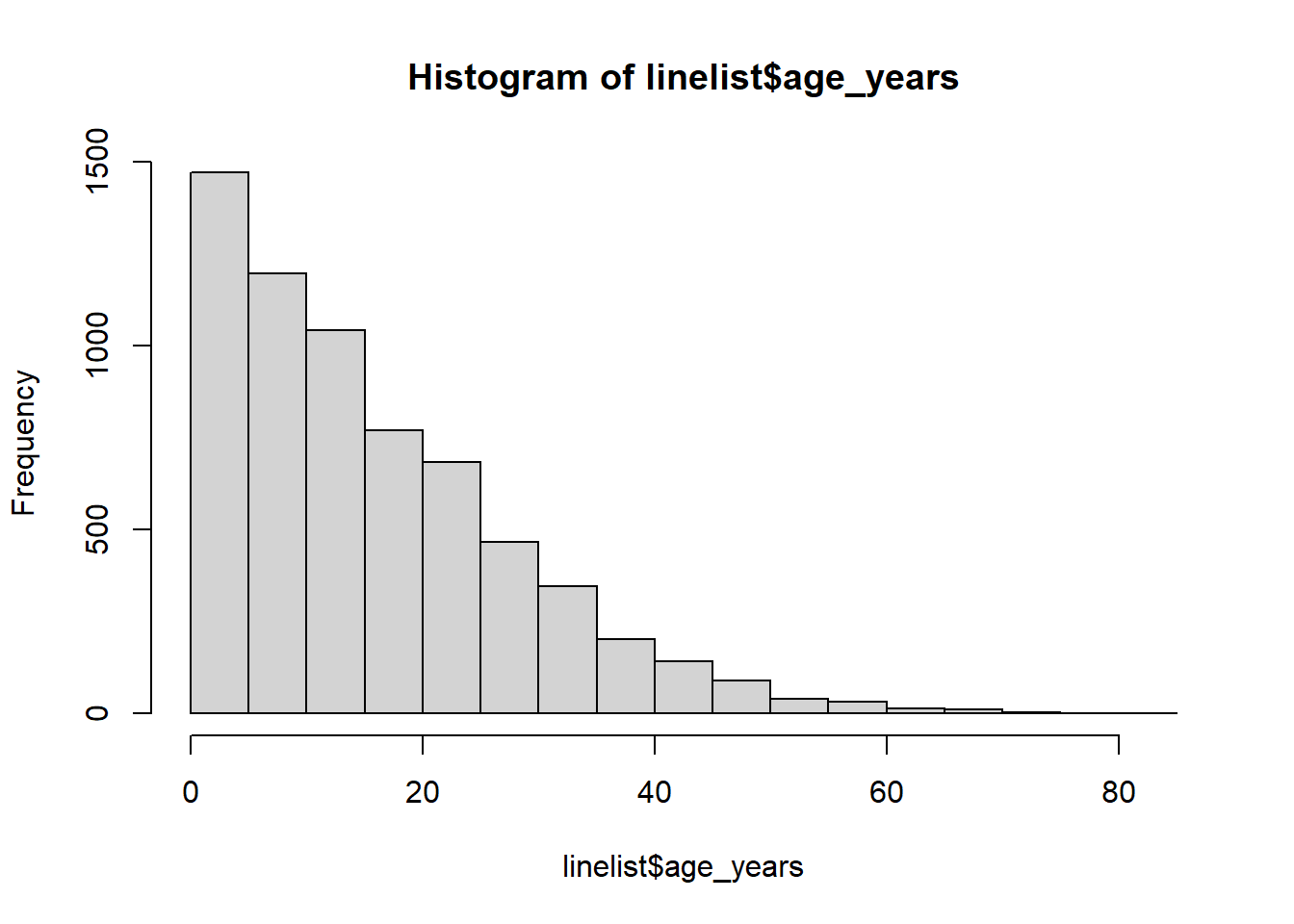8 データクリーニングと主要関数
この章では、データクリーニング(データの前処理)の一般的な手順を示し、R を使ったデータ管理に不可欠な機能について説明します。
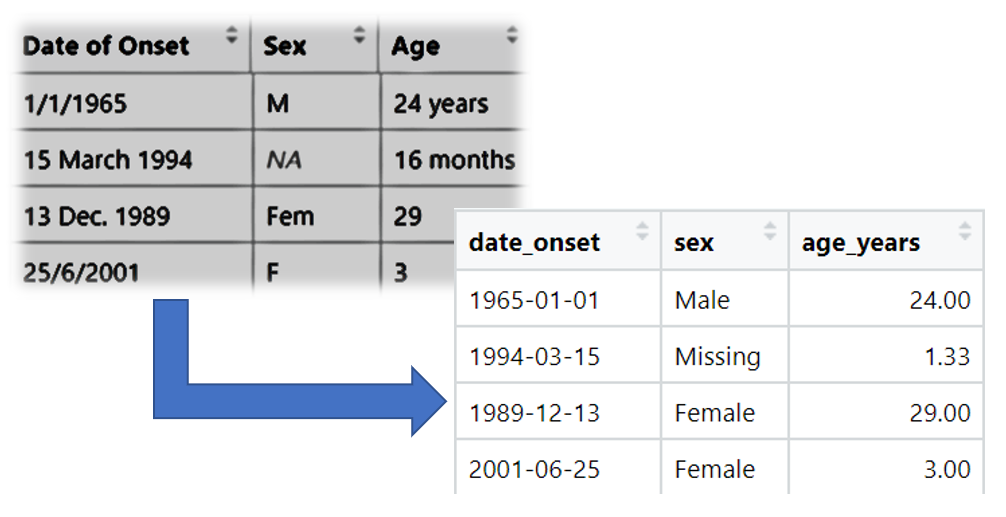
例として、未加工のラインリスト(症例データ)を使用し、ラインリストのインポートを始め、データクリーニングの手順を段階的に解説していきます。上の図のような処理は、R コードでは「パイプ(“pipe”)」チェーンとして行われ、「パイプ」演算子 %>%によって、対象のデータセットをある操作から次の操作に渡すことができます。
主要関数
このハンドブックでは、R パッケージの一つである tidyverse 系関数を主に使用します。以下の表は、本章で使用する基本の R パッケージおよびパッケージに含まれる関数の一覧です。
関数の多くは、データ操作に関する課題を解決するための「動詞」関数を提供する R パッケージ dplyr に属しています(dplyr という名前は”data frame-plier” にちなんでいます)。dplyr パッケージは、ggplot2、tidyr、stringr、tibble、purrr、magrittr、forcats などのパッケージを含む R パッケージである tidyverse ファミリーの一部です。
| 関数 | 機能 | パッケージ |
|---|---|---|
%>% |
ある関数から次の関数へデータを「パイプで渡す」 | magrittr |
mutate() |
列の作成、変換、再定義 | dplyr |
select() |
列の保持、削除、選択、列名の変更 | dplyr |
rename() |
列名の変更 | dplyr |
clean_names() |
列名の構文を標準化 | janitor |
as.character(), as.numeric(), as.Date() 等 |
列のデータ型を変換 | base R |
across() |
複数の列を一度に変換 | dplyr |
| tidyselect 系関数 | 論理条件を使った列の選択 | tidyselect |
filter() |
特定の行を選択 | dplyr |
distinct() |
重複した行の削除 | dplyr |
rowwise() |
各行の中の操作、または各行による操作 | dplyr |
add_row() |
手動で行を追加 | tibble |
arrange() |
行の並べ替え | dplyr |
recode() |
列の値を再定義 | dplyr |
case_when() |
複数の論理基準を使用し列値を再コーディング | dplyr |
replace_na(), na_if(), coalesce()
|
再コーティングのための専用関数 | tidyr |
age_categories(), cut()
|
数値列からカテゴリグループを作成 | epikit, base R |
match_df() |
データ辞書を使用した値の再定義・処理 | matchmaker |
which() |
論理的基準を適用し、インデックスを抽出 | base R |
これらの関数を Stata や SAS のコマンドと比較したい場合は、Excel・Stata・SASとの比較 の章を参照にしてください。
また、R パッケージの data.table では、:= のような演算子や角括弧 [ ] が頻繁に使用されており、別の形式のデータ管理フレームワークを目にすることがあるかもしれません。この手法と構文については、データテーブル の章で簡単に説明しています。
用語体系
このハンドブックでは、一般的に「変数」や「観測値」の代わりに「列」や「行」を使用します。“tidy data” の入門書で説明されているように、疫学統計データセットのほとんどは、行、列、値の構造で構成されています。
「変数」は、同じ基本属性について測定された値(年齢層、アウトカム、発症日など)を示しています。「観測値」は、同じ単位(人、場所、ラボのサンプルなど)で測定されたすべての値を示します。そのため、これらの値を明確に定義することは困難です。
“tidy” データセット(綺麗に整理されたデータセット)では、各列が変数、各行が観測値、そして各セルが 1 つの値となっています。しかし、この型に当てはまらないデータセットもあります。“wide” 型(横型)のデータセットでは、1 つの変数が複数の列に分かれて記録されていることがあります(詳細は、データの縦横変換 の章で紹介する例をご参照ください)。同様に、観測値が複数の行に分かれていることもあります。
このハンドブックでは、主にデータの管理と変換について取り扱うため、抽象的な観測値や変数よりも、行や列といった具体的なデータ構造を示す呼び方を使用していきます。例外として、データ分析の章では、変数や観測値と呼ぶことがあります。
8.1 パイプラインによるデータクリーニング
この章では、代表的なデータクリーニングの手順を実行し、パイプに処理を順次追加しながら進めていきます。
疫学的な分析やデータ処理では、クリーニングを行う際、複数の手順が連続して実行されることが多く、手順どうしが互いに関連していることがよくあります。Rでは多くの場合、このように連続した手順はクリーニングを行う一貫したパイプラインとして作成され、データが処理されます。作成されたパイプラインでは、未加工のデータセットが 1 つのデータ処理のステップから別のデータ処理のステップに渡されます。
このような連続処理には、dplyr パッケージの「動詞」関数と magrittr パッケージのパイプ演算子 %>% が使用されます。パイプ処理は、始めに未加工データ(“linelist_raw.xlsx”)を扱い、最終的に、使用や保存、エクスポートなどが行えるクリーニングされた R データフレーム(linelist)を作成することを目標としています。
パイプラインのデータ処理は、各手順の順番が重要です。クリーニングの手順には以下のようなものがあります。
データをインポートする
列名を修正、または変更する
重複したデータを排除する
列を作成し、変換する(例:値の再定義または標準化)
行を抽出、または追加する
8.2 パッケージの読み込み
以下のコードを実行すると、分析に必要なパッケージが読み込まれます。このハンドブックでは、パッケージを読み込むために、pacman パッケージの p_load() を主に使用しています。p_load() は、必要に応じてパッケージをインストールし、現在の R セッションで使用するためにパッケージを読み込む関数です。また、すでにインストールされたパッケージは、R の基本パッケージである base の library() を使用して読み込むこともできます。R パッケージに関する詳細は、R の基礎 の章をご覧ください。
pacman::p_load(
rio, # データを読み込むためのパッケージ
here, # 相対ファイルパスを設定するためのパッケージ
janitor, # データ前処理と表の作成のためのパッケージ
lubridate, # 日付操作のためのパッケージ
matchmaker, # データ辞書に基づいたデータ前処理のためのパッケージ
epikit, # age_categories()を含むパッケージ
tidyverse # データ管理と可視化のためのパッケージ
)8.3 データのインポート
データをインポートする
ここでは、rio パッケージの import() を使って、未加工の症例リスト(Excel ファイル)をインポートします。rio パッケージは、様々な形式のファイル(.xlsx、.csv、.tsv、.rds など)に対応できます。rio パッケージの詳細や、通常とは異なる状況(行のスキップ、欠損値の設定、Googleシートのインポートなど)での対処法を知りたい方は、インポートとエクスポート の章をご覧ください。
お手元の環境でこの章の内容を実行したい方は、こちら から「未加工の」ラインリストをダウンロードしてください(.xlsx ファイルとしてダウンロードされます)。
データサイズが大きいためにインポートに時間がかかる場合は、インポートするためのコードを、インポート後のデータ前処理を行うパイプラインとは別の場所に書き、未加工データを元データとしてオブジェクトに保存しておくと便利です。コードを分けて書くことで、未加工のデータと前処理済みのデータの比較が容易になります。
linelist_raw <- import("linelist_raw.xlsx")データフレームの最初の 50 行を確認してみましょう。
注釈:base R に含まれている関数 head(n) を使うと、R コンソールに最初の n 行が表示されます。
インポートしたデータを確認する
skimr パッケージの skim() を使うと、データフレーム全体の概要を把握することができます(詳しくは、記述統計表の作り方 を参照ください)。列は、文字値や数値などのデータ型別にまとめられています。
注釈:“POSIXct” は未加工の日付データ型の一つです(詳細は、日付型データ の章をご覧ください)。
skimr::skim(linelist_raw)| Name | linelist_raw |
| Number of rows | 6611 |
| Number of columns | 28 |
| _______________________ | |
| Column type frequency: | |
| character | 17 |
| numeric | 8 |
| POSIXct | 3 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| case_id | 137 | 0.98 | 6 | 6 | 0 | 5888 | 0 |
| date onset | 293 | 0.96 | 10 | 10 | 0 | 580 | 0 |
| outcome | 1500 | 0.77 | 5 | 7 | 0 | 2 | 0 |
| gender | 324 | 0.95 | 1 | 1 | 0 | 2 | 0 |
| hospital | 1512 | 0.77 | 5 | 36 | 0 | 13 | 0 |
| infector | 2323 | 0.65 | 6 | 6 | 0 | 2697 | 0 |
| source | 2323 | 0.65 | 5 | 7 | 0 | 2 | 0 |
| age | 107 | 0.98 | 1 | 2 | 0 | 75 | 0 |
| age_unit | 7 | 1.00 | 5 | 6 | 0 | 2 | 0 |
| fever | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| chills | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| cough | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| aches | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| vomit | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| time_admission | 844 | 0.87 | 5 | 5 | 0 | 1091 | 0 |
| merged_header | 0 | 1.00 | 1 | 1 | 0 | 1 | 0 |
| …28 | 0 | 1.00 | 1 | 1 | 0 | 1 | 0 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 |
|---|---|---|---|---|---|---|---|---|---|
| generation | 7 | 1.00 | 16.60 | 5.71 | 0.00 | 13.00 | 16.00 | 20.00 | 37.00 |
| lon | 7 | 1.00 | -13.23 | 0.02 | -13.27 | -13.25 | -13.23 | -13.22 | -13.21 |
| lat | 7 | 1.00 | 8.47 | 0.01 | 8.45 | 8.46 | 8.47 | 8.48 | 8.49 |
| row_num | 0 | 1.00 | 3240.91 | 1857.83 | 1.00 | 1647.50 | 3241.00 | 4836.50 | 6481.00 |
| wt_kg | 7 | 1.00 | 52.69 | 18.59 | -11.00 | 41.00 | 54.00 | 66.00 | 111.00 |
| ht_cm | 7 | 1.00 | 125.25 | 49.57 | 4.00 | 91.00 | 130.00 | 159.00 | 295.00 |
| ct_blood | 7 | 1.00 | 21.26 | 1.67 | 16.00 | 20.00 | 22.00 | 22.00 | 26.00 |
| temp | 158 | 0.98 | 38.60 | 0.95 | 35.20 | 38.30 | 38.80 | 39.20 | 40.80 |
Variable type: POSIXct
| skim_variable | n_missing | complete_rate | min | max | median | n_unique |
|---|---|---|---|---|---|---|
| infection date | 2322 | 0.65 | 2012-04-09 | 2015-04-27 | 2014-10-04 | 538 |
| hosp date | 7 | 1.00 | 2012-04-20 | 2015-04-30 | 2014-10-15 | 570 |
| date_of_outcome | 1068 | 0.84 | 2012-05-14 | 2015-06-04 | 2014-10-26 | 575 |
8.4 列名
Rでは、データセットの「ヘッダー」または各列の「一番上」の値が列名になります。列名は、コード内で列を参照するために使用されるほか、図を作成する際はデフォルトのラベルとして使用されます。
SAS や STATA など他の統計ソフトでは、短い列名を具体的に説明する「ラベル」が列名と共に使用されることが多いですが、R ではほとんど使用されません(R でもデータに列ラベルを追加することは可能です)。列名を図の表示に合わせて変更したい場合は、通常、図をプロットするコードの中で調整します(例えば、図の軸や凡例のタイトル、表示される表のヘッダーなどを調整したい場合です。詳細は、ggplot のヒントの章の 31.2 スケールと見やすい表の作り方 の章を参照してください)。データに列ラベルを付けたい場合は、こちら と こちら のページをご覧ください。
R の列名はデータ分析やデータ管理において非常に頻繁に使用されるため、「きれいに」整えられている必要があります。列名を設定する際の一般的なルールとして、以下のように提案します。
短い名前にする
スペースを使わない(スペースが必要な場合は、アンダースコア _ に置き換えてください)
&、#、< > などの特殊な文字を使用しない
各列似たようなスタイルの用語を使用する(例:異なる種類の日付データを含む列が複数ある場合に、date_onset、date_report、date_death とするなど)
以下に、base R の names() を使用して linelist_raw の列名を表示しました。表示された列名には、次のような特徴がみられます。
スペースを含む名前がある(例:
infection date)日付データを含む列が複数あるが、各列異なるルールで名付けられている (例:
date onsetとinfection date)最後の 2 列の名前(“merged_header” と “…28”)を見ると、ダウンロードされた元の .xlsx ファイルの最後の 2 列には、マージされたヘッダーがあったことがわかります。2 つの列がマージされてできた列が R によって分割され、マージされてできた列の名前(“merged_header”)は元々の最初の列に割り当てられ、元々の 2 番目の列にはセルを保持するために一時的に “…28” が割り当てられています(マージされる前は空欄の列であり、28番目の列であるため)。
names(linelist_raw) [1] "case_id" "generation" "infection date" "date onset"
[5] "hosp date" "date_of_outcome" "outcome" "gender"
[9] "hospital" "lon" "lat" "infector"
[13] "source" "age" "age_unit" "row_num"
[17] "wt_kg" "ht_cm" "ct_blood" "fever"
[21] "chills" "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header" "...28" 注釈:スペースを含む列名を参照するには、その名前をバックティック(`)で囲みます。例えば、linelist$` '\x60infection date\x60'` というように使用します。キーボードでは、バックティック(`)とシングルクォーテーションマーク(’)が異なることに注意してください。
自動クリーニング
janitor パッケージの clean_names() は、以下のように列名を標準化し、ユニークにします。
すべての列名をアンダースコア(_)、数字、文字のみで構成されるように変換します。
アクセント記号付きの文字は ASCII に音訳されます(例:ドイツ語のウムラウト記号付きの o は単に “o” に変換され、スペイン語の “enye”は “n”に変換されます)。
変換後の列名の大文字と小文字の区別は、
case =引数を使って指定できます(デフォルトは “snake” になっていますが、他に “sentence”、“title”、“small_camel” などがあります)。replace =引数にベクトルを与えることで、特定の名前の置き換えを指定することができます(例:replace = c(onset = "date_of_onset"))clean_names()に関する公式ドキュメントは、こちらをご覧ください。
では、前処理のパイプラインを作成していきます。まず初めに、以下では、未加工のラインリストに対して clean_names() を使用し、列名を整えていきます。
# 未加工のデータセットをclean_names()にパイプし、出力結果をlinelistとして保存する
linelist <- linelist_raw %>%
janitor::clean_names()
# 新しく作成された linelist の列名を確認する
names(linelist) [1] "case_id" "generation" "infection_date" "date_onset"
[5] "hosp_date" "date_of_outcome" "outcome" "gender"
[9] "hospital" "lon" "lat" "infector"
[13] "source" "age" "age_unit" "row_num"
[17] "wt_kg" "ht_cm" "ct_blood" "fever"
[21] "chills" "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header" "x28" 注釈:最終列の列名 “…28” が “x28” に変化したことに注目してください。
手動による列名のクリーニング
上で行った自動クリーニングによる列名の標準化の後に、列名を個別に、一つずつ変更することが必要な場合があります。 rename() は NEW = OLD というスタイルを採用しており、新しい列名を既存の列名の前に書く必要があります。
以下では、上で作成した自動クリーニングのパイプラインに名前を個別に変更するコマンドを追加しています。コードを読みやすくするため、戦略的にスペースを追加しています。
# パイプチェーン処理 (未加工データから始まり、クリーニングステップを経由してパイプで処理される)
##################################################################################
linelist <- linelist_raw %>%
# 列名の標準化
janitor::clean_names() %>%
# 列名を個別に変更
# 新しい列名 # 既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome)これで、列名が変更されていることがわかります。
[1] "case_id" "generation" "date_infection"
[4] "date_onset" "date_hospitalisation" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" 列の位置による名前の変更
列名ではなく、列の位置によって列の名前を変更することもできます。
rename(newNameForFirstColumn = 1,
newNameForSecondColumn = 2)
select() および summarise() による列名の変更
ショートカットとして、dplyr パッケージの select() や summarise() でも列の名前を変更することができます。select() は特定の列だけを残すために使用します(本章で後述します)。summarise() については、データのグループ化 の章や、記述統計表の作り方 の章で説明します。これらの関数も rename() と同様に、new_name = old_name というフォーマットを使用します。以下はその例です。
linelist_raw %>%
select(#新しい列名 # 既存の列名
date_infection = `infection date`,
date_hospitalisation = `hosp date`) #列名を変更し、変更した列だけを残すその他の課題
空白の Excel 列名
R は、列名(ヘッダー)のないデータセット列を処理することはできません。したがって、データはあるがヘッダーがない Excel データセットをインポートした場合、R は “…1” や “…2” といった名前でヘッダーを埋めます。数字は列番号を表します(例:データセットの 4 列目にヘッダーがない場合、R はその列を “…4” と命名します)。
R によって付与された名前は、ポジション番号(上記の例を参照)または割り当てられた名前(linelist_raw$...1)を参照することで、手動でクリーニングすることができます。
Excel の列名とセルの結合
Excel ファイル内のマージされたセルは、データを受け取る際によく問題になります。Excel・Stata・SASとの比較 の章で説明したように、マージされたセルは、人間がデータを読むときには便利ですが、 コンピュータにとっては「整頓されたデータ」ではないため、 データを読み込む際に多くの問題が発生します。R はマージされたセルに対応できないからです。
人間が読めるデータとコンピュータが読めるデータは別物であることを、データ入力をする担当者に伝えてください。また、整頓されたデータの原則 をユーザーに教えるよう努めてください。可能であれば、セルが結合されていない整頓されたフォーマットでデータが手元に届くように、データ入力や収集の手順を変更しましょう。
- 各変数はそれぞれ個別の列でなければなりません。
- 各観測値は、それぞれ個別の行である必要があります。
- それぞれの値は、それぞれ別のセルに入力されていなければなりません。
rio パッケージの import() を使用した場合、マージされたセルの値は最初のセルに割り当てられ、それ以降のセルは空になります。
結合されたセルを処理するための一つの解決策は、openxlsx パッケージの readWorkbook() を使用してデータをインポートすることです。引数 fillMergedCells = TRUE を設定すると、マージされたセルの値を、マージ範囲内のすべてのセルに入れることができます。
linelist_raw <- openxlsx::readWorkbook("linelist_raw.xlsx", fillMergedCells = TRUE)警告:readWorkbook() で列名をマージすると、列名が重複してしまうので、手動で修正する必要があります。R は列名が重複するとうまく動作しません!重複した列名がある場合は、手動による列名のクリーニングのセクションで説明したように、列の位置(例えば 5 列目)を参照し、列名を再設定することができます。
8.5 列の選択と並び替え
dplyr パッケージの select() を使って、残したい列を選択し、データフレーム内における列の順番を指定します。
注意:以下の例では、linelist データフレームを select() で変更して表示していますが、これは例としてデータを示すためであり、変更されたデータは保存はされていないことに注意してください。変更された列名は、データフレームを names() にパイプすることで表示されます。
まず、現時点での linelist データフレームのすべての列名を以下に表示します。
names(linelist) [1] "case_id" "generation" "date_infection"
[4] "date_onset" "date_hospitalisation" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" 列の保持
残しておきたい列のみを選択する
select() の中で、列の名前を引用符(” “)を使わずに指定します。指定された列は、指定された順序でデータフレームに表示されます。存在しない列を指定した場合、R がエラーを返すことに注意してください(このような場合にエラーを出さないようにするには、後述の any_of() の使い方を参照してください)。
# ラインリストのデータセットはselect()に渡され、name()で列名を表示する
linelist %>%
select(case_id, date_onset, date_hospitalisation, fever) %>%
names() # 列名を表示する[1] "case_id" "date_onset" "date_hospitalisation"
[4] "fever" “tidyselect” ヘルパー関数
tidyverse パッケージに含まれる tidyselect パッケージのヘルパー関数は、列の保持や除外、または変換する列を簡単に指定するための関数であり、dplyr 系関数で列を選択する方法の基礎となっています。
例えば、列の順番を変更したい場合、everything() は「記載されていない他のすべての列」を意味する便利な関数です。以下のコードは、date_onset と date_hospitalisation の列をデータセットの最初(左)に移動させますが、それ以外の列はすべてそのままにします。everything() の括弧内は空であることに注意してください。
# date_onset と date_hospitalisation を先頭に移動させる
linelist %>%
select(date_onset, date_hospitalisation, everything()) %>%
names() [1] "date_onset" "date_hospitalisation" "case_id"
[4] "generation" "date_infection" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" 以下に、select()、across()、summary() などの dplyr 系関数内で使える他の “tidyselect” ヘルパー関数を紹介します。
everything():指定されていない他のすべての列を指定します。last_col():最後の列を指定します。where():すべての列に関数を適用し、TRUEとなるものを選択します。-
contains():ある文字列を含む列を指定します。- 例)
select(contains("time"))
- 例)
-
starts_with():指定された接頭語を指定します。
- 例)
select(starts_with("date_"))
- 例)
-
ends_with():指定された接尾語を指定します。- 例)
select(ends_with("_post"))
- 例)
-
matches():正規表現(regex)を適用し、列を指定します。- 例)
select(matches("[pt]al"))
- 例)
num_range():x01、x02、x03 のような数値の範囲を指定します。-
any_of():関数内で使用した場合、指定する列が存在する場合は、その関数が適用され、存在しない場合は適用されず、エラーを表示しません。- 例)
select(any_of(date_onset, date_death, cardiac_arrest))
- 例)
さらに、複数の列を列挙する場合には c()、連続した列には :、反対の列には !、AND には &、OR には | などの通常の演算子が使用できます。
列の論理的な条件を指定したい場合は、where() を使用します。where() の中に関数を入れる場合は、関数の後に空の括弧をつけないでください。以下のコードは、 数字型データの列をすべて選択します。
# 数字型データの列を選択する
linelist %>%
select(where(is.numeric)) %>%
names()[1] "generation" "lon" "lat" "row_num" "wt_kg"
[6] "ht_cm" "ct_blood" "temp" ある特定の文字列が含まれている列のみを選択したい場合は、contains() を使用します。 また、ends_with() や starts_with() を使用すると、より詳細に列を指定することができます。
# 特定の文字を含む列を選択する
linelist %>%
select(contains("date")) %>%
names()[1] "date_infection" "date_onset" "date_hospitalisation"
[4] "date_outcome" さらに、contains() と同様の動作をする matches() は、 OR バー(|)で区切られた複数の文字列などの正規表現を括弧内で指定することができます(正規表現の詳細については、文字型・文字列型データ の章を参照ください)。
# 複数の文字列に当てはまる列を検索する
linelist %>%
select(matches("onset|hosp|fev")) %>% # "|" は OR を意味する
names()[1] "date_onset" "date_hospitalisation" "hospital"
[4] "fever" 注意:指定した列名がデータセットに存在しない場合、エラーが返されてコードの処理が停止することがあります。存在するかどうかわからない列を指定したい場合は、any_of() を使用することをおすすめします。any_of() は、列の除外など、否定的な列の選択をする場合においてと特に有用です。
以下のコードでは、指定されている列のうち 1 つだけがデータセットに存在しますが、エラーは発生せず、コードは処理を停止することなく実行されます。
linelist %>%
select(any_of(c("date_onset", "village_origin", "village_detection", "village_residence", "village_travel"))) %>%
names()[1] "date_onset"列の除外
データセットから除外したい列がある場合は、列名の前にマイナス記号(-)を付ける(例:select(-outcome))、または以下のように列名のベクトルの前にマイナス記号(-)を付けて指定すると、除外できます。指定された以外の列はすべて残ります。
linelist %>%
select(-c(date_onset, fever:vomit)) %>%
# date_onsetと、feverからvomitまでのすべての列を削除する
names() [1] "case_id" "generation" "date_infection"
[4] "date_hospitalisation" "date_outcome" "outcome"
[7] "gender" "hospital" "lon"
[10] "lat" "infector" "source"
[13] "age" "age_unit" "row_num"
[16] "wt_kg" "ht_cm" "ct_blood"
[19] "temp" "time_admission" "merged_header"
[22] "x28" R の基本的な構文を使用し、除外したい列を NULL と定義して削除することもできます。例えば、以下のようになります。
linelist$date_onset <- NULL # Rの基本的な構文で列を削除する独立型
select() は、パイプ内で使用するだけでなく、独立したコマンドとして使用することもできます。この場合、最初の引数には前処理したい元のデータフレームを指定します。
# IDと年齢に関連した列を持つ新しいラインリストを作成する
linelist_age <- select(linelist, case_id, contains("age"))
# 列名を表示する
names(linelist_age)[1] "case_id" "age" "age_unit"パイプラインに追加
linelist_raw データセットには、row_num、merged_header、x28 という必要のない列があります。以下に、前処理パイプラインの select() コマンドで削除する例を示します。
# パイプライン処理 (未加工データから始まり、クリーニングステップを経由してパイプで処理される)
##################################################################################
# パイプラインを開始する
###########################
linelist <- linelist_raw %>%
# 列名の標準化
janitor::clean_names() %>%
# 列名を個別に変更
#新しい列名 #既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# ここまでは、すでに説明したクリーニング手順
#####################################################
# 列の除外
select(-c(row_num, merged_header, x28))8.6 重複行の削除
重複したデータを削除する方法の詳細については、このハンドブックの 重複データの排除 の章を参照してください。本章では、重複した行の削除について、非常に簡単な例のみを紹介します。
dplyr パッケージの distinct() は、データフレーム内のすべての行をチェックし、重複していない行のみを残します。つまり、完全に重複している行をデータフレームから削除します。
重複する行をチェックする際には、列の範囲を指定できますが、デフォルトではデータフレーム内のすべての列が対象となります。重複データの排除 の章にあるように、対象となる列の範囲を調整して特定の列に関してのみ、重複した行があるかをチェックすることもできます。
以下は、空のコマンド distinct() をパイプチェーンに追加するだけのシンプルな例です。これにより、他の行と完全に同一である行がない(重複している行がない)ことが保証されます(データフレーム内のすべての列がチェックされます)。
現時点の linelist データフレームには、全部で nrow(linelist) 行が含まれています。
linelist <- linelist %>%
distinct()上のコードを実行して重複削除すると、データフレームの行数は nrow(linelist) になります。他の行と完全に同一である行が削除されました。
以下では、パイプラインに distinct() コマンドを追加しています。
# パイプチェーン処理 (未加工データから始まり、クリーニングステップを経由してパイプで処理される)
##################################################################################
# パイプラインを開始する
###########################
linelist <- linelist_raw %>%
# 列名の標準化
janitor::clean_names() %>%
# 列名を個別に変更
# 新しい列名 # 既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# 列の除去
select(-c(row_num, merged_header, x28)) %>%
# ここまでは、すでに説明したクリーニング手順
#####################################################
# 重複の除去
distinct()8.7 列の作成と変換
新しい列の追加や、既存の列の修正が必要な場合には、dplyr 系関数の mutate() を使用することをおすすめします。
以下に、mutate() を使用して新しい列を作成する例を紹介します。次のような構文を使用します:mutate(new_column_name = value or transformation)
Stata の generate コマンドに似ていますが、R の mutate() は既存の列を修正する場合にも使用できます。
新しい列の作成
以下は、mutate() を使用して新しい列を作成する最も基本的なコマンドです。ここでは、すべての行の値が 10 である新しい列 new_col を作成します。
linelist <- linelist %>%
mutate(new_col = 10)また、他の列の値を参照して、計算を行うこともできます。以下では、bmi という新しい列が作成され、各症例(各行)の Body Mass Index(BMI)を格納しています。これは、既存の列である ht_cm と wt_kg を使用して、BMI = kg/m^2 の式で計算されたものです。
linelist <- linelist %>%
mutate(bmi = wt_kg / (ht_cm/100)^2)新しい列を複数作成する場合は、それぞれをコンマで区切って改行します。以下は、stringr パッケージの str_glue() を使って他列の値を組み合わせた列を作成するなど、複数の新しい列を作成する場合の例です(str_glue() についての詳細は、文字型・文字列型データ の章を参照ください)。
new_col_demo <- linelist %>%
mutate(
new_var_dup = case_id, # 新しい列=既存の列のコピーを作成する
new_var_static = 7, # 新しい列=全ての値が同じ
new_var_static = new_var_static + 5, # 列を上書きしたり、他の変数を使って計算したりすることができる
new_var_paste = stringr::str_glue("{hospital} on ({date_hospitalisation})")
# 新しい列 = 他の列の値を貼り付ける
) %>%
select(case_id, hospital, date_hospitalisation, contains("new"))
# デモ用に新しい列のみを表示以下では、新しい列を確認するために、新しい列とそれを作成するために使用された列のみが表示されています。
ヒント:mutate() の変化形として、transmute() という関数があります。この関数は、mutate() と同じように新しい列を追加しますが、括弧の中に書かれていないすべての列を削除または除外します。
# HIDDEN FROM READER(ウェブ版には表示されない)
# 上で新しく作成されたデモ用の列を除外する
# linelist <- linelist %>%
# select(-contains("new_var"))列のデータ型の変換
日付、数字、理論値(TRUE/FALSE)を含む列は、正しく分類された場合のみ期待通りの動きをします。例えば、文字型の “2” と数字型の 2 は異なることに注意してください。
データをインポートするコードの中に列のデータ型を指定する方法もありますが、扱いにくい場合が多いです。オブジェクトや列のデータ型を変換する方法については、R の基礎 の章の 3.10 オブジェクトのデータ型のセクションを参照してください。
本章では、まず、重要な列について、正しいデータ型であるかどうかのチェックを行ってみましょう。これは本章の最初に skim() を実行したときに確認しました。
では、具体的に見ていきましょう。現在、age 列のデータ型は 文字列型(character)ですが、定量的な分析を行うためには、age 列の数字が数値として認識される必要があります。
class(linelist$age)[1] "character"以下で、date_onset 列のデータ型も確認してみると、文字列型であることがわかります。分析を行うためは、date_onset 列の日付が日付として認識される必要があります。
class(linelist$date_onset)[1] "character"このようなデータ型の相違を解決するために、mutate() の機能を使用し、変換で列を再定義していきます。列の定義は変更しませんが、データ型を変更します。以下では、基本的な例として、age 列を数字型(numeric)に変換します。
linelist <- linelist %>%
mutate(age = as.numeric(age))同様に、as.character() や as.logical() を使用することもできます。因子型(factor)に変換したい場合は、base R の factor() や forcats パッケージの as_factor() を使いましょう。詳しくは、因子(ファクタ)型データ の章をご覧ください。
日付型(date)への変換には注意が必要です。日付型データ の章でいくつか方法を紹介しますが、通常、日付型への変換を正しく行うためには、日付データがすべて同じ形式である必要があります(例:“MM/DD/YYYY”、または “DD MM YYYY”)。データを日付型に変換した後にチェックして、各値が正しく変換されたことを確認してください。
グループ化されたデータ
データフレームがすでにグループ化されている場合(グループ化の詳細は、データのグループ化 の章を参照ください)は、mutate() の動作がグループ化されていない場合と異なるかもしれません。データフレームがすでにグループ化されている場合、mean()、median()、max() などの要約関数は、すべての行ではなくグループごとに計算されます。
# 全行の平均値で正規化された年齢
linelist %>%
mutate(age_norm = age / mean(age, na.rm=T))
# 年齢を病院グループの平均値で正規化
linelist %>%
group_by(hospital) %>%
mutate(age_norm = age / mean(age, na.rm=T))グループ化されたデータフレームでの mutate () の使用については、こちらの tidyverse mutate に関するドキュメント で詳しく説明されています。必要な方はご覧ください。
複数列の一斉変換
コードを簡潔にするために、同じ変換を行うコマンドを複数の列に適用したい場合があります。dplyr パッケージ( tidyverse パッケージにも含まれています)の across() を使用すると、データの変換を行うコマンドを一度に複数の列に適用することができます。 across() はすべての dplyr 系関数で使用することができますが、一般的には select()、mutate()、filter()、summarise() 内でよく使用されます。across() を summarise() 内でどのように使用するかについて知りたい方は、記述統計表の作り方 の章をご覧ください。
across() では、.cols = 引数には列を指定し、.fns = 引数には適用する関数を指定します。.fns に与える追加の引数は、コンマの後に含めることができます。
across() を使用して列を選択する
.cols = 引数に列を指定します。 個別に列名を指定することもできますし、“tidyselect” ヘルパー関数を使用することもできます。関数を .fns = 引数に指定します。以下に示すように、関数機能を使用すると、関数はその括弧( )なしで記述することに注意してください。
以下の例では、as.character() を使用した文字型への変換が、 across() で指定された特定の列に適用されます。
linelist <- linelist %>%
mutate(across(.cols = c(temp, ht_cm, wt_kg), .fns = as.character))列を指定する際には、“tidyselect” ヘルパー関数を使用すると便利です。“tidyselect” ヘルパー関数には、everything()、last_col()、where()、starts_with()、ends_with()、contains()、matches()、num_range()、any_of() などがあり、本章上部の列の選択と並び替えのセクションで詳述されています。
まず、データフレーム内のすべての列を文字列型(character)に変換する例を以下に示します。
# すべての列を文字列型に変換する
linelist <- linelist %>%
mutate(across(.cols = everything(), .fns = as.character))他の例として、例えば列名に “date”という文字列が含まれている列を文字列型に変換したい場合のコマンドを以下に紹介します(コンマと括弧の配置に注意してください)。
# date という文字が列名に含まれている列を文字列型に変換する
linelist <- linelist %>%
mutate(across(.cols = contains("date"), .fns = as.character))また、別の例として、現在 POSIXct 型(未加工の日付時分秒を示すデータ型)の列を日付型(date)に変換する例を示します。まず、is.POSIXct() の評価値が TRUE である列を where() で検索し、次に、これらの列に as.Date() を適用して、通常の 日付型(date)に変換します。
linelist <- linelist %>%
mutate(across(.cols = where(is.POSIXct), .fns = as.Date))across()内でwhere()を使用し、is.POSIXct()で各列が TRUE または FALSE か評価できるようにしていることに注意してください。is.POSIXct()は lubridate パッケージの関数であることに注意してください。is.character()、is.numeric()、is.logical()などの他の類似した “is” 系関数は base R の関数です。
across()
across() に関数を与える方法の詳細については、?across をコンソールで実行して表示されるドキュメントをお読みください。要約すると、対象の列に対して実行する関数を指定する方法はいくつかあり、独自の関数を指定することもできます。
関数名のみを指定することができます(例:
meanまたはas.character)。関数を purrr スタイルで使用することができます(例:
~ mean(.x, na.rm = TRUE))(詳細は、ループと反復処理・リストの操作 の章を参照してください)。-
リストを使用することで、複数の関数を指定することができます(例:
list(mean = mean, n_miss = ~ sum(is.na(.x))))。- 複数の関数を指定した場合は、入力された列ごとに複数の変換後の列が返され、
col_fnという形式で一意の名前が付けられます。返された列の名前を調整したい場合は、.names =引数で glue パッケージの構文を使用してください(glue パッケージに関する詳細は、文字型・文字列型データ の章を参照ください)。{.col}と{.fn}は入力された列と関数の省略形です。
- 複数の関数を指定した場合は、入力された列ごとに複数の変換後の列が返され、
coalesce()
coalesce() は、dplyr パッケージに含まれており、指定された値のうち、欠損値ではない最初の値を見つける関数です。この関数は、指定された順序で、取得された欠損値ではない最初の値で欠損値を埋めます。
ここでは、データフレーム以外の例を示します。2 つのベクトルがあるとします。1 つは患者の発見された村、もう 1 つは患者の居住地の村です。coalesce() を使用し、各ベクトルの欠損値ではない最初の値を見つけることができます。
village_detection <- c("a", "b", NA, NA)
village_residence <- c("a", "c", "a", "d")
village <- coalesce(village_detection, village_residence)
village # 結果を表示する[1] "a" "b" "a" "d"coalesce() は、データフレームの列を対象とした場合にも同じように動作します。この関数は、指定した列の中で欠損地ではない最初の値を、新しい列の値として各行ごとに割り当てます。
linelist <- linelist %>%
mutate(village = coalesce(village_detection, village_residence))これは、データフレームの行ごとに処理をしていく「行処理(row-wise 演算)」の一例です。より複雑な行処理については、本章下部の 8.12 行単位の計算 のセクションをご覧ください。
累積計算
データフレームの行を計算し、その時点までの累積和、平均、最小、最大などを列に反映させたい場合は、以下の関数を使用します。
cumsum() は、以下のように、累積和を返します。
sum(c(2,4,15,10)) # 1つの数字のみを返す[1] 31cumsum(c(2,4,15,10)) # 各ステップでの累積和を返す[1] 2 6 21 31これは、データフレームで新しい列を作るときに使用できます。例えば、アウトブレイクにおける一日あたりの累積症例数を知りたい場合は、次のようなコードを使って計算できます。
cumulative_case_counts <- linelist %>% # 症例ラインリスト
count(date_onset) %>% # 一日当たりの行数をカウント
mutate(cumulative_cases = cumsum(n)) # 累積和の新しい列以下に、最初の 10 行を表示します。
head(cumulative_case_counts, 10) date_onset n cumulative_cases
1 2012-04-15 1 1
2 2012-05-05 1 2
3 2012-05-08 1 3
4 2012-05-31 1 4
5 2012-06-02 1 5
6 2012-06-07 1 6
7 2012-06-14 1 7
8 2012-06-21 1 8
9 2012-06-24 1 9
10 2012-06-25 1 10流行曲線(エピカーブ)で累積罹患率をプロットする方法については、流行曲線(エピカーブ) の章をご覧ください。
R の基本パッケージ base を使用する
base R を使って新しい列を定義する(または列を再定義する)こともできます。その場合は、新しい列(または修正する列)にデータフレームの名前を $ でつないで書き、代入演算子 <- を使って新しい値を定義します。base R を使用する際には、毎回、列名の前にデータフレーム名を指定しなければならないことを覚えておいてください(例:dataframe$column)。ここでは、base R を使用して 新しく bmi という列を作成する例を示します。
linelist$bmi = linelist$wt_kg / (linelist$ht_cm / 100) ^ 2)パイプラインへの追加
以下では、パイプラインに新しい列を追加し、いくつかの列のデータ型を変換しています。
# パイプライン処理 (未加工データから始まり、クリーニングステップを経由してパイプで処理される)
##################################################################################
# パイプラインの開始
###########################
linelist <- linelist_raw %>%
# 列名の標準化
janitor::clean_names() %>%
# 列名を個別に変更
# 新しい列名 #既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# 列の除去
select(-c(row_num, merged_header, x28)) %>%
# 重複行の削除
distinct() %>%
# ここまでは、すでに説明したクリーニング手順
###################################################
# 新しい列の追加
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# 列のデータ型を変換
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) 8.8 値の再定義
以下のような場合には、値を再定義(変更)する必要があります。
特定の値を編集する場合(例:日付の年号や書式が間違っている場合など)。
同じ綴りでない値を調整する場合。
カテゴリ値の新しい列を作成する場合。
数値カテゴリの新しい列を作成する(例:年齢カテゴリ)。
特定の値
値を手動で変更するためには、mutate() 内で recode() を使用します。
例えば、データの中に意味のない日付(例:“2014-14-15”)があるとします。未加工の元データ上でマニュアルで日付を修正することもできますし、mutate() と recode() を使用して前処理パイプラインに日付を修正するコマンドを書き込むこともできます。後者の方がより透明性と再現性が高く、行われた分析を理解しようとする第三者や同じように分析しようとする人にとって、分析を再現しやすくなります。
# 間違っている値の修正 # 既存の値 # 新しい値
linelist <- linelist %>%
mutate(date_onset = recode(date_onset, "2014-14-15" = "2014-04-15"))上の例の mutate() の行は、「date_onset 列内の間違っている値を新しい値に変換し、date_onset 列に再定義する」と読むことができます。recode() のパターン(既存の値 = 新しい値)は、ほとんどの R の関数のパターン(新しい値 = 既存の値)とは逆であることに注意してください。R の開発コミュニティでは、この相違の統一化に取り組んでいます。
以下に、別の例として、1 つの列において複数の値を再定義する例を紹介します。
linelist の hospital 列には、いくつか綴り(つづり)が間違った値があることに加え、多くの欠損値があるため、hospital 列の値をクリーニングする必要があります。
table(linelist$hospital, useNA = "always")
Central Hopital Central Hospital
11 457
Hospital A Hospital B
290 289
Military Hopital Military Hospital
32 798
Mitylira Hopital Mitylira Hospital
1 79
Other Port Hopital
907 48
Port Hospital St. Mark's Maternity Hospital (SMMH)
1756 417
St. Marks Maternity Hopital (SMMH) <NA>
11 1512 # 欠損値を含むすべての一意の値のテーブルを表示する以下の recode() コマンドでは、指定した複数の値を書き換え、hospital 列を再定義しています。既存の値から新しい値に書き換えるそれぞれのコードの後にコンマを忘れないようにしてください。
linelist <- linelist %>%
mutate(hospital = recode(hospital,
# 参照: 既存の値 = 新しい値
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
))上のコードを実行すると、 hospital 列内で間違った綴りの値が修正され、正しい綴りの値のグループに統合されていることがわかります。
table(linelist$hospital, useNA = "always")
Central Hospital Hospital A
468 290
Hospital B Military Hospital
289 910
Other Port Hospital
907 1804
St. Mark's Maternity Hospital (SMMH) <NA>
428 1512 ヒント:等号(=)の前後には、スペースをいくつか入れても問題ありません。コードを読みやすくするために、すべての行またはほとんどの行で等号(=)の場所を揃えることをおすすめします。また、ハッシュタグでコメント行を追加し、どちらが古い値でどちらが新しい値なのかを、第三者に明確にわかるようにすることも大事なポイントです。
ヒント:データセットの中には、空白文字の値が存在することがあります(これは、R の欠損値である NA として認識されません)。このような値を参照するためには、2 つの引用符をスペースを空けずに(““)使用します。
論理条件
以下では、論理条件を使用して列の値を再定義する方法を説明します。
単純な論理条件:
replace()、ifelse()、if_else()を使う複雑な論理条件:
case_when()を使う
単純な論理条件
replace()
replace() は base R の関数で、論理条件を使って変更する行を指定します。一般的な構文は以下の通りです。
mutate(col_to_change = replace(col_to_change, criteria for rows, new value))
replace() を使用する一般的な状況としては、一意の行識別子(症例ごとの ID 番号など)を使用して、1 つの行の 1 つの値だけを変更する場合です。下の例では、case_id が「2195」である行の性別を “Female”に変更しています。
# 例:ある特定の観測データの性別を "Female" に変更する
linelist <- linelist %>%
mutate(gender = replace(gender, case_id == "2195", "Female"))R の基本的な構文と角括弧 [ ] を使った同等のコマンドは、以下の通りです。linelist データフレームの gender 列の値を(linelist の case_id 列の値が「2195」である行について)“Female” に変更する、と読めます。
linelist$gender[linelist$case_id == "2195"] <- "Female"
ifelse() と if_else()
簡潔な論理条件のためのツールとして、ifelse() とそのパートナーである if_else() があります。しかし、ほとんどの場合、再定義のためにはcase_when()(以下に詳述)を使ったほうがわかりやすいでしょう。これらの “if else” コマンドは、if とelse のプログラミング文を簡略化したものです。一般的な構文は以下の通りです。
ifelse(condition, value to return if condition evaluates to TRUE, value to return if condition evaluates to FALSE)
下の例では、source_known という列が新しく定義されています。既存の列 source の値が欠損していなければ、source_known 列の値は “known” に設定され、source 列の値が欠落している場合は、 “unknown” に設定されます。
linelist <- linelist %>%
mutate(source_known = ifelse(!is.na(source), "known", "unknown"))if_else() は、日付を扱う dplyr の特別バージョンです。真(TRUE)の値が日付の場合、「偽(FALSE)」の値も日付でなければならないことに注意してください。そのため、単に NA ではなく NA_real_ という特別な値を使用しています。
# 患者が死亡していない場合はNAとなる死亡日の列を作成する
linelist <- linelist %>%
mutate(date_death = if_else(outcome == "Death", date_outcome, NA_real_))ifelseコマンドをたくさん並べるのは避けましょう…代わりにcase_when()を使ってください!case_when() の方がずっと読みやすく、エラーも少なくなります。
データフレーム以外で、コード内で使用するオブジェクトの値を切り替えたい場合は、base R のswitch() の使用を検討してください。
複雑な論理条件
ある変数を複数のグループに分ける場合や、複雑な論理条件文を使って値を再定義する必要がある場合は、dplyr の case_when() を使用します。この関数は、データフレーム内のすべての行を計算し、その行が指定された基準を満たしているかどうかを評価して、正しい値を新しく割り当てます。
case_when() コマンドは、右側(Right-Hand Side: RHS)左側(Left-Hand Side: LHS)をチルダ ~ で区切ったコードで構成されています。各コードの左側には論理条件が、右側には準拠値が記述されて、コンマで区切られています。
例えば、ここでは age と age_unit の列を利用して age_years の列を作成しています。
linelist <- linelist %>%
mutate(age_years = case_when(
age_unit == "years" ~ age, # 年齢が年単位の場合
age_unit == "months" ~ age/12, # 年齢が月単位の場合、値を 12 で割る
is.na(age_unit) ~ age)) # 年齢の単位が見つからない場合は、年を仮定
# その他の状況では、欠損値 NA とするデータの各行が評価されるとき、case_when() 内に書かれたコードの順に、上から下へと適用・評価されます。ある行で論理式が TRUE と評価された場合、その行の評価式の右側(RHS)の値がオブジェクトに割り当てられ、残りの論理式は同じ行では評価されません。したがって、もっとも明確な範囲の論理式(狭い範囲の論理式)を最初に記述して、もっとも一般的な範囲の論理式(広い範囲の論理式)を最後に書くのがよいでしょう。いずれの論理式の右側 (RHS) にも当てはまらなかった場合は、オブジェクトに NA が割り当てられます。
ときには、最後の行にそれ以前の行で記述しなかったすべての場合に値を割り当てる論理式を記述する必要があるかもしれません。この場合の論理式は、左辺 (LHS) に TRUE と記述すると実現できます。この記述方法は、この行より前の行の論理式のいずれも当てはまらない場合に対応します。左辺に TRUE と書いた論理式の右辺 (RHS) には、“check me!” や “missing” のような文字列型を指定できます。
以下は、case_when() の別例です。確定症例と疑義症例を定義する論理式に従い、症例各行の分類を示す新しい列を作成する例です。
linelist <- linelist %>%
mutate(case_status = case_when(
# 患者が血液検査を受けて、結果が陽性だった場合、
# 該当行の case_status 列に "Confirmed"(確定症例)を代入する
ct_blood < 20 ~ "Confirmed",
# 患者の血液検査が陽性ではなく、かつ
# "source"(疫学的関係あり)であり、かつ、発熱している (fever) 場合
# 該当行の case_status 列に "Suspect"(疑義症例)を代入する
!is.na(source) & fever == "yes" ~ "Suspect",
# 上記の論理式に該当しない患者の場合
# "To investigate"(要調査)を代入する
TRUE ~ "To investigate"))警告:右側の値はすべて同じデータ型でなければなりません。欠損値(NA)を割り当てるには、NA_character_、NA_real_(数値や POSIX の場合)、as.Date(NA)など、データ型によって異なる NA を使用する必要があるかもしれません。詳しくは、日付型データの章の日付の操作 のセクションをご覧ください。
欠損値
以下は、データクリーニングの観点から欠損値を処理するための特別な関数です。
論理的に欠損値をテストする is.na() などの関数について知りたい場合など、欠損値の識別と処理に関して詳しく学びたい方は、欠損データの処理 の章を参照してください。
replace_na()
欠損値(NA)を “Missing” などの特定の値に変更する場合は、dplyr パッケージに含まれる replace_na() をmutate() 内で使用します。これは、上記の recode と同じように使用されることに注意してください 。対象となる変数の名前は、replace_na() 内で再度明記される必要があります。
linelist <- linelist %>%
mutate(hospital = replace_na(hospital, "Missing"))fct_explicit_na()
fct_explicit_na() は、forcats パッケージに含まれている関数です。forcats パッケージは、因子(ファクタ)型の列を扱います。因子型は、c("First", "Second", "Third") のような順序付きの値を扱うための R の方法であり、表やプロットに表示される値(病院など)の順序を設定します。詳細は、因子(ファクタ)型データ の章を参照してください。
データが因子型で、replace_na() を使って NA を “Missing” に変換しようとすると、以下のようなエラーが表示されます。
invalid factor level, NA generated.
“Missing” を因子型がとれるレベルとして定義せずに追加しようとしたため、エラーとなります。
この問題を解決する最も簡単な方法は、forcats パッケージの fct_explicit_na() を使用することです。この関数は、列を因子型に変換し、NA 値を文字型の “(Missing)” に変換します。
linelist %>%
mutate(hospital = fct_explicit_na(hospital))もう一つ、さらに手間がかかる方法ですが、fct_expand() を用いて因子型を追加し、その後、欠損値を変換しても同じ結果を得ることができます。
na_if()
特定の値を NA に変換するには、dplyr の na_if() を使います。以下のコマンドは replace_na() とは逆の操作を行います。以下の例では、列 hospital の “Missing” の値はすべて NA に変換されます。
linelist <- linelist %>%
mutate(hospital = na_if(hospital, "Missing"))注意: na_if() は論理的条件(例:“all values > 99”)には使用できません。このような論理的条件には、replace() または case_when() を使用してください。
# 40度以上の温度をNAに変換する
linelist <- linelist %>%
mutate(temp = replace(temp, temp > 40, NA))
# 2000年1月1日以前の発症日を欠損に変換する
linelist <- linelist %>%
mutate(date_onset = replace(date_onset, date_onset > as.Date("2000-01-01"), NA))クリーニングディクショナリ(Cleaning dictionary)
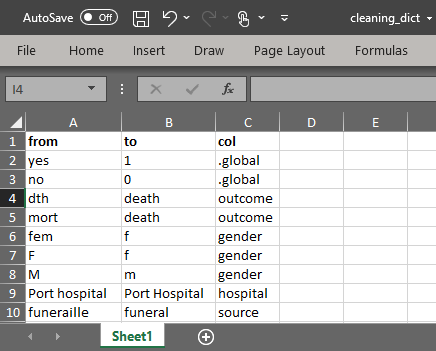
R の matchemaker パッケージに含まれる関数 match_df() を使用し、データフレームをクリーニングディクショナリを用いて前処理します。
- まず、クリーニングディクショナリを作成します。クリーニングディクショナリは、以下の 3 つの列で構成される必要があります。
from 列(不正な値)
to 列(正しい値)
変更を適用する列を指定する列(すべての列に変更を適用する場合は、“.global” と記入する)
注意:.globalのディクショナリ項目は、列固有のディクショナリ項目によって上書きされます。
- 本章では、ハンドブックとデータのダウンロード の章にあるクリーニングディクショナリを例として使用しますので、上のリンクからダウンロードし、インポートしてください。
cleaning_dict <- import("cleaning_dict.csv")-
未加工のラインリストを
match_df()に渡し、distionary =引数にインポートしたクリーニングディクショナリデータフレームを指定します。引数from =には「置き換え前」の値を含むディクショナリの列名を指定します。引数by =には対応する「置き換え後」の値を含む列名を指定し、データフレームの 3 列目には置き換えする列名を列挙します。すべての列に対して置き換えを実行する場合は、by =列に.globalを指定します。4 列目orderは、変更後の値の並び順を指定します。
詳しくは ?match_df を実行すると表示されるパッケージドキュメントを参照してください。この関数は、サイズの大きなデータセットでは、実行時間が長いことに注意してください。
linelist <- linelist %>% # match_df() にデータセットを渡す
matchmaker::match_df(
dictionary = cleaning_dict, # ディクショナリデータフレームのオブジェクト名を指定
from = "from", # 置き換える値を格納した列を指定(デフォルトでは、1 列目)
to = "to", # 置き換え後の値を格納した列を指定(デフォルトでは 2 列目)
by = "col" # 置き換え対象の列名を列挙した列を指定(デフォルトでは 3 列目)
)以下の表を右側にスクロールすると、上のコマンドを実行後に値がどのように変化したかがわかります。特に、性別の変化(小文字から大文字に変換された)や、すべての症状の列が yes/no の 2 択から 1/0 の 2 択に変化したことに注目してください。
クリーニングディクショナリの col 列に記入される列名は、クリーニングする時点での列名と同一でなければならないことに注意してください。詳細については、こちら の linelist パッケージのドキュメントを参照してください。
パイプチェーンへの追加
以下では、いくつかの新しい列と列変換がパイプチェーンに追加されています。
#クリーニングパイプチェーン (未加工データから始まり、クリーニングステップ経由してパイプで処理される)
##################################################################################
# パイプチェーンの処理開始
###########################
linelist <- linelist_raw %>%
# 列名の構文の標準化
janitor::clean_names() %>%
# 手動で列名を変更
# 新しい列名 # 既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# 列の除去
select(-c(row_num, merged_header, x28)) %>%
# 重複除去
distinct() %>%
# 列を追加
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# 列のデータ型を変換
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# 列の追加: 入院の遅れ
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# ここまでは、すでに説明したクリーニング手順
###################################################
# 病院の列の値をクリーニングする
mutate(hospital = recode(hospital,
# OLD = NEW
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# age_years 列を新しく作成する (age 列と age_unit 列を使用する)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age,
TRUE ~ NA_real_))8.9 数値カテゴリ
ここでは、数値データをカテゴリ化する方法を説明します。一般的な例としては、年齢のカテゴリー化や、検査結果のグループ化などがあります。ここでは、以下について説明します。
age_categories()( epikit パッケージより)cut()(base Rより)case_when()quantile()とntile()を使った分位数による分割
分布を確認する
例として、age_years 列を使用して新たに age_cat 列を作成していきます。
# linelistのage列のデータ型をチェックする
class(linelist$age_years)[1] "numeric"まずデータの分布を確認し、適切な分割点(カットポイント)を作ります。詳しくは ggplot の基礎 の章をご覧ください。
summary(linelist$age_years, na.rm=T) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.04 23.00 84.00 107 注意:数字型のデータが文字列型としてインポートされることがあります。これは、値の一部に数字以外の文字が含まれている場合に発生します。例えば、年齢を「2ヶ月」と入力した場合や、(R のロケール設定にもよりますが)小数点の代わりにコンマが使用されている場合などです(例:“4,5” が 4 年半の意味で使われている場合)。
age_categories()
epikit パッケージの age_categories() を使用すると、数値列の分類やラベル付けを簡単に行うことができます(注:この関数は、年齢以外の数値変数にも適用できます)。結果として出力される列は、自動的に順序付けられたカテゴリーとなります。
入力する必要がある項目は、以下の通りです。
数値のベクトル(列)
breakers =引数:新しいグループの分割点の数値ベクトルを指定する。
まず、簡単な例を示します。
# 簡単な例
################
pacman::p_load(epikit) #パッケージの読み込み
linelist <- linelist %>%
mutate(
age_cat = age_categories( # 新しい列の作成
age_years, # 分割点によってグループ化する数値列
breakers = c(0, 5, 10, 15, 20, # 分割点
30, 40, 50, 60, 70)))
# テーブルを表示
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-39 40-49 50-59 60-69 70+ <NA>
1227 1223 1048 827 1216 597 251 78 27 7 107 指定した分割点によるグループ分けは、デフォルトでは下限の値から始まります。つまり、下限の値は「上位」のグループに含まれ、グループが下または左に「開いている」状態になります。下図のように、各分割点に1を加えることで、上または右で開いたグループにすることができます。
# 同じカテゴリーの上端を含める
############################################
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
breakers = c(0, 6, 11, 16, 21, 31, 41, 51, 61, 71)))
# テーブルを表示
table(linelist$age_cat, useNA = "always")
0-5 6-10 11-15 16-20 21-30 31-40 41-50 51-60 61-70 71+ <NA>
1469 1195 1040 770 1149 547 231 70 24 6 107 separator = を使用すると、ラベルの表示方法を調整できます。 デフォルトは “-” です。
また、ceiling = を使用し、分割点として指定された数値のうち最大の値の扱い方を調整できます。ceiling = TRUE と設定すると、指定された分割点の最大値が「上限(ceiling)」となり、最大値を超えるカテゴリ “XX+” は作成されません。分割点の最大値(または upper = 引数が使用されている場合は、その値)を超える値は、NA に分類されます。以下は、ceiling = TRUE の例で、XX+ のカテゴリは作成されず、70(分割点の最大値)を超える値は NA として割り当てられます。
# 上限をTRUEに設定した場合
##########################
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
breakers = c(0, 5, 10, 15, 20, 30, 40, 50, 60, 70),
ceiling = TRUE)) # 70 が上限となり、70 以上の値は NA となる
# テーブルを表示
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-39 40-49 50-59 60-70 <NA>
1227 1223 1048 827 1216 597 251 78 28 113 また、breakers = の代わりに、lower=、upper=、by= のすべてを指定することもできます。
lower =下限となる値(デフォルトは 0)upper =上限となる値by =グループ内の年の数
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
lower = 0,
upper = 100,
by = 10))
# テーブルの表示
table(linelist$age_cat, useNA = "always")
0-9 10-19 20-29 30-39 40-49 50-59 60-69 70-79 80-89 90-99 100+ <NA>
2450 1875 1216 597 251 78 27 6 1 0 0 107 詳細は、age_categories() のヘルプページをご覧ください(R コンソールで ?age_categories と入力してください)。
cut()
cut() は age_categories() に代わる base R の関数ですが、age_categories() と比較すると複雑です。以下の説明を読むと、この処理を簡単にするために age_categories() が開発されたことがわかると思います。age_categories() との注目すべき違いは、以下のとおりです。
別のパッケージをインストール・ロードする必要がない。
グループが右か左に開いているか閉じているかを指定できる。
正確なラベルを自分で用意する必要がある。
一番下のグループに “0” を入れたい場合は、その旨を明記する必要がある。
cut() の基本的な構文は、まず分割したい数値列(age_years)を指定し、次に breaks 引数に分割点の数値ベクトル c() を指定します。cut() を使用すると、結果として出力される列は順序付けられたカテゴリとなります。
デフォルトでは、右または上側が「オープン」で包括的(左または下側が「クローズ」で排他的)になるように分類されます。これは、age_categories() 関数とは逆の動作です。デフォルトのラベルでは “(A, B]” という表記が使われており、これは「Aは含まれないがBは含まれる」という意味です。right = TRUE に設定することで、この動作を逆にすることができます。
デフォルトでは、“0” の値は一番下のグループから除外され、NA に分類されることに注意してください。乳幼児が 0 歳としてコードされているデータもあるので、注意が必要です。これを変更するには、引数 include.latest = TRUE を追加して、“0” の値が一番下のグループに含まれるようにします。この場合、自動的に作成される一番下のカテゴリのラベルは “[A],B]” となります。なお、include.latest = TRUE を指定し、かつ right = TRUE を指定した場合、含まれるのは最小値ではなく最大値の分割点とカテゴリーに適用されることに注意してください。
labels = 引数を使って、カスタマイズされたラベルのベクトルを指定することができます。手作業で指定する必要があるため、間違えのないように十分注意してください。後述するように、クロス集計を用いて作業を確認してください。
age_years に cut() を使用して、新しい変数 age_cat を作成した例を以下に示します。
# 年齢変数を分割してカテゴリ化した変数を新しく作る
# 下限の分割点は除外されるが、上限の分割点は各カテゴリに含まれる
linelist <- linelist %>%
mutate(
age_cat = cut(
age_years,
breaks = c(0, 5, 10, 15, 20,
30, 50, 70, 100),
include.lowest = TRUE
)) # 一番下のグループに0を入れる
# グループごとの観測値の数を集計する
table(linelist$age_cat, useNA = "always")
[0,5] (5,10] (10,15] (15,20] (20,30] (30,50] (50,70] (70,100]
1469 1195 1040 770 1149 778 94 6
<NA>
107 分割した結果を確認してください!!!数値列とカテゴリ列をクロス集計して、各年齢値が正しいカテゴリに割り当てられていることを検証します。特に、境界値の割り当てを確認してください(例:隣接するカテゴリが 10-15 と 16-20 の場合、15 の値がどこに割り当てられているか)。
# 数値列とカテゴリ列のクロス集計
table("Numeric Values" = linelist$age_years, # 分かりやすくするために、表の中に名前を入れる
"Categories" = linelist$age_cat,
useNA = "always") # NA値の検証を忘れずに Categories
Numeric Values [0,5] (5,10] (10,15] (15,20] (20,30] (30,50] (50,70]
0 136 0 0 0 0 0 0
0.0833333333333333 1 0 0 0 0 0 0
0.25 2 0 0 0 0 0 0
0.333333333333333 6 0 0 0 0 0 0
0.416666666666667 1 0 0 0 0 0 0
0.5 6 0 0 0 0 0 0
0.583333333333333 3 0 0 0 0 0 0
0.666666666666667 3 0 0 0 0 0 0
0.75 3 0 0 0 0 0 0
0.833333333333333 1 0 0 0 0 0 0
0.916666666666667 1 0 0 0 0 0 0
1 275 0 0 0 0 0 0
1.5 2 0 0 0 0 0 0
2 308 0 0 0 0 0 0
3 246 0 0 0 0 0 0
4 233 0 0 0 0 0 0
5 242 0 0 0 0 0 0
6 0 241 0 0 0 0 0
7 0 256 0 0 0 0 0
8 0 239 0 0 0 0 0
9 0 245 0 0 0 0 0
10 0 214 0 0 0 0 0
11 0 0 220 0 0 0 0
12 0 0 224 0 0 0 0
13 0 0 191 0 0 0 0
14 0 0 199 0 0 0 0
15 0 0 206 0 0 0 0
16 0 0 0 186 0 0 0
17 0 0 0 164 0 0 0
18 0 0 0 141 0 0 0
19 0 0 0 130 0 0 0
20 0 0 0 149 0 0 0
21 0 0 0 0 158 0 0
22 0 0 0 0 149 0 0
23 0 0 0 0 125 0 0
24 0 0 0 0 144 0 0
25 0 0 0 0 107 0 0
26 0 0 0 0 100 0 0
27 0 0 0 0 117 0 0
28 0 0 0 0 85 0 0
29 0 0 0 0 82 0 0
30 0 0 0 0 82 0 0
31 0 0 0 0 0 68 0
32 0 0 0 0 0 84 0
33 0 0 0 0 0 78 0
34 0 0 0 0 0 58 0
35 0 0 0 0 0 58 0
36 0 0 0 0 0 33 0
37 0 0 0 0 0 46 0
38 0 0 0 0 0 45 0
39 0 0 0 0 0 45 0
40 0 0 0 0 0 32 0
41 0 0 0 0 0 34 0
42 0 0 0 0 0 26 0
43 0 0 0 0 0 31 0
44 0 0 0 0 0 24 0
45 0 0 0 0 0 27 0
46 0 0 0 0 0 25 0
47 0 0 0 0 0 16 0
48 0 0 0 0 0 21 0
49 0 0 0 0 0 15 0
50 0 0 0 0 0 12 0
51 0 0 0 0 0 0 13
52 0 0 0 0 0 0 7
53 0 0 0 0 0 0 4
54 0 0 0 0 0 0 6
55 0 0 0 0 0 0 9
56 0 0 0 0 0 0 7
57 0 0 0 0 0 0 9
58 0 0 0 0 0 0 6
59 0 0 0 0 0 0 5
60 0 0 0 0 0 0 4
61 0 0 0 0 0 0 2
62 0 0 0 0 0 0 1
63 0 0 0 0 0 0 5
64 0 0 0 0 0 0 1
65 0 0 0 0 0 0 5
66 0 0 0 0 0 0 3
67 0 0 0 0 0 0 2
68 0 0 0 0 0 0 1
69 0 0 0 0 0 0 3
70 0 0 0 0 0 0 1
72 0 0 0 0 0 0 0
73 0 0 0 0 0 0 0
76 0 0 0 0 0 0 0
84 0 0 0 0 0 0 0
<NA> 0 0 0 0 0 0 0
Categories
Numeric Values (70,100] <NA>
0 0 0
0.0833333333333333 0 0
0.25 0 0
0.333333333333333 0 0
0.416666666666667 0 0
0.5 0 0
0.583333333333333 0 0
0.666666666666667 0 0
0.75 0 0
0.833333333333333 0 0
0.916666666666667 0 0
1 0 0
1.5 0 0
2 0 0
3 0 0
4 0 0
5 0 0
6 0 0
7 0 0
8 0 0
9 0 0
10 0 0
11 0 0
12 0 0
13 0 0
14 0 0
15 0 0
16 0 0
17 0 0
18 0 0
19 0 0
20 0 0
21 0 0
22 0 0
23 0 0
24 0 0
25 0 0
26 0 0
27 0 0
28 0 0
29 0 0
30 0 0
31 0 0
32 0 0
33 0 0
34 0 0
35 0 0
36 0 0
37 0 0
38 0 0
39 0 0
40 0 0
41 0 0
42 0 0
43 0 0
44 0 0
45 0 0
46 0 0
47 0 0
48 0 0
49 0 0
50 0 0
51 0 0
52 0 0
53 0 0
54 0 0
55 0 0
56 0 0
57 0 0
58 0 0
59 0 0
60 0 0
61 0 0
62 0 0
63 0 0
64 0 0
65 0 0
66 0 0
67 0 0
68 0 0
69 0 0
70 0 0
72 1 0
73 3 0
76 1 0
84 1 0
<NA> 0 107NA値の再ラベル付け
NA 値に “Missing” などのラベルを付けたい場合があります。新しく作成した列は因子型(制限付き型)であるため、この値は拒否されるので、単純に replace_na() で変換させることはできません。代わりに、因子(ファクタ)型データ の章で説明されているように、forcats パッケージの fct_explicit_na() を使用してください。
linelist <- linelist %>%
# cut()すると、因子型のage_catが自動的に作成される
mutate(age_cat = cut(
age_years,
breaks = c(0, 5, 10, 15, 20, 30, 50, 70, 100),
right = FALSE,
include.lowest = TRUE,
labels = c("0-4", "5-9", "10-14", "15-19", "20-29", "30-49", "50-69", "70-100")),
# 欠損値を明示する
age_cat = fct_explicit_na(
age_cat,
na_level = "Missing age")
) # ラベルを指定することができるWarning: There was 1 warning in `mutate()`.
ℹ In argument: `age_cat = fct_explicit_na(age_cat, na_level = "Missing age")`.
Caused by warning:
! `fct_explicit_na()` was deprecated in forcats 1.0.0.
ℹ Please use `fct_na_value_to_level()` instead.# 各カテゴリに含まれる数値の数を表で表示する
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-49
1227 1223 1048 827 1216 848
50-69 70-100 Missing age <NA>
105 7 107 0 切れ目やラベルを素早く作る
ベクトルの改行やラベルを素早く作成するには、以下のような方法があります。seq() や rep() については、R の基礎 の章を参照してください。
# 0から90までのブレークポイントを5で割る
age_seq = seq(from = 0, to = 90, by = 5)
age_seq
# デフォルトのcut()の設定を前提に、上記のカテゴリのラベルを作成する
age_labels = paste0(age_seq + 1, "-", age_seq + 5)
age_labels
# 両方のベクトルが同じ長さであることを確認する
length(age_seq) == length(age_labels)cut() の詳細については、Rコンソールで ?cut と入力し、表示されるヘルプページを参照してください。
四分位数による分割
一般的な理解では、「クォンタイル(quantile)」または「パーセンタイル(percentile)」は、通常、下になる値の割合を指します。例えば、linelist の年齢の 95 パーセンタイルは、年齢の 95 %が該当する年齢になります。
しかし、一般的には、「四分位数」や「十分位数」は、データのグループを 4 つまたは 10 のグループに均等に分けたものを指すことがあります(グループよりも分割点が 1 つ多いことに注意してください)。
四分位数を取得するには、base R に含まれている stats の quantile() を使用します。データセットの列などの数値ベクトルと、0 から 1.0 までの確率の数値ベクトルを指定します。分割点は、数値ベクトルとして返されます。?quantile を入力すると、統計的手法の詳細を調べることができます。
入力された数値ベクトルに欠損値がある場合は、na.rm = TRUE を設定することをお勧めします。
names = FALSE を設定すると、名前のない数値ベクトルが得られます。
quantile(linelist$age_years, # 演算する数値ベクトルを指定
probs = c(0, .25, .50, .75, .90, .95), # 欲しいパーセンタイルを指定
na.rm = TRUE) # 欠損値を無視する 0% 25% 50% 75% 90% 95%
0 6 13 23 33 41 quantile() の結果は、age_categories() や cut() の分割点として使用できます。以下では、cut() を使って新しい列 deciles を作成し、age_years に対して quantiles() を使って区切りを定義しています。janitor の tabyl() を使って結果を表示し、各グループのパーセンテージを見ることができます(詳細は、記述統計表の作り方 の章を参照ください)。各グループ、正確に 10 %ではないことに注意してください。
linelist %>% #linelistから開始。
mutate(deciles = cut(age_years, # 新しい列を作る。 age_years列をcut()でdecileに格納。
breaks = quantile( # quantile()を使ってカットオフを定義する。
age_years, # age_yearsの操作。
probs = seq(0, 1, by = 0.1), # 0.0から1.0へ0.1倍。
na.rm = TRUE), # 欠損値を無視する
include.lowest = TRUE)) %>% # cut()でage 0を含む。
janitor::tabyl(deciles) # 表示するテーブルへのパイプ。 deciles n percent valid_percent
[0,2] 748 0.11319613 0.11505922
(2,5] 721 0.10911017 0.11090601
(5,7] 497 0.07521186 0.07644978
(7,10] 698 0.10562954 0.10736810
(10,13] 635 0.09609564 0.09767728
(13,17] 755 0.11425545 0.11613598
(17,21] 578 0.08746973 0.08890940
(21,26] 625 0.09458232 0.09613906
(26,33] 596 0.09019370 0.09167820
(33,84] 648 0.09806295 0.09967697
<NA> 107 0.01619249 NA均等な大きさのグループ
数値グループを作成するもう 1 つの方法として、dplyr の関数 ntile() で、データを n 個の均等な大きさのグループに分割する方法があります。quantile() と異なり、同じ値が複数のグループに入ることがあることに注意してください。この方法では、数値ベクトルを指定し、そして分割するグループの数を指定することが必要ですが、作成される新しい列の値は、グループの「番号」(例:1 から 10)だけで、cut() を使ったときのように値の範囲そのものではありません。
# ntile()でグループを作る
ntile_data <- linelist %>%
mutate(even_groups = ntile(age_years, 10))
# グループ別の数と割合の表を作る
ntile_table <- ntile_data %>%
janitor::tabyl(even_groups)
# 範囲を示すために最小値と最大値を添付する
ntile_ranges <- ntile_data %>%
group_by(even_groups) %>%
summarise(
min = min(age_years, na.rm=T),
max = max(age_years, na.rm=T)
)Warning: There were 2 warnings in `summarise()`.
The first warning was:
ℹ In argument: `min = min(age_years, na.rm = T)`.
ℹ In group 11: `even_groups = NA`.
Caused by warning in `min()`:
! no non-missing arguments to min; returning Inf
ℹ Run `dplyr::last_dplyr_warnings()` to see the 1 remaining warning.# 結合して表示する(値が複数のグループに存在することに注意)
left_join(ntile_table, ntile_ranges, by = "even_groups") even_groups n percent valid_percent min max
1 651 0.09851695 0.10013844 0 2
2 650 0.09836562 0.09998462 2 5
3 650 0.09836562 0.09998462 5 7
4 650 0.09836562 0.09998462 7 10
5 650 0.09836562 0.09998462 10 13
6 650 0.09836562 0.09998462 13 17
7 650 0.09836562 0.09998462 17 21
8 650 0.09836562 0.09998462 21 26
9 650 0.09836562 0.09998462 26 33
10 650 0.09836562 0.09998462 33 84
NA 107 0.01619249 NA Inf -Infcase_when()
dplyr の関数 case_when() を使って数値列からカテゴリを作成することは可能ですが、epikit のage_categories() や cut() を使った方が、自動的に順序付けられた要素を作成してくれるので、より簡単です。
case_when() を使用する場合は、この章の「値の再定義」のセクションで説明した方法を確認してください。また、右辺にくるすべての値は同じデータ型でなければならないことに注意してください。したがって、右辺の値を NA にしたい場合は、“Missing”と書くか、特別な NA 値である NA_character_ を使用する必要があります。
パイプチェーンへの追加
以下では、カテゴリ化された 2 つの年齢列を作成するコードを、前処理パイプラインに追加しています。
# パイプチェーン処理 (未加工データから始まり、クリーニングステップを経由してパイプで処理される)
##################################################################################
# パイプチェーンのクリーニング開始
###########################
linelist <- linelist_raw %>%
#列名の構文の標準化
janitor::clean_names() %>%
# 手動で列名を変更
# 新しい列名 # 既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# remove column
# 列を削除
select(-c(row_num, merged_header, x28)) %>%
# 重複除去
distinct() %>%
# 列の追加
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# convert class of columns
# 列のデータ型を変換
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# 列の追加:入院の遅延
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# 病院の列の値をクリーニングする
mutate(hospital = recode(hospital,
# 既存の値 = 新しい値
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# age_years列の作成(age 列と age_unit 列を使用する)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age)) %>%
# ここまでは、すでに説明したクリーニング手順
###################################################
mutate(
# 年齢別カテゴリ:カスタマイズする
age_cat = epikit::age_categories(age_years, breakers = c(0, 5, 10, 15, 20, 30, 50, 70)),
# 年齢カテゴリ:0~85歳までの5段階
age_cat5 = epikit::age_categories(age_years, breakers = seq(0, 85, 5)))8.10 行の追加
一行ずつ追加する
手作業で一行ずつ追加するのは面倒ですが、dplyr の add_row() を使えば可能です。各列は 1 つのデータ型(因子型、数字型、ロジカル型など)の値のみを含む必要があることを覚えておいてください。そのため、行の追加にはこれを維持するための微妙な調整が必要です。
linelist <- linelist %>%
add_row(row_num = 666,
case_id = "abc",
generation = 4,
`infection date` = as.Date("2020-10-10"),
.before = 2).before と .after. を使って、追加したい行の位置を指定します。.before = 3 は、新しい行を現在の 3 行目の前に配置します。デフォルトでは、新しい行は最後に追加されます。指定されていない列は空欄(NA)になります。
新しい行番号は奇妙に見えるかもしれません(“…23”)。既存の行の行番号は変更されているため、このコマンドを 2 回使用する場合は、慎重に検討・テストしてください。
指定したデータ型が実際のデータ型と異なる場合は、以下のようなエラーが表示されます。
Error: Can't combine ..1$infection date <date> and ..2$infection date <character>.(日付の値を持つ行を挿入する際には、as.Date("2020-10-10") のように as.Date() という関数で日付を囲うことを忘れないでください)。
複数行を結合する
あるデータフレームの行を別のデータフレームの下部に結合してデータセットを結合するには、dplyr の bind_rows() を使用します。これについては、データの結合 の章で詳しく説明しています。
8.11 行の抽出(フィルタリング)
列を整理し、値を再定義した後の典型的なクリーニングステップは、dplyr の filter() を使ってデータフレームの特定の行を抽出(フィルタリング)することです。
filter() では、データセット内の特定の行を抜き出すために、TRUE でなければならない論理を指定します。以下では、単純な論理条件と複雑な論理条件に基づいて行を抽出する方法を示します。
単純なフィルタリング
以下の単純な例では、論理的な条件を満たすように行を抽出した上で、linelist データフレームを再定義します。括弧内の論理条件が評価され、 TRUE となる行のみが残ります。
この例では、論理条件は gender == "f" で、列 gender の値が "f" に等しいかどうかを問いています(大文字と小文字は区別されます)。
フィルタを適用する前の linelist の行数は nrow(linelist) です。
linelist <- linelist %>%
filter(gender == "f") # 性別が "f" である行だけを残すフィルタを適用した後の linelist の行数は linelist %>% filter(gender == "f") %>% nrow() となります。
欠損値のフィルタリング
欠損値のある行をフィルタリングしたいことはよくあります。filter(!is.na(column) & !is.na(column)) と書きたい気持ちを抑えて、代わりにこのためにカスタマイズされた tidyr の関数である drop_na() を使いましょう。空の括弧をつけて実行すると、欠損値のある行を削除します。あるいは、欠落があるか評価したい特定の列の名前を指定したり、上述の “tidyselect”ヘルパー関数を使用することもできます。
linelist %>%
drop_na(case_id, age_years)
# case_idやage_yearsの値が欠損している行を削除する。データの欠落の分析や管理の詳細ついては、欠損データの処理 の章をご覧ください。
行番号によるフィルタリング
データフレームや Tibble では、各行には通常「行番号(row number)」があり、R Viewer で見ると最初の列の左側に表示されます。これは、それ自体はデータの列ではありませんが、filter() を使用する際に参照することができます。
「行番号」に基づいてフィルタリングしたい場合は、フィルタリングの論理条件のとして、dplyr の関数 row_number() を空括弧付きで使用します。多くの場合、以下のように、論理条件文の一部として、%in% 演算子を使用して数値の範囲を指定します。最初の N 行を表示するには、特別な dplyr 関数 head() を使用することもできます。
# 最初の100行を表示する
linelist %>% head(100)
# またはtail()を使って最後のn行を見る
# 5列目のみ表示する
linelist %>% filter(row_number() == 5)
# 2行目から20行目までと、3つの特定の列を見る
linelist %>% filter(row_number() %in% 2:20) %>% select(date_onset, outcome, age)また、データフレームを tibble 関数 rownames_to_column() にパイプすることで、行番号を列として変換することができます(括弧の中には何も入れないでください)。
複雑なフィルタリング
括弧 ()、OR |、ネゲート !、%in%、AND & の各演算子を使って、より複雑な論理条件を作ることができます。その例を以下に示します。
注: 論理的条件の前に ! 演算子を使うと、その後の論理的条件が否定されます。例えば、!is.na(column) は、列の値が欠損していない場合に真(TRUE)と評価されます。同様に、!column %in% c("a", "b", "c") は、列の値がベクトルに含まれていない場合に真(TRUE)と評価されます。
データを調べる
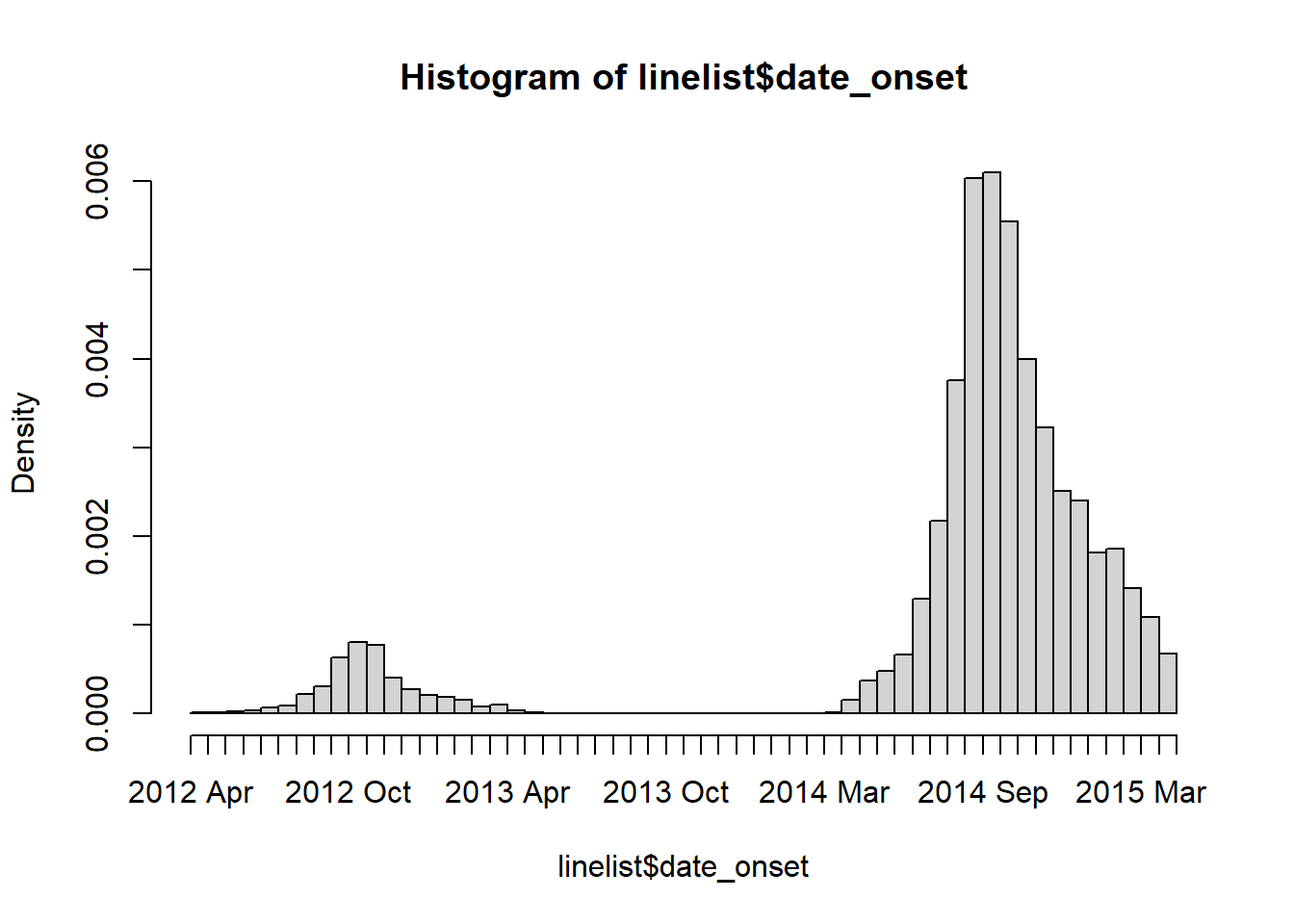
以下は、発症日のヒストグラムを作成するためのシンプルなコマンドです。この未加工データセットには、二つのアウトブレイクが含まれていることはわかります(2012~2013 年に発生した小規模なアウトブレイクと 2014~2015 年に発生した大規模なアウトブレイク)。今回の分析では、最初の小規模なアウトブレイクをラインリストから削除していきます。
数値や日付の欠損をフィルターで処理する方法
date_onset 列で 2013 年 6 月以降の行を抽出することはできるでしょうか。これには、注意が必要です。filter(date_onset > as.Date("2013-06-01"))) というコードを適用すると、発症日が欠落している後期流行の行がすべて削除されてしまいます!
警告:日付や数値の「大(>)」または「小(<)」にフィルタリングすると、欠損値(NA)のある行が削除されてしまいます。これは、NA が無限に大きい、または小さいものとして扱われるためです。
(日付データと lubridate パッケージを使用した処理については、日付データの章の 日付の操作のセクションを参照してください。)
フィルターを作成する
クロス集計を行い、正しい行だけを除外していることを確認して下さい。
table(Hospital = linelist$hospital, #病院名
YearOnset = lubridate::year(linelist$date_onset), #発症した年
useNA = "always") #欠損値の表示 YearOnset
Hospital 2012 2013 2014 2015 <NA>
Central Hospital 0 0 351 99 18
Hospital A 229 46 0 0 15
Hospital B 227 47 0 0 15
Military Hospital 0 0 676 200 34
Missing 0 0 1117 318 77
Other 0 0 684 177 46
Port Hospital 9 1 1372 347 75
St. Mark's Maternity Hospital (SMMH) 0 0 322 93 13
<NA> 0 0 0 0 0データセットから最初のアウトブレイク(2012~2013 年に発生したアウトブレイク)を除外するために、他にどのような基準でフィルタリングできるでしょうか。次のことがわかりました。
2012 年と 2013 年に発生した最初のアウトブレイクは、A 病院(Hospital A)と B 病院(Hospital B)で発生し、ポート病院(Port Hospital)でも10件の症例が確認されました。
2 回目のアウトブレイク(2014~2015 年に発生したアウトブレイク)では、A 病院(Hospital A)と B 病院(Hospital B)には症例は確認されませんでしたが、ポート病院(Port Hospital)には症例が確認されました。
除外したい項目:
-
A 病院(Hospital A)、 B 病院(Hospital B)、ポート病院(Port Hospital)のいずれかで 2012 年と 2013 年に発症した
nrow(linelist %>% filter(hospital %in% c("Hospital A", "Hospital B") | date_onset < as.Date("2013-06-01")))の行。2012 年および 2013 年に発症した
nrow(linelist %>% filter(date_onset < as.Date("2013-06-01"))の行を除外する。nrow(linelist %>% filter(hospital %in% c('Hospital A', 'Hospital B') & is.na(date_onset)))発症日が欠落している A 病院および B 病院の行を除外する。nrow(linelist %>% filter(!hospital %in% c('Hospital A', 'Hospital B') & is.na(date_onset))発症日が見つからない他の行は除外しない。
6608 のラインリストから始めます。以下がフィルタリングのコードです。
linelist <- linelist %>%
# 発症日が2013年6月1日以降の行、または発症日が不明でA病院またはB病院以外の病院であった行を残す
filter(date_onset > as.Date("2013-06-01") | (is.na(date_onset) & !hospital %in% c("Hospital A", "Hospital B")))
nrow(linelist)[1] 6019再度クロス集計を行うと、A 病院と B 病院が完全に取り除かれ、2012 年と 2013 年のポート病院で発生した 10 件が取り除かれ、その他の値はすべて同じになっていることがわかります。
table(Hospital = linelist$hospital, # 病院名
YearOnset = lubridate::year(linelist$date_onset), # 発症した年
useNA = "always") # 欠損値の表示 YearOnset
Hospital 2014 2015 <NA>
Central Hospital 351 99 18
Military Hospital 676 200 34
Missing 1117 318 77
Other 684 177 46
Port Hospital 1372 347 75
St. Mark's Maternity Hospital (SMMH) 322 93 13
<NA> 0 0 0複数のステートメントを 1 つの filter() コマンドの中に含めることができます(カンマで区切ってください)。または、分かりやすくするために、常に別の filter() コマンドにパイプすることもできます。
注:date_hospitalisation には欠損値がないことから、読者の中には date_hospitalisation でフィルタリングする方が簡単だと思われる方もいるかもしれません。これは事実です。ここでは、複雑なフィルタを理解するために date_onset を使用しました。
独立型
フィルタリングは、(パイプチェーンの一部ではなく)独立したコマンドとしても実行できます。他の dplyr 系の関数と同様に、この場合、一番最初の引数はデータセットそのものでなければなりません。
# dataframe <- filter(dataframe, condition(s) for rows to keep)
linelist <- filter(linelist, !is.na(case_id))base Rを使う場合、保持したい [行、列] を、角括弧 [] を使ってサブセット(フィルタリング)することもできます。
# dataframe <- dataframe[row conditions, column conditions] (blank means keep all)
linelist <- linelist[!is.na(case_id), ]データを素早く確認
数少ない行や列のデータを素早く確認したいことがよくあります。base R の View() を使用すると、RStudio データフレームが表示されます。
以下のコマンドを実行すると、RStudio でラインリストが表示されます。
View(linelist)特定のセル(特定の行、特定の列)を表示する 2 つの例を紹介します。
dplyrの関数 filter() と select() を使用する
View() の中で、データセットを filter() にパイプして特定の行を残し、select() にパイプして特定の列を残します。例えば、特定の 3 つの症例の発症日と入院日を確認する場合です。
View(linelist %>%
filter(case_id %in% c("11f8ea", "76b97a", "47a5f5")) %>%
select(date_onset, date_hospitalisation))base R でも [ ] を使用してサブセットすると、同じことができます。
View(linelist[linelist$case_id %in% c("11f8ea", "76b97a", "47a5f5"), c("date_onset", "date_hospitalisation")])パイプチェーンに追加
# パイプチェーン処理 (未加工データから始まり、クリーニングステップを経由してパイプで処理される)
##################################################################################
# パイプチェーンのクリーニング開始
###########################
linelist <- linelist_raw %>%
#列名構文の標準化
janitor::clean_names() %>%
# 手動で列名を変更
#新しい列名 # 既存の列名
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# 列の除去
select(-c(row_num, merged_header, x28)) %>%
# 重複除去
distinct() %>%
# 列を追加する
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# 列のデータ型を変換
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# 列の追加: 入院までの時間
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# 病院列の値を整える
mutate(hospital = recode(hospital,
# OLD = NEW
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# age_years 列を作成する (age列とage_unit列から)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age)) %>%
mutate(
# 年齢別カテゴリ:カスタマイズする
age_cat = epikit::age_categories(age_years, breakers = c(0, 5, 10, 15, 20, 30, 50, 70)),
# 年齢カテゴリ:0~85歳までの5段階
age_cat5 = epikit::age_categories(age_years, breakers = seq(0, 85, 5)))%>%
# ここまでは、すでに説明したクリーニング手順
###################################################
filter(
# case_idが欠落していない行のみを残す
!is.na(case_id),
# 2回目のアウトブレイクだけを残すようにフィルタリングする
date_onset > as.Date("2013-06-01") | (is.na(date_onset) & !hospital %in% c("Hospital A", "Hospital B")))8.12 行単位の計算
行内で計算を行いたい場合は、dplyr の rowwise() を利用することができます。例えば、以下のコードでは rowwise() を使用し、ラインリストの各行について、値が “yes” である指定された症状の列の数を合計する新しい列を作成しています。rowwise() は本質的には特殊な group_by() であるため、終わったら ungroup() を使うのがベストです(詳細は、データのグループ化 の章を参照ください)。
linelist %>%
rowwise() %>%
mutate(num_symptoms = sum(c(fever, chills, cough, aches, vomit) == "yes")) %>%
ungroup() %>%
select(fever, chills, cough, aches, vomit, num_symptoms) # 表示のため# A tibble: 5,888 × 6
fever chills cough aches vomit num_symptoms
<chr> <chr> <chr> <chr> <chr> <int>
1 no no yes no yes 2
2 <NA> <NA> <NA> <NA> <NA> NA
3 <NA> <NA> <NA> <NA> <NA> NA
4 no no no no no 0
5 no no yes no yes 2
6 no no yes no yes 2
7 <NA> <NA> <NA> <NA> <NA> NA
8 no no yes no yes 2
9 no no yes no yes 2
10 no no yes no no 1
# ℹ 5,878 more rows評価する列を指定する際に、この章の select() セクションで説明されている “tidyselect” ヘルパー関数を使用したいと思うかもしれません。その場合は、1 つだけ調整が必要です( select() や summarise() のような dplyr 関数内で使用していないため)。
列指定の基準を dplyr の c_across() の中に入れます。これは、c_across(ドキュメントはこちら)が、特に rowwise() と連動するように設計されているからです。例えば、次のようなコードです。
rowwise()を適用して、各行の中で以下の演算(sum())が適用されます(列全体の合計ではありません)。新しい列
num_NA_datesを作成します。これは、各行について、is.na()が TRUE と評価された列 (列名に “date” を含むもの) の数として定義されます(これらはデータが欠損しています)。ungroup()により、後続のステップでrowwise()の影響を取り除きます。
linelist %>%
rowwise() %>%
mutate(num_NA_dates = sum(is.na(c_across(contains("date"))))) %>%
ungroup() %>%
select(num_NA_dates, contains("date")) # for display# A tibble: 5,888 × 5
num_NA_dates date_infection date_onset date_hospitalisation date_outcome
<int> <date> <date> <date> <date>
1 1 2014-05-08 2014-05-13 2014-05-15 NA
2 1 NA 2014-05-13 2014-05-14 2014-05-18
3 1 NA 2014-05-16 2014-05-18 2014-05-30
4 1 2014-05-04 2014-05-18 2014-05-20 NA
5 0 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 0 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 0 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 0 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 1 NA 2014-06-05 2014-06-06 2014-06-18
10 1 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rowsまた、max() のような他の関数を使用し、各行の最新または直近の日付を取得することもできます。
linelist %>%
rowwise() %>%
mutate(latest_date = max(c_across(contains("date")), na.rm=T)) %>%
ungroup() %>%
select(latest_date, contains("date")) # for display# A tibble: 5,888 × 5
latest_date date_infection date_onset date_hospitalisation date_outcome
<date> <date> <date> <date> <date>
1 2014-05-15 2014-05-08 2014-05-13 2014-05-15 NA
2 2014-05-18 NA 2014-05-13 2014-05-14 2014-05-18
3 2014-05-30 NA 2014-05-16 2014-05-18 2014-05-30
4 2014-05-20 2014-05-04 2014-05-18 2014-05-20 NA
5 2014-05-29 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 2014-05-24 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 2014-06-01 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 2014-06-07 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 2014-06-18 NA 2014-06-05 2014-06-06 2014-06-18
10 2014-06-09 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rows8.13 並び替え(アレンジとソート)
dplyr の arrange()を使用し、列の値で行を並べ替えたり、順番に並べたりします。
単純に、並び替えたい順に列を列挙します。データに適用されたグループ化を最初並べ替えたい場合は、.by_group = TRUE と指定します(データのグループ化 の章を参照)。
デフォルトでは、列は「昇順」で並び替えされます(これは、数字や文字の列にも適用されます)。変数を desc() で囲むと、「降順」で並び替えできます。
arrange() によるデータの並び替えは、プレゼンテーション用のテーブルを作成するとき や、slice() を使ってグループごとに「上位」の行を抽出するとき、あるいは因子レベルの順序を出現順に設定するときなどに特に便利です。
例えば、linelist デーフレームの行を病院別(hospital 列)に並び替え、次に出現日(date_onset 列)の降順で並び替える場合は、次のようになります。
linelist %>%
arrange(hospital, desc(date_onset))