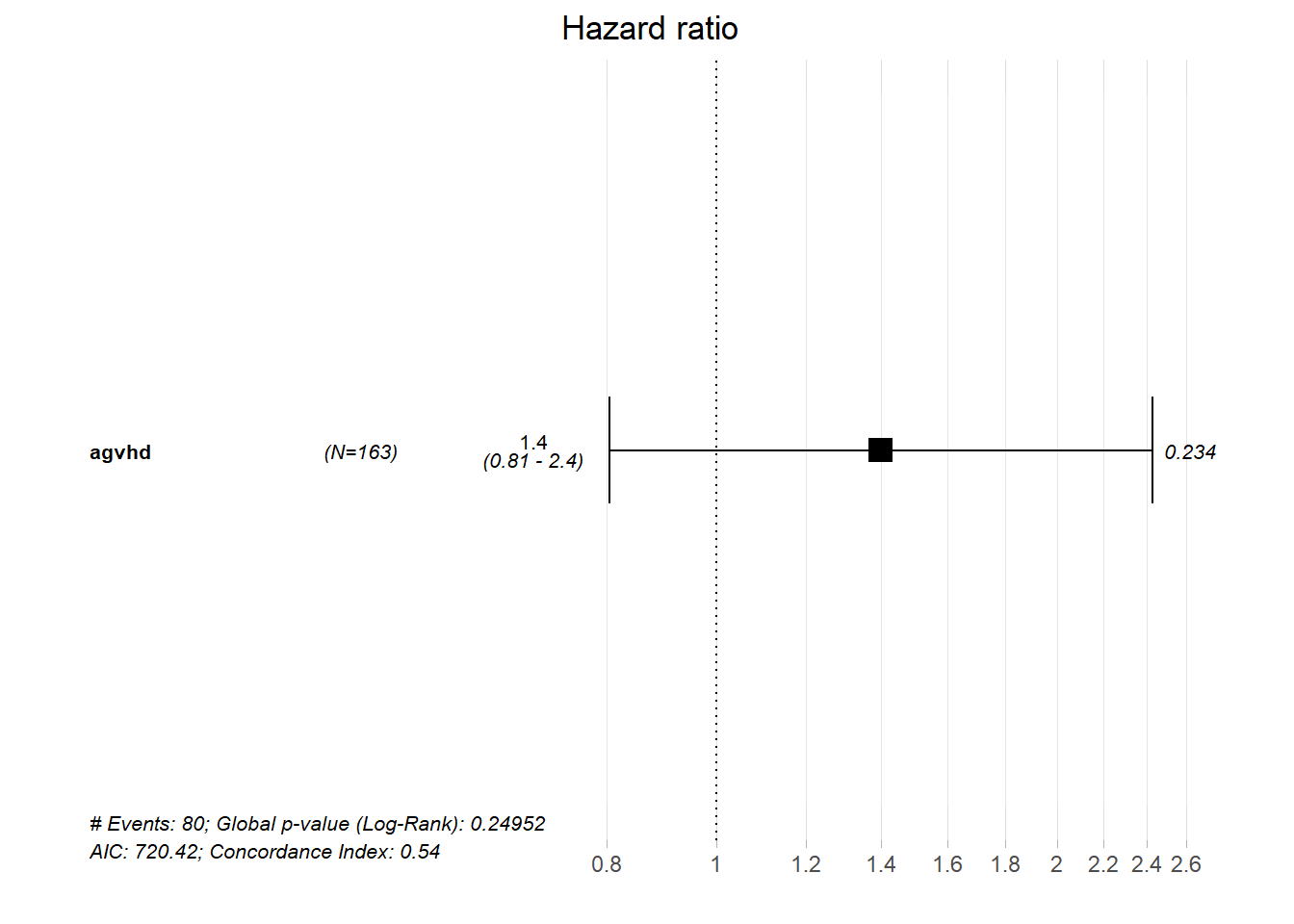27 Анализ выживаемости
27.1 Обзор
Анализ выживаемости фокусируется на описании для отдельного лица или группы лиц определенной точки событий, называемой происшествие (возникновение заболевания, излечение от заболевания, смерть, рецидив после ответа на лечение…), которая происходит в период времени, называемый время происшествия (или время отслеживания в когортных/популяционных исследованиях), во время которого ведется наблюдение за лицами. Чтобы определить время происшествия, необходимо определить отправную точку (это может быть дата включения, дата постановки диагноза…).
В этом случае объектом вывода при анализе выживаемости является время между отправной точкой и событием. В современных медицинских исследованиях оно широко используется, например, в клинических исследованиях для оценки эффекта лечения или в онкологической эпидемиологии для оценки большого числа показателей выживаемости при раке.
Как правило, выражается в вероятности выживания, которая является вероятностью того, что интересующее событие не возникло до длительности t.
Цензурировуание: Цензурирование возникает в том случае, если в конце наблюдения у некоторых индивидуумов не произошло интересующее событие, и, таким образом, истинное время наступления события неизвестно. Здесь мы в основном рассматриваем правое цензурирование, но более подробную информацию о цензурировании и анализе выживаемости в целом можно найти в разделе Ресурсы.
27.2 Подготока
Загрузка пакетов
Чтобы выполнить анализ выживаемости в R, одним из наиболее часто используемых пакетов является пакет survival. Сначала мы его установим, затем загрузим его и другие пакеты, которые используются в данном разделе:
В данном руководстве мы фокусируемся на использовании p_load() из пакета pacman, которая устанавливает пакет, если необходимо, и загружает его для использования. Вы можете также загрузить установленные пакеты с помощью library() из базового R. См. страницу Основы R для получения дополнительной информации о пакетах R.
На этой странице мы рассмотрим анализ выживаемости на примере построчного списка, использованного на большинстве предыдущих страниц, который мы немного изменим, чтобы иметь надлежащие данные о выживаемости.
Импорт набора данных
Мы импортируем набор данных о случаях из имитированной эпидемии Эболы. Если вы хотите выполнять действия параллельно, кликните, чтобы скачать “чистый” построчный список (как файл .rds). Импортируйте данные с помощью функции import() из пакета rio (она работает с разными типами файлов, такиим как .xlsx, .csv, .rds - см. детальную информацию на странице Импорт и экспорт).
# импортируем построчный список
linelist_case_data <- rio::import("linelist_cleaned.rds")Управление данными и преобразования
Если говорить кратко, данные о выживаемости можно описать как имеющие следующие три характеристики:
- зависимая переменная или отклик - время ожидания до возникновения хорошо определенного события,
- наблюдения цензурированы в том смысле, что для некоторых единиц интересующее событие не возникло на момент анализа данных, и
- существуют предикторы или независимые переменные, чей эффект на время ожидания мы хотим оценить или контролировать
Следовательно, мы создаем разные переменные, необходимые для соблюдения этой структуры и выполнения анализа выживаемости.
Мы определяем:
- новый датафрейм
linelist_survдля этого анализа
- интересующее нас событие, как “смерть” (следовательно, вероятность выживания будет вероятностью того, что человек жив через определнный промежуток времени после отправной точки),
- время отслеживания (
futime) как время от возникновения до времени исхода в днях, - цензурированных пациентов, каак тех, кто выздоровел или по кому исход неизвестен, т.е. событие “смерть” не наблюдалось (
event=0).
ВНИМАНИЕ: Поскольку в реальном когортном исследовании информация об отправной точке и окончании отслеживания известна, поскольку люди находятся под наблюдением, мы будем удалять наблюдения, в которых дата возникновения или дата исхода неизвестна. Также будут удалены случаи, когда дата возникновения заболевания позже даты исхода, так как они считаются ошибочными.
СОВЕТ: Учитывая, что фильтрация по дате больше чем (>) или меньше чем (<) позволяет удалить строки с пропущенными значениями, применение фильтра к неправильным датам также приведет к удалению строк с пропущенными датами.
Затем используем case_when(), чтобы создать столбец age_cat_small, в котором будет только 3 возрастных категории.
#создаем новые данные под названием linelist_surv из linelist_case_data
linelist_surv <- linelist_case_data %>%
dplyr::filter(
# удаляем наблюдения с неправильными или отсутствующими датами возникновения или датой исхода
date_outcome > date_onset) %>%
dplyr::mutate(
# создаем переменную event (событие), которая будет равна 1, если пациент умер и 0, если было применено правое цензурирование
event = ifelse(is.na(outcome) | outcome == "Recover", 0, 1),
# создаем переменную по времени отслеживания в днях
futime = as.double(date_outcome - date_onset),
# создаем новую переменную возрастной категории только с 3 уровнями страт
age_cat_small = dplyr::case_when(
age_years < 5 ~ "0-4",
age_years >= 5 & age_years < 20 ~ "5-19",
age_years >= 20 ~ "20+"),
# на предыдущем шаге переменная age_cat_small была создана как текстовая.
# теперь конвертируем в фактор и задаем уровни
# Обратите внимание, что значения NA остаются NA, а не размещаются, например, в уровень "unknown" (неизвестно),
# поскольку при следующем анализе их нужно будет убрать
age_cat_small = fct_relevel(age_cat_small, "0-4", "5-19", "20+")
)СОВЕТ: Мы можем верифицировать новые столбцы, которые мы создали, посмотрев суммарную статистику по futime и кросс-табуляцию между event и outcome, из которых они были созданы. Кроме этой верификации полезной привычкой будет сообщение медианного времени отслеживания при интерпретации результатов анализа выживаемости.
summary(linelist_surv$futime) Min. 1st Qu. Median Mean 3rd Qu. Max.
1.00 6.00 10.00 11.98 16.00 64.00 # кросс-табуляция новой переменной event и переменной outcome, из которой она была создана
# чтобы проверить, что код сделал то, что нужно
linelist_surv %>%
tabyl(outcome, event) outcome 0 1
Death 0 1952
Recover 1547 0
<NA> 1040 0Теперь проведем кросс-табуляцию новой переменной age_cat_small и старого столбца age_cat, чтобы убедиться в правильности присваивания
linelist_surv %>%
tabyl(age_cat_small, age_cat) age_cat_small 0-4 5-9 10-14 15-19 20-29 30-49 50-69 70+ NA_
0-4 834 0 0 0 0 0 0 0 0
5-19 0 852 717 575 0 0 0 0 0
20+ 0 0 0 0 862 554 69 5 0
<NA> 0 0 0 0 0 0 0 0 71Рассмотрим первые 10 наблюдений из данных linelist_surv, рассматривая конкретные переменные (включая те, которые только что созданы).
linelist_surv %>%
select(case_id, age_cat_small, date_onset, date_outcome, outcome, event, futime) %>%
head(10) case_id age_cat_small date_onset date_outcome outcome event futime
1 8689b7 0-4 2014-05-13 2014-05-18 Recover 0 5
2 11f8ea 20+ 2014-05-16 2014-05-30 Recover 0 14
3 893f25 0-4 2014-05-21 2014-05-29 Recover 0 8
4 be99c8 5-19 2014-05-22 2014-05-24 Recover 0 2
5 07e3e8 5-19 2014-05-27 2014-06-01 Recover 0 5
6 369449 0-4 2014-06-02 2014-06-07 Death 1 5
7 f393b4 20+ 2014-06-05 2014-06-18 Recover 0 13
8 1389ca 20+ 2014-06-05 2014-06-09 Death 1 4
9 2978ac 5-19 2014-06-06 2014-06-15 Death 1 9
10 fc15ef 5-19 2014-06-16 2014-07-09 Recover 0 23Мы также можем провести кросс-табуляцию столбцов age_cat_small и gender, чтобы получить дополнительную информацию о распределении этого нового столбца по полу. Мы используем tabyl() и функции adorn из janitor, как описано на странице [Описательные таблицы].
linelist_surv %>%
tabyl(gender, age_cat_small, show_na = F) %>%
adorn_totals(where = "both") %>%
adorn_percentages() %>%
adorn_pct_formatting() %>%
adorn_ns(position = "front") gender 0-4 5-19 20+ Total
f 482 (22.4%) 1,184 (54.9%) 490 (22.7%) 2,156 (100.0%)
m 325 (15.0%) 880 (40.6%) 960 (44.3%) 2,165 (100.0%)
Total 807 (18.7%) 2,064 (47.8%) 1,450 (33.6%) 4,321 (100.0%)27.3 Основы анализа выживаемости
Построение объекта типа surv
Сначала мы используем Surv() из survival, чтобы построить объект выживаемости (survival) из столбцов времени отслеживания и события.
В результате такого шага создается объект типа Surv, объединяющий информацию о времени и о том, наблюдалось ли интересующее нас событие (смерть). Этот объект в конечном итоге будет использоваться в правой части последующих формул модели (см. документацию).
# используем синтаксис Suv() для данных с правым цензурированием
survobj <- Surv(time = linelist_surv$futime,
event = linelist_surv$event)Для рассмотрения представим первые 10 строк данных linelist_surv, и рассмотрим только некоторые важные столбцы.
linelist_surv %>%
select(case_id, date_onset, date_outcome, futime, outcome, event) %>%
head(10) case_id date_onset date_outcome futime outcome event
1 8689b7 2014-05-13 2014-05-18 5 Recover 0
2 11f8ea 2014-05-16 2014-05-30 14 Recover 0
3 893f25 2014-05-21 2014-05-29 8 Recover 0
4 be99c8 2014-05-22 2014-05-24 2 Recover 0
5 07e3e8 2014-05-27 2014-06-01 5 Recover 0
6 369449 2014-06-02 2014-06-07 5 Death 1
7 f393b4 2014-06-05 2014-06-18 13 Recover 0
8 1389ca 2014-06-05 2014-06-09 4 Death 1
9 2978ac 2014-06-06 2014-06-15 9 Death 1
10 fc15ef 2014-06-16 2014-07-09 23 Recover 0Вот первые 10 элементов survobj. Они печатаются по сути как вектор времени отслеживания с “+”, который отражает, было ли проведено правое цензурирование для наблюдения. Посмотрите, как соотносятся числа выше и ниже.
#печать первых 50 элементов вектора, чтобы посмотреть, как он выглядит
head(survobj, 10) [1] 5+ 14+ 8+ 2+ 5+ 5 13+ 4 9 23+Проведение начального анализа
Затем мы начинаем наш анализ с использования функции survfit() для создания объекта survfit, который создает расчеты по умолчанию для оценок Каплана Мейера (КМ) для общей (предельной) кривой выживания, которые по факту являются ступенчатой функцией со скачками во время наблюдаемого события. Итоговый объект survfit содержит одну или более кривых выживания и создается, используя объект Surv как переменную отклика в формуле модели.
ПРИМЕЧАНИЕ: Оценка Каплана-Мейера представляет собой непараметрическую оценку максимального правдоподобия функции выживания. (см. дополнительную информацию в Ресурсах).
Сводная информауия по этому объекту survfit выдаст то, что называется таблица вероятности дожития. Для каждого временного шага отслеживания (time), когда произошло событие (в возрастающем порядке):
- количество людей, находящихся в группе риска по развитию события (люди, у которых событие еще не наступило и которые не подвергались цензурированию:
n.risk)
- те, у кого произошло событие (
n.event)
- и из указанных выше: вероятность не развить событие (вероятность не умереть или выжить дольше определенного времени)
- наконец, рассчитывается и отображается стандартная ошибка и доверительный интервал для этой вероятности
Мы строим оценки КМ по формуле, где объект, ранее являвшийся Surv, “survobj”, является переменной отклика. “~ 1” уточняет, что мы выполняем модель для общей выживаемости.
# строим оценки КМ, используя формулу, где объект Surv "survobj" является переменной отклика.
# "~ 1" указывает, что мы выполняем модель для общей выживаемости
linelistsurv_fit <- survival::survfit(survobj ~ 1)
#печать сводной информации для большей детальности
summary(linelistsurv_fit)Call: survfit(formula = survobj ~ 1)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
1 4539 30 0.993 0.00120 0.991 0.996
2 4500 69 0.978 0.00217 0.974 0.982
3 4394 149 0.945 0.00340 0.938 0.952
4 4176 194 0.901 0.00447 0.892 0.910
5 3899 214 0.852 0.00535 0.841 0.862
6 3592 210 0.802 0.00604 0.790 0.814
7 3223 179 0.757 0.00656 0.745 0.770
8 2899 167 0.714 0.00700 0.700 0.728
9 2593 145 0.674 0.00735 0.660 0.688
10 2311 109 0.642 0.00761 0.627 0.657
11 2081 119 0.605 0.00788 0.590 0.621
12 1843 89 0.576 0.00809 0.560 0.592
13 1608 55 0.556 0.00823 0.540 0.573
14 1448 43 0.540 0.00837 0.524 0.556
15 1296 31 0.527 0.00848 0.511 0.544
16 1152 48 0.505 0.00870 0.488 0.522
17 1002 29 0.490 0.00886 0.473 0.508
18 898 21 0.479 0.00900 0.462 0.497
19 798 7 0.475 0.00906 0.457 0.493
20 705 4 0.472 0.00911 0.454 0.490
21 626 13 0.462 0.00932 0.444 0.481
22 546 8 0.455 0.00948 0.437 0.474
23 481 5 0.451 0.00962 0.432 0.470
24 436 4 0.447 0.00975 0.428 0.466
25 378 4 0.442 0.00993 0.423 0.462
26 336 3 0.438 0.01010 0.419 0.458
27 297 1 0.436 0.01017 0.417 0.457
29 235 1 0.435 0.01030 0.415 0.455
38 73 1 0.429 0.01175 0.406 0.452При использовании summary() мы можем добавить опцию times и уточнить определенное время, для которого нам нужна информация о выживаемости
#печатаем сводную информацию на конкретные моменты времени
summary(linelistsurv_fit, times = c(5,10,20,30,60))Call: survfit(formula = survobj ~ 1)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
5 3899 656 0.852 0.00535 0.841 0.862
10 2311 810 0.642 0.00761 0.627 0.657
20 705 446 0.472 0.00911 0.454 0.490
30 210 39 0.435 0.01030 0.415 0.455
60 2 1 0.429 0.01175 0.406 0.452Мы можем также использовать функцию print(). Аргумент print.rmean = TRUE используется, чтобы получить среднее время выживания и его стандартную ошибку (se).
ПРИМЕЧАНИЕ: Ограниченное среднее время выживания (RMST) - это специфическая мера выживаемости, которая все чаще используется в анализе выживаемости при раке и часто определяется как площадь под кривой выживания, если мы наблюдаем пациентов до ограниченного времени T (подробнее в разделе “Ресурсы”)..
# печать объекта linelistsurv_fit со средним временем выживания и его стандартной ошибкой
print(linelistsurv_fit, print.rmean = TRUE)Call: survfit(formula = survobj ~ 1)
n events rmean* se(rmean) median 0.95LCL 0.95UCL
[1,] 4539 1952 33.1 0.539 17 16 18
* restricted mean with upper limit = 64 СОВЕТ: Мы можем создать объект surv напрямую в функции survfit() и сохранить строку кода. Это будет выглядеть следующим образом: linelistsurv_quick <- survfit(Surv(futime, event) ~ 1, data=linelist_surv).
Совокупный риск
Кроме функции summary() мы также можем использовать функцию str(), которая дает больше деталей о структуре объекта survfit(). Это список из 16 элементов.
Среди этих элементов есть важный: cumhaz, который является числовым вектором. С ним можно построить график, чтобы показать совокупный моментный риск, где моментный риск - это моментнй коэффициент возникновения события (см. Ресурсы).
str(linelistsurv_fit)List of 16
$ n : int 4539
$ time : num [1:59] 1 2 3 4 5 6 7 8 9 10 ...
$ n.risk : num [1:59] 4539 4500 4394 4176 3899 ...
$ n.event : num [1:59] 30 69 149 194 214 210 179 167 145 109 ...
$ n.censor : num [1:59] 9 37 69 83 93 159 145 139 137 121 ...
$ surv : num [1:59] 0.993 0.978 0.945 0.901 0.852 ...
$ std.err : num [1:59] 0.00121 0.00222 0.00359 0.00496 0.00628 ...
$ cumhaz : num [1:59] 0.00661 0.02194 0.05585 0.10231 0.15719 ...
$ std.chaz : num [1:59] 0.00121 0.00221 0.00355 0.00487 0.00615 ...
$ type : chr "right"
$ logse : logi TRUE
$ conf.int : num 0.95
$ conf.type: chr "log"
$ lower : num [1:59] 0.991 0.974 0.938 0.892 0.841 ...
$ upper : num [1:59] 0.996 0.982 0.952 0.91 0.862 ...
$ call : language survfit(formula = survobj ~ 1)
- attr(*, "class")= chr "survfit"Построение кривых Каплана-Мейера
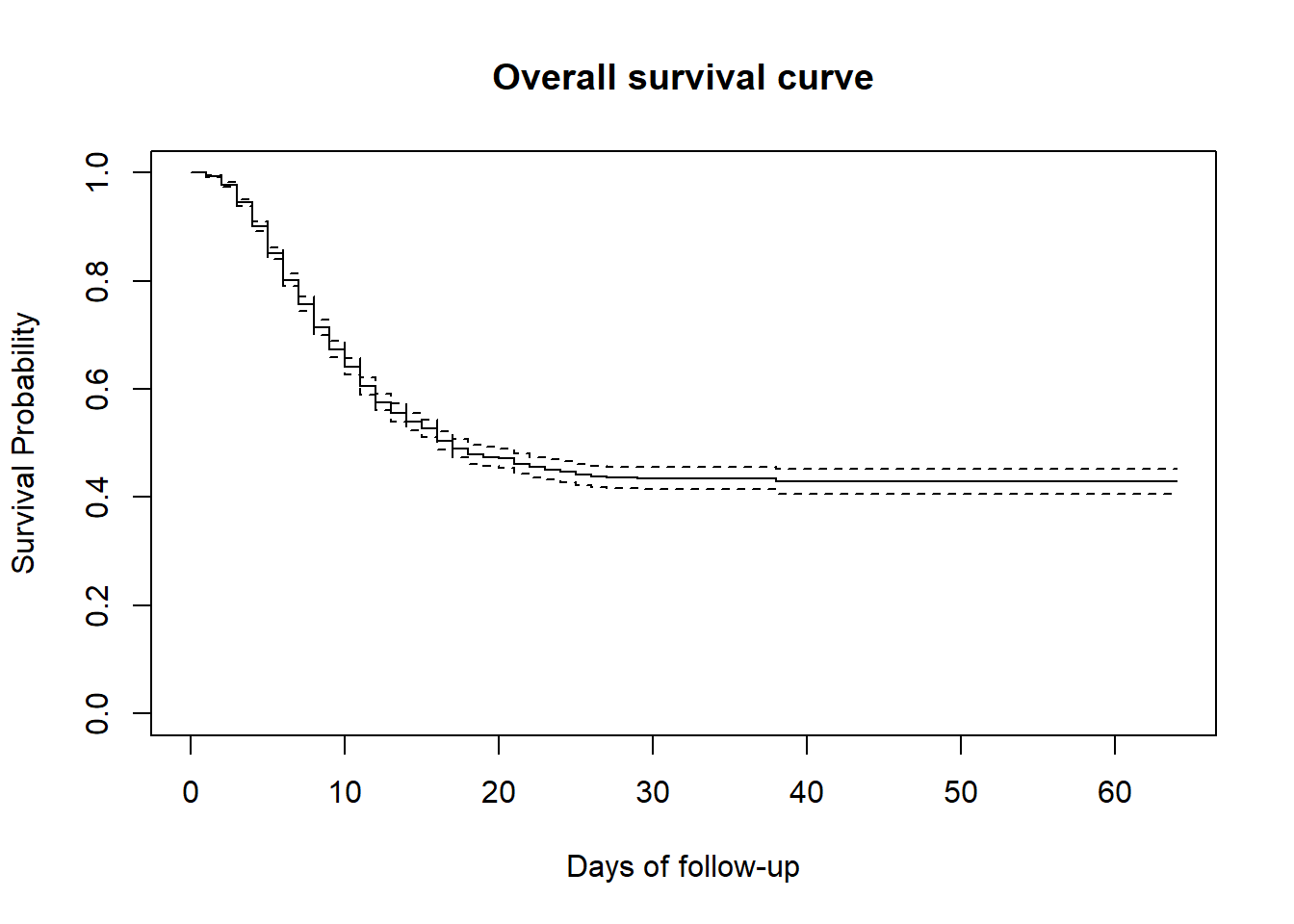
Как только построены оценки КМ, мы можем визуализировать вероятность быть живым через определенное время, используя базовую функцию plot(), которая рисует “Кривую Каплана-Мейера”. Иными словами, кривая ниже является традиционной иллюстрацией опыта выживания в двух полных группах пациентов.
Мы можем быстро верифицировать минимум и максимум времени отслеживания на кривой.
Простой способ интерпретации заключается в том, что в нулевой момент времени все участники еще живы, поэтому вероятность выживания равна 100%. С течением времени эта вероятность уменьшается, поскольку пациенты умирают. Доля участников, выживших после 60 дней отслеживания, составляет около 40%.
plot(linelistsurv_fit,
xlab = "Days of follow-up", # подпись оси x
ylab="Survival Probability", # подпись оси y
main= "Overall survival curve" # заголовок рисунка
)Доверительный интервал оценок выживаемости КМ также строится по умолчанию и может быть отменен добавлением опции conf.int = FALSE в команду plot().
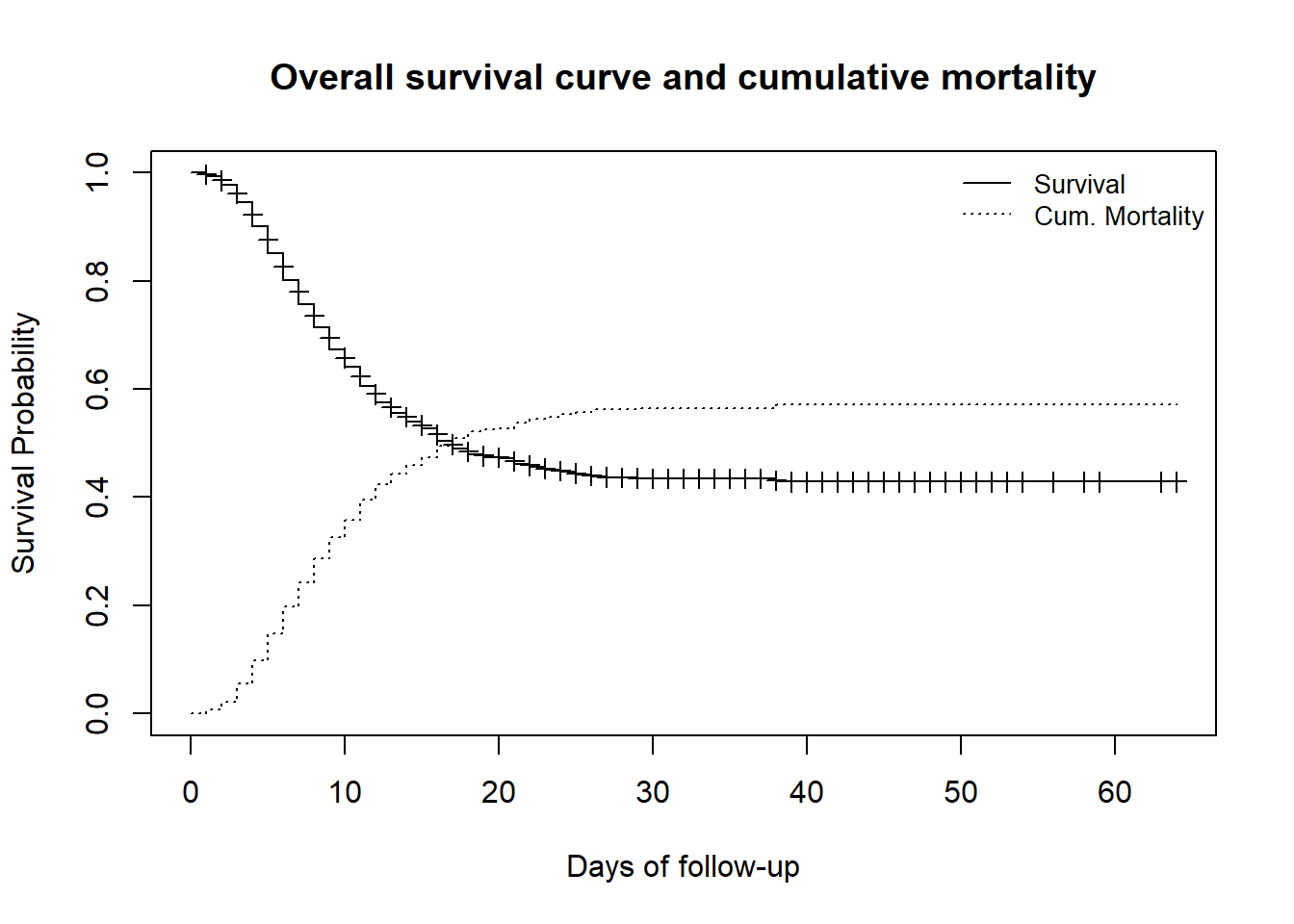
Поскольку интересующее событие - это “смерть”, построение кривой, описывающей дополнения долей выживаемости, приведет к построению кумулятивных долей смертности. Это можно сделать с помощью lines(), которая добавит информацию на существующий график.
# оригинальный график
plot(
linelistsurv_fit,
xlab = "Days of follow-up",
ylab = "Survival Probability",
mark.time = TRUE, # отмечаем события на кривой: "+" печатается при каждом событии
conf.int = FALSE, # не наносить на график доверительный интервал
main = "Overall survival curve and cumulative mortality"
)
# рисуем дополнительную кривую к предыдущему графику
lines(
linelistsurv_fit,
lty = 3, # используем другой тип линии для ясности
fun = "event", # рисуем кумулятивные события вместо выживания
mark.time = FALSE,
conf.int = FALSE
)
# добавляем легенду на график
legend(
"topright", # положение легенды
legend = c("Survival", "Cum. Mortality"), # текст легенды
lty = c(1, 3), # типы линий для использования в легенде
cex = .85, # параметры, которые задают размер текст легенды
bty = "n" # тип без рамки для легенды
)27.4 Сравнение кривых выживаемости
Для сравнения выживаемости в различных группах наблюдаемых нами участников или пациентов может потребоваться сначала рассмотреть соответствующие кривые выживаемости, а затем провести тесты для оценки различий между независимыми группами. Это сравнение может касаться групп по полу, возрасту, лечению, сопутствующим заболеваниям…
Логарифмический ранговый критерий
Логарифмический ранговый критерий является популярным тестом, который сравнивает весь опыт выживания между двумя или более независимыми группами, его можно рассматривать как тест того, являются ли кривые выживаемости идентичными (накладывающимися) или нет (нулевая гипотеза - нет разницы в выживаемости между группами). Функция survdiff() из пакета survival позволяет применить логарифмический ранговый критерий, когда мы уточняем rho = 0 (значение по умолчанию). Результаты теста дают статистику хи-квадрата и p-значение, поскольку статистика логарифмического ранга примерно распределена как статистика теста хи-квадрат.
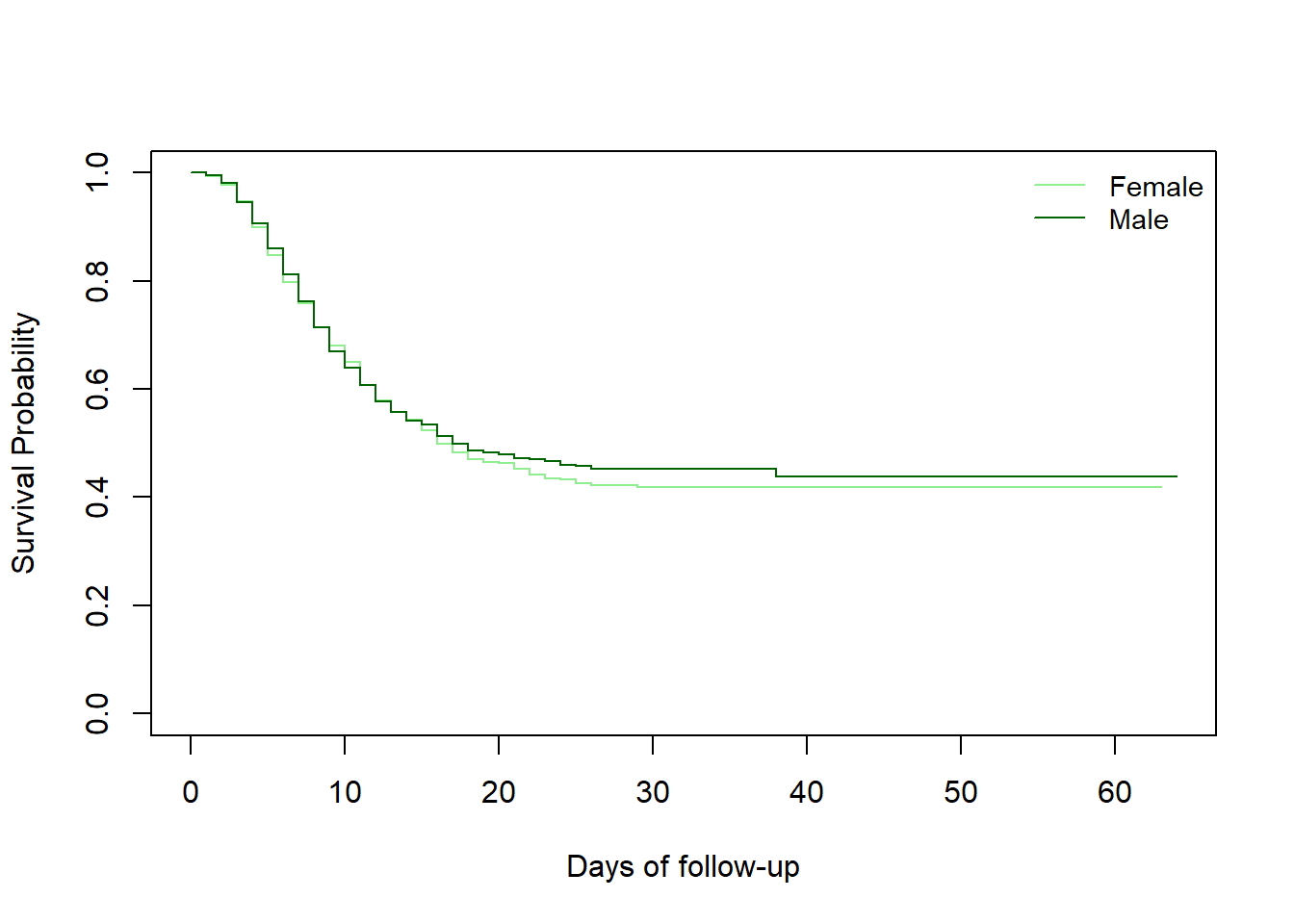
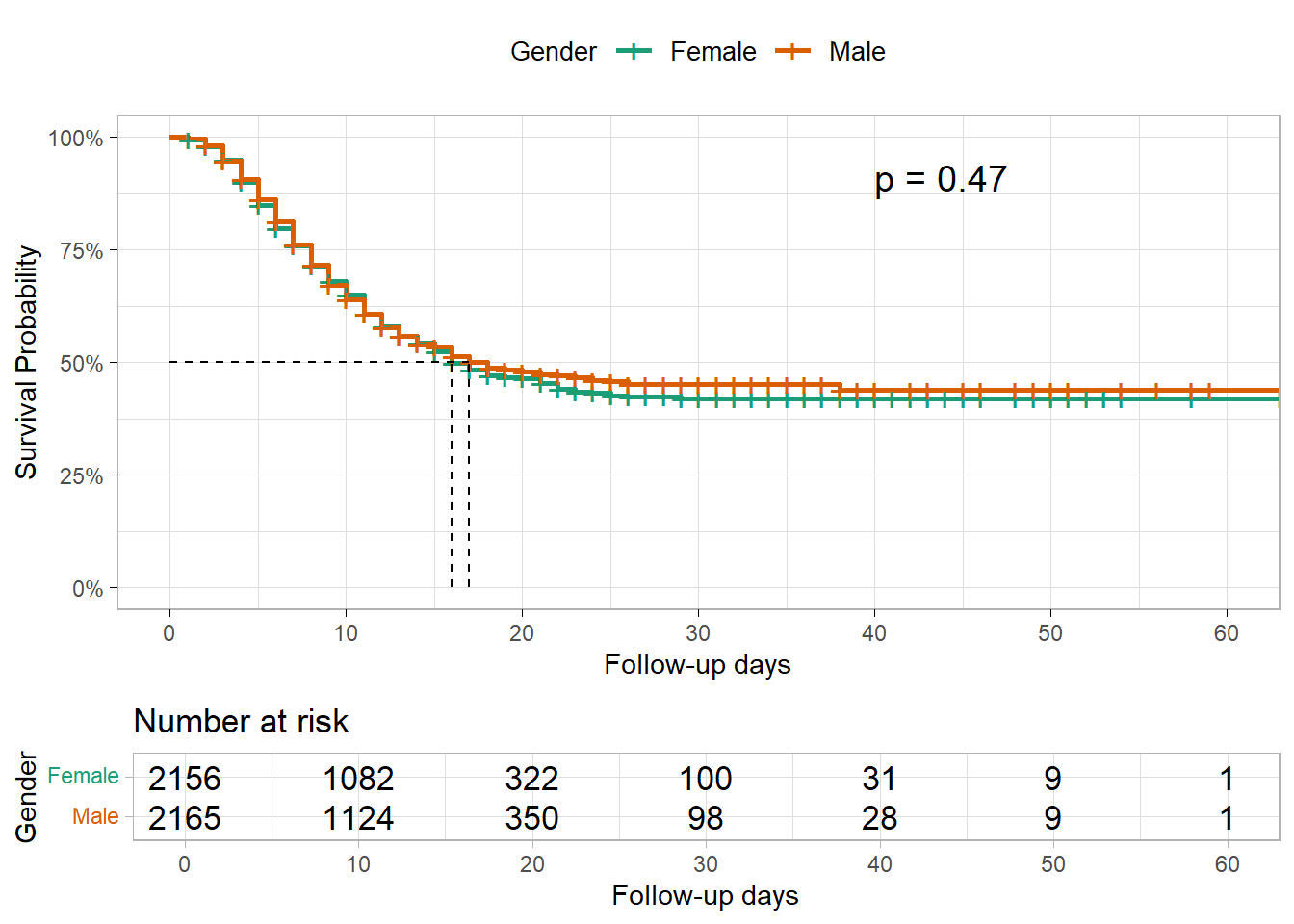
Сначала мы попытаемся сравнить кривые выживаемости по гендерным группам. Для этого сначала попробуем визуализировать их (проверить, накладываются ли две кривые выживания друг на друга). Необходимо создать новый объект survfit с немного другой формулой. Затем будет создан объект survdiff.
Задав ~ gender в качестве правой стороны формулы, мы строим уже не общую выживаемость, а выживаемость по полу.
# создаем новый объект survfit на основе пола
linelistsurv_fit_sex <- survfit(Surv(futime, event) ~ gender, data = linelist_surv)Теперь мы можем построить график кривых выживания по полу. Посмотрите на порядок уровней страты в столбце пол до определения цветов и легенды.
# задаем цвета
col_sex <- c("lightgreen", "darkgreen")
# создаем график
plot(
linelistsurv_fit_sex,
col = col_sex,
xlab = "Days of follow-up",
ylab = "Survival Probability")
# добавляем легенду
legend(
"topright",
legend = c("Female","Male"),
col = col_sex,
lty = 1,
cex = .9,
bty = "n")Теперь мы можем рассчитать тест разницы между кривыми выживаемости, используя survdiff()
#рассчитываем тест разницы между кривыми выживаемости
survival::survdiff(
Surv(futime, event) ~ gender,
data = linelist_surv
)Call:
survival::survdiff(formula = Surv(futime, event) ~ gender, data = linelist_surv)
n=4321, 218 observations deleted due to missingness.
N Observed Expected (O-E)^2/E (O-E)^2/V
gender=f 2156 924 909 0.255 0.524
gender=m 2165 929 944 0.245 0.524
Chisq= 0.5 on 1 degrees of freedom, p= 0.5 Мы видим, что кривая выживаемости для женщин и кривая для мужчин накладываются друг на друга, а логарифмический ранговый критерий не дает доказательств различий в выживаемости между женщинами и мужчинами.
Некоторые другие пакеты R позволяют иллюстрировать кривые выживаемости для разных групп и тестировать разницу одновременно. Используя функцию ggsurvplot() из пакета survminer, мы также можем включить на нашу кривую напечатанные таблицы рисков для каждой группы, а также p-значение из логарифмического ранкового критерия.
ВНИМАНИЕ: функции survminer требуют, чтобы вы задавали объект выживаемости и снова задавали данные, используемын для построения объекта выживаемости. Не забудьте это сделать, иначе получите неспецифическое сообщение об ошибке.
survminer::ggsurvplot(
linelistsurv_fit_sex,
data = linelist_surv, # снова задаем данные, используемые для построения linelistsurv_fit_sex
conf.int = FALSE, # не показывать доверительный интервал оценок КМ
surv.scale = "percent", # представляет вероятности на оси y в %
break.time.by = 10, # представляет ось времени с шагом в 10 дней
xlab = "Follow-up days",
ylab = "Survival Probability",
pval = T, # печать p-значения логарифмического рангового критерия
pval.coord = c(40,.91), # печать p-значения на этих координатах графика
risk.table = T, # печать таблицы рисков внизу
legend.title = "Gender", # характеристики легенды
legend.labs = c("Female","Male"),
font.legend = 10,
palette = "Dark2", # уточняем цветовую палитру
surv.median.line = "hv", # рисуем горизонтальные и вертикальные линии по медианному выживанию
ggtheme = theme_light() # упрощаем фон графика
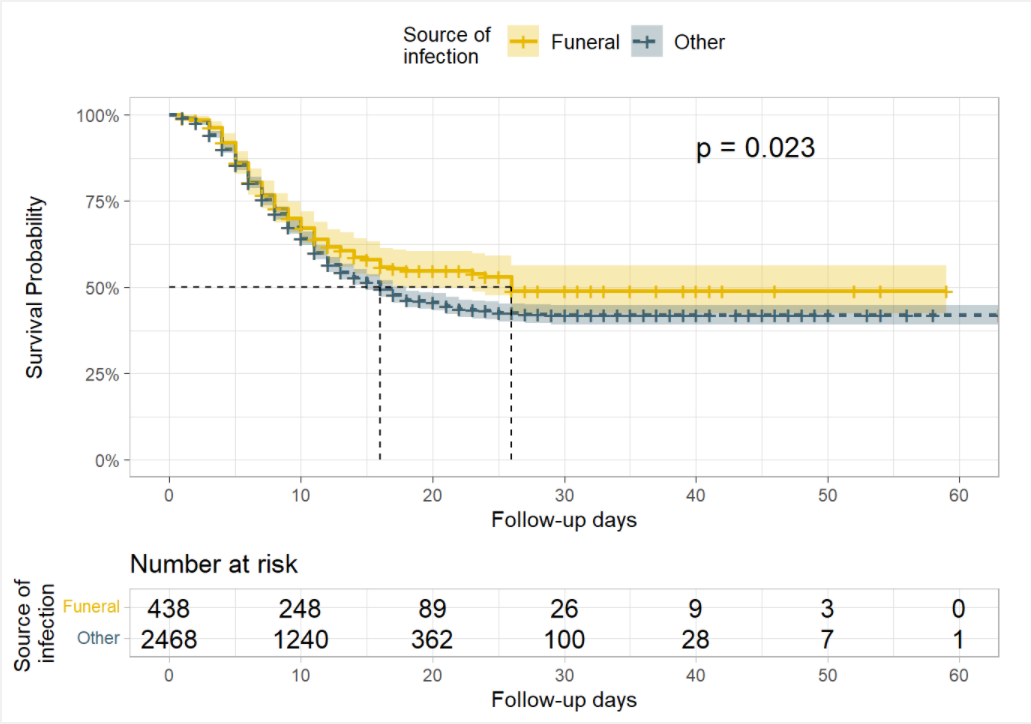
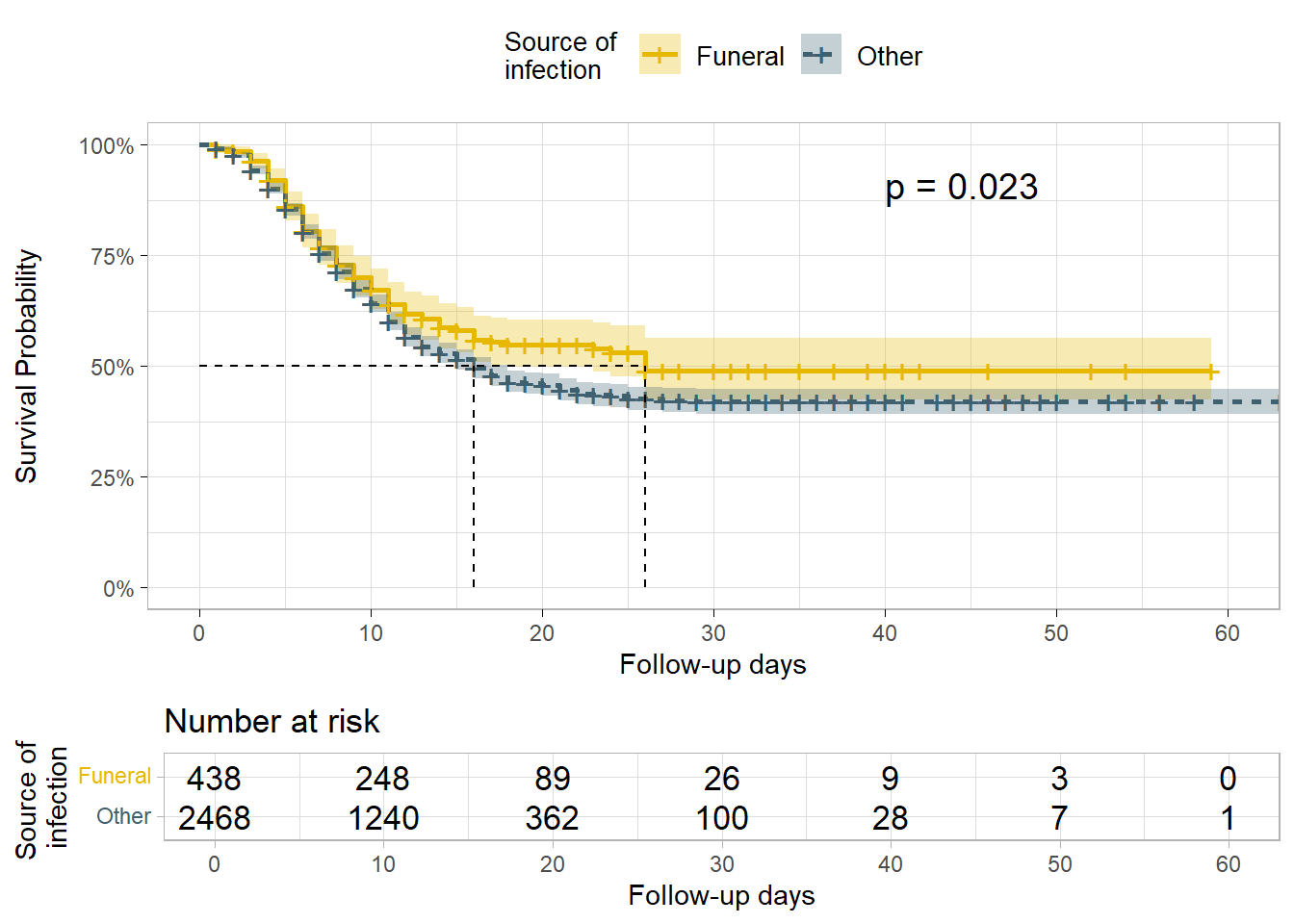
)Мы можем также протестировать различия в выживаемости по источнику инфекции (источнику загрязнения).
В данном случае логарифмический ранговый критерий дает достаточно доказательств различий вероятности выживания при alpha= 0.005. Вероятности выживания для пациентов, которые были заражены на похоронах, выше, чем вероятности выживания пациентов, которые заразились в других местах, что указывает на преимущество в выживаемости.
linelistsurv_fit_source <- survfit(
Surv(futime, event) ~ source,
data = linelist_surv
)
# plot
ggsurvplot(
linelistsurv_fit_source,
data = linelist_surv,
size = 1, linetype = "strata", # line types
conf.int = T,
surv.scale = "percent",
break.time.by = 10,
xlab = "Follow-up days",
ylab= "Survival Probability",
pval = T,
pval.coord = c(40,.91),
risk.table = T,
legend.title = "Source of \ninfection",
legend.labs = c("Funeral", "Other"),
font.legend = 10,
palette = c("#E7B800","#3E606F"),
surv.median.line = "hv",
ggtheme = theme_light()
)Warning in geom_segment(aes(x = 0, y = max(y2), xend = max(x1), yend = max(y2)), : All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?
All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?
All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?
All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?27.5 Регрессия Кокса
Регрессия пропорциональных рисков Кокса является одним из наиболее популярных методов регрессии для анализа выживаемости. Можно использовать и другие модели, поскольку модель Кокса требует важных допущений, которые необходимо верифицировать для правильного использования, например, допущение о пропорциональных моментных рисках: см. ссылки.
В регрессии пропорциональных рисков Кокса мерой эффекта является коэффициент моментных рисков (HR), который представляет собой риск происшествия (или риск смерти в нашем примере), учитывая, что участник дожил до определенного времени. Как правило, нас интересует сравнение независимых групп по отношению к их моментным рискам, и мы используем отношение моментных рисков, корторое похоже на отношение шансов в плане создания множественной логистической регрессии. Функция cox.ph() из пакета survival используется для построения этой модели. Для тестирования допущения о пропорциональных моментных рисках при построении регрессии Кокса можно использовать функцию cox.zph() из пакета survival.
ПРИМЕЧАНИЕ: Вероятность должна лежать в диапазоне от 0 до 1. Однако моментный риск представляет собой ожидаемое число событий за единицу времени.
- Если коэффициент моментного риска для предиктора близок к 1, то этот предиктор не влияет на выживаемость,
- Если коэффициент моментного риска менее 1, тогда предиктор является защитным (т.е. связан с улучшением выживаемости),
- А если коэффициент моментного риска больше 1, тогда предиктор связан с повышенным риском (или снижением выживаемости).
Построение модели Кокса
Сначала мы можем построить модель для оценки влияния возраста и пола на выживаемость. Просто напечатав модель, мы получим информацию о:
- оценочных коэффициентах регрессии
coef, которые количественно выражают связь между предикторами и исходом, - их экспоненциальной функции (для интерпретируемости,
exp(coef)), которая создает коэффициент моментного риска, - их стандартной ошибке
se(coef), - z-балле: на сколько стандартных ошибок расчетный коэффициент удален от 0,
- и p-значение: вероятность того, что расчетный коэффициент мог бы быть 0.
Функция summary(), примененная к объекту модели Кокса даст вам больше информации, например, доверительный интервал оцененного коэффициента моментного риска и разные баллы тестов.
Эффект первой ковариаты gender (пол) представлен в первой строке. genderm (мужской) напечатан, что указывает на то, что первый уровень страты (“f”), т.е. группа женского пола, является референтной группой для пола. Таким образом, интерпретация тестового параметра относится к мужчинам по сравнению с женщинами. p-значение указывает на отсутствие достаточных доказательств влияния пола на ожидаемый моментный риск или связи между полом и смертностью от всех причин.
То же отсутствие доказательств отмечается в отношении возрастной группы.
#строим модель Кокса
linelistsurv_cox_sexage <- survival::coxph(
Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv
)
#печать построенной модели
linelistsurv_cox_sexageCall:
survival::coxph(formula = Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv)
coef exp(coef) se(coef) z p
genderm -0.03149 0.96900 0.04767 -0.661 0.509
age_cat_small5-19 0.09400 1.09856 0.06454 1.456 0.145
age_cat_small20+ 0.05032 1.05161 0.06953 0.724 0.469
Likelihood ratio test=2.8 on 3 df, p=0.4243
n= 4321, number of events= 1853
(218 observations deleted due to missingness)#сводная информация по модели
summary(linelistsurv_cox_sexage)Call:
survival::coxph(formula = Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv)
n= 4321, number of events= 1853
(218 observations deleted due to missingness)
coef exp(coef) se(coef) z Pr(>|z|)
genderm -0.03149 0.96900 0.04767 -0.661 0.509
age_cat_small5-19 0.09400 1.09856 0.06454 1.456 0.145
age_cat_small20+ 0.05032 1.05161 0.06953 0.724 0.469
exp(coef) exp(-coef) lower .95 upper .95
genderm 0.969 1.0320 0.8826 1.064
age_cat_small5-19 1.099 0.9103 0.9680 1.247
age_cat_small20+ 1.052 0.9509 0.9176 1.205
Concordance= 0.514 (se = 0.007 )
Likelihood ratio test= 2.8 on 3 df, p=0.4
Wald test = 2.78 on 3 df, p=0.4
Score (logrank) test = 2.78 on 3 df, p=0.4Было интересно выполнить модель и посмотреть на результаты, но первый взгляд на то, соблюдается ли предположение о пропорциональных моментных рисках, мог бы помочь сэкономить время.
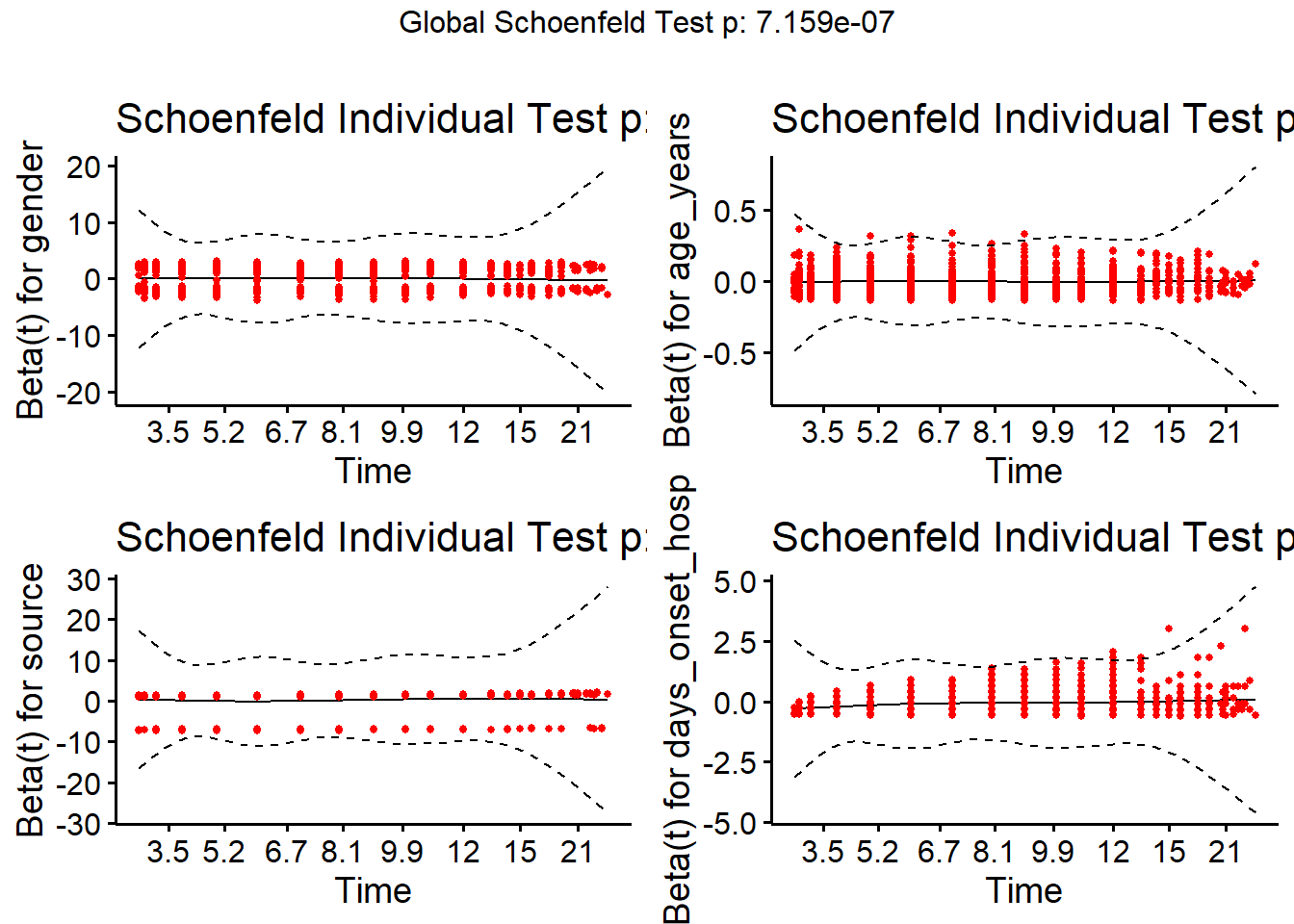
test_ph_sexage <- survival::cox.zph(linelistsurv_cox_sexage)
test_ph_sexage chisq df p
gender 0.454 1 0.50
age_cat_small 0.838 2 0.66
GLOBAL 1.399 3 0.71ПРИМЕЧАНИЕ: Второй аргумент под названием method (метод) может быть задан при расчете модели Кокса, которая определяет как работать с одинаковыми результатами. По умолчанию используется “efron”, а другие опции включают “breslow” и “exact”.
В другой модели мы добавляем дополнительные факторы риска, такие как источник инфекции и количество дней между датой возникновения заболевания и госпитализацией. На этот раз мы сначала проверяем предположение о пропорциональном моментном риске, прежде чем двигаться дальше.
В этой модели мы включили непрерывный предиктор (days_onset_hosp). В этом случае мы интерпретируем оценки параметров как увеличение ожидаемого логарифма относительного моментного риска на каждую единицу увеличения предиктора при постоянстве других предикторов. Сначала мы проверим предположение о пропорциональных моментных рисках.
#строим модель
linelistsurv_cox <- coxph(
Surv(futime, event) ~ gender + age_years+ source + days_onset_hosp,
data = linelist_surv
)
#тестируем модель пропорциональных рисков
linelistsurv_ph_test <- cox.zph(linelistsurv_cox)
linelistsurv_ph_test chisq df p
gender 0.45062 1 0.50
age_years 0.00199 1 0.96
source 1.79622 1 0.18
days_onset_hosp 31.66167 1 1.8e-08
GLOBAL 34.08502 4 7.2e-07Графическая верификация этого допущения может выполнена с помощью функции ggcoxzph() из пакета survminer.
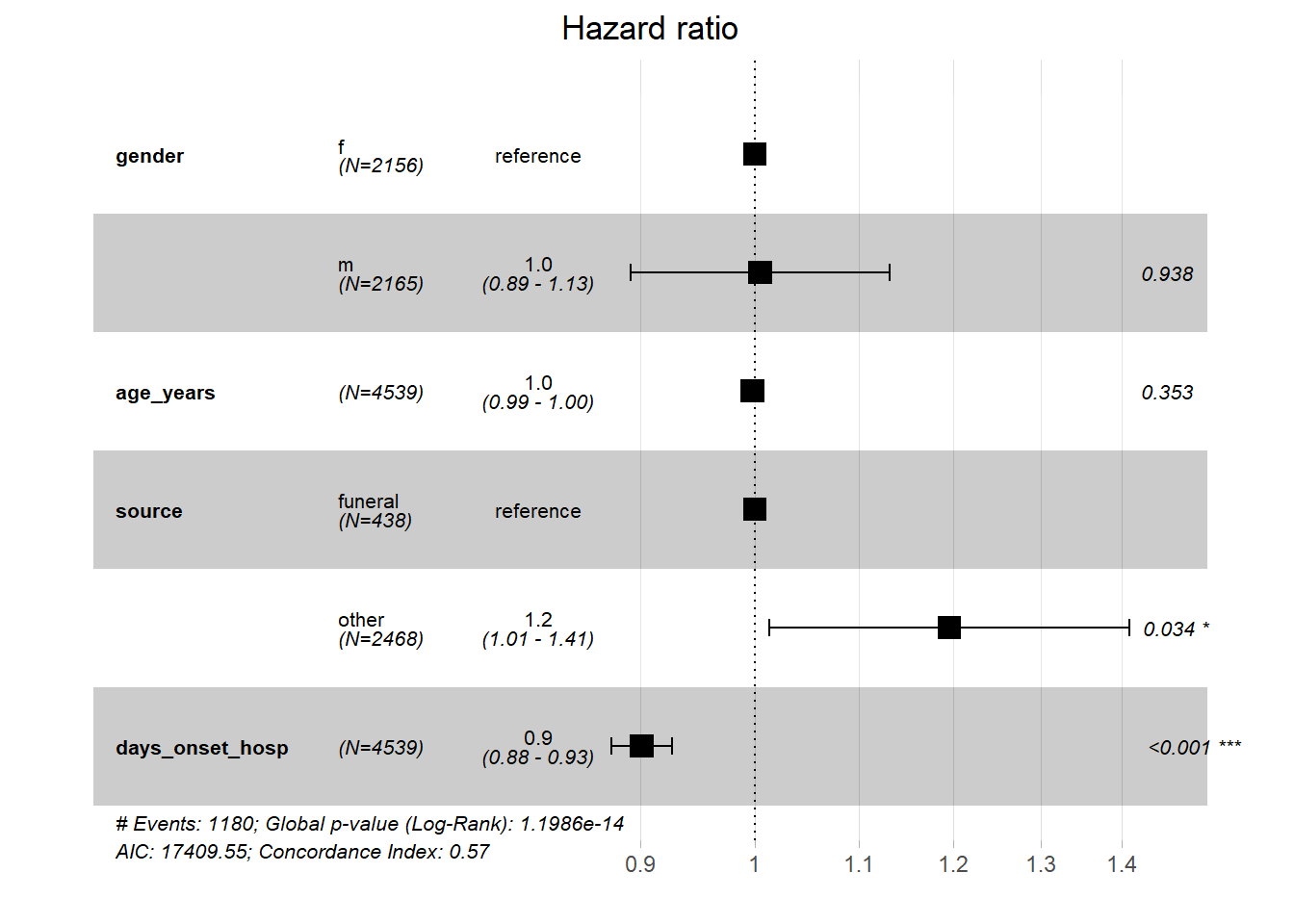
Результаты моделирования свидетельствуют о наличии отрицательной связи между продолжительностью госпитализации и смертностью от всех причин. Ожидаемый риск в 0,9 раза ниже у человека, поступившего на один день позже, чем у другого, при постоянном учете пола. Или, в более простом варианте, увеличение продолжительности госпитализации на одну единицу связано с уменьшением риска смерти на 10,7% (coef *100).
Полученные результаты свидетельствуют также о положительной связи между источником инфекции и смертностью от всех причин. То есть риск смерти (в 1,21 раза) повышается у пациентов, у которых источником инфекции были не похороны.
#печатаем сводную информацию по модели
summary(linelistsurv_cox)Call:
coxph(formula = Surv(futime, event) ~ gender + age_years + source +
days_onset_hosp, data = linelist_surv)
n= 2772, number of events= 1180
(1767 observations deleted due to missingness)
coef exp(coef) se(coef) z Pr(>|z|)
genderm 0.004710 1.004721 0.060827 0.077 0.9383
age_years -0.002249 0.997753 0.002421 -0.929 0.3528
sourceother 0.178393 1.195295 0.084291 2.116 0.0343 *
days_onset_hosp -0.104063 0.901169 0.014245 -7.305 2.77e-13 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
exp(coef) exp(-coef) lower .95 upper .95
genderm 1.0047 0.9953 0.8918 1.1319
age_years 0.9978 1.0023 0.9930 1.0025
sourceother 1.1953 0.8366 1.0133 1.4100
days_onset_hosp 0.9012 1.1097 0.8764 0.9267
Concordance= 0.566 (se = 0.009 )
Likelihood ratio test= 71.31 on 4 df, p=1e-14
Wald test = 59.22 on 4 df, p=4e-12
Score (logrank) test = 59.54 on 4 df, p=4e-12Мы можем верифицировать это отношение с помощью таблицы:
linelist_case_data %>%
tabyl(days_onset_hosp, outcome) %>%
adorn_percentages() %>%
adorn_pct_formatting() days_onset_hosp Death Recover NA_
0 44.3% 31.4% 24.3%
1 46.6% 32.2% 21.2%
2 43.0% 32.8% 24.2%
3 45.0% 32.3% 22.7%
4 41.5% 38.3% 20.2%
5 40.0% 36.2% 23.8%
6 32.2% 48.7% 19.1%
7 31.8% 38.6% 29.5%
8 29.8% 38.6% 31.6%
9 30.3% 51.5% 18.2%
10 16.7% 58.3% 25.0%
11 36.4% 45.5% 18.2%
12 18.8% 62.5% 18.8%
13 10.0% 60.0% 30.0%
14 10.0% 50.0% 40.0%
15 28.6% 42.9% 28.6%
16 20.0% 80.0% 0.0%
17 0.0% 100.0% 0.0%
18 0.0% 100.0% 0.0%
22 0.0% 100.0% 0.0%
NA 52.7% 31.2% 16.0%Необходимо рассмотреть и исследовать, почему такая связь существует в данных. Одно из возможных объяснений может заключаться в том, что пациенты, прожившие достаточно долго, чтобы быть госпитализированными позже, изначально имели менее тяжелое заболевание. Другое, возможно, более вероятное объяснение состоит в том, что, поскольку мы использовали смоделированный фальшивый набор данных, эта закономерность не отражает реальности!
Форест-графики
Затем мы можем визуализировать результаты модели Кокса, используя практичные форест-графики с помощью функции ggforest() из пакета survminer.
27.6 Ковариаты, зависящие от времени, в моделях выживания
Некоторые из нижеследующих разделов были адаптированы с разрешения автора из превосходной книги Введение в анализ выживаемости в R, написанной Доктором Эмили Забор
В предыдущем разделе мы рассмотрели использование регрессии Кокса для изучения ассоциаций между интересующими нас ковариатами и результатами выживания. Однако эти анализы основаны на том, что ковариата измеряется на исходном уровне, то есть до начала периода отслеживания события.
Что произойдет, если вас интересует ковариата, которая измеряется после того, как началось время отслеживания? Или если у вас ковариата, которая может меняться со временем?
Например, вы работаете с клиническими данными, в которых повторяются измерения показателей больничных лабораторий, которые могут меняться с течением времени. Это является примером Ковариаты, зависящей от времени. Для решения этой задачи необходима специальная настройка, но, к счастью, модель Кокса очень гибкая, и этот тип данных также может быть смоделирован с помощью инструментов из пакета survival.
Настройка ковариаты, зависящей от времени
Анализ ковариат, зависящих от времени, в R требует создания специального набора данных. Если интересно, см. более подробную статью об этом от автора пакета survival Использование зависящих от времени ковариат и зависящих от времени коэффициентов в модели Кокса.
Для этого используем новый набор данных из пакета SemiCompRisks под названием BMT, который включает данные о 137 пациентах после пересадки костного мозга. Переменные, на которых мы сфокусируемся:
-
T1- время (в днях) до смерти или последнего отслеживания
-
delta1- индикатор смерти; 1-умер, 0-жив
-
TA- время (в днях) до острой реакции трансплантат против хозяина
-
deltaA- индикатор острой реакции трансплантат против хозяина;- 1 - развилась острая реакция трансплантат против хозяина
- 0 - никогда не было острой реакции трансплантат против хозяина
- 1 - развилась острая реакция трансплантат против хозяина
Мы загрузим этот набор данных из пакета survival, используя команду из базового R data(), которую можно использовать для загрузки данных, которые уже включены в пакет R, который загружается. Датафрейм BMT появится в рабочей среде R (environment).
data(BMT, package = "SemiCompRisks")Добавляем уникальный идентификатор пациента
В данных BMT нет столбца с уникальным ID, который необходим для создания такого типа набора данных, который нам нужен. Поэтому мы используем функцию rowid_to_column() из пакета tidyverse tibble для создания нового столбца с id под именем my_id (добавляет столбец в начале датафрейма с последовательными идентификаторами строк, начиная с 1). Назовем датафрейм bmt.
bmt <- rowid_to_column(BMT, "my_id")Набор данных теперь выглядит так:
Расширяем строки по пациентам
Далее используем функцию tmerge() с функциями-помощниками event() и tdc(), чтобы создать реструктурированный набор данных. Наша цель - реструктурировать набор данных, чтобы создать отдельную строку для каждого пациента в каждый временной интервал, когда у них отличаются значения deltaA. В этом случае у каждого пациента может быть не более двух строк в зависимости от того, развилась ли у него острая реакция “трансплантат против хозяина” за период сбора данных. Назовем наш новый показатель развития острой реакции трансплантат против хозяина agvhd.
-
tmerge()создает длинный набор данных с несколькими временными интервалами для разных значений ковариат для каждого пациента -
event()создает новый индикатор события для новых созданных временных интервалов -
tdc()создает столбец зависящей от времени ковариаты,agvhd, по новым созданным временным интервалам
td_dat <-
tmerge(
data1 = bmt %>% select(my_id, T1, delta1),
data2 = bmt %>% select(my_id, T1, delta1, TA, deltaA),
id = my_id,
death = event(T1, delta1),
agvhd = tdc(TA)
)Чтобы увидеть, что это даст, давайте рассмотрим данные по первым 5 пациентам.
Интересующие переменные в оригинальном наборе данных выглядели так:
bmt %>%
select(my_id, T1, delta1, TA, deltaA) %>%
filter(my_id %in% seq(1, 5)) my_id T1 delta1 TA deltaA
1 1 2081 0 67 1
2 2 1602 0 1602 0
3 3 1496 0 1496 0
4 4 1462 0 70 1
5 5 1433 0 1433 0Новый набор данных для тех же пациентов выглядит так:
td_dat %>%
filter(my_id %in% seq(1, 5)) my_id T1 delta1 tstart tstop death agvhd
1 1 2081 0 0 67 0 0
2 1 2081 0 67 2081 0 1
3 2 1602 0 0 1602 0 0
4 3 1496 0 0 1496 0 0
5 4 1462 0 0 70 0 0
6 4 1462 0 70 1462 0 1
7 5 1433 0 0 1433 0 0Теперь у некоторых наших пациентов есть две строки в наборе данных, соответствующие интервалам, в которых они имеют разные значения нашей новой переменной, agvhd. Например, у Пациента 1 сейчас две строки со значением agvhd ноль с момента 0 до времени 67, и значение 1 с времени 67 до времени 2081.
Регрессия Кокса с ковариатами, зависящими от времени
Теперь, когда мы переформатировали наши данные и добавили новую зависящую от времени переменную aghvd, давайте построим простую регрессию Кокса с одной переменной. Мы можем использовать ту же функцию coxph() как ранее, нам только нужно изменить нашу функцию Surv(), чтобы уточнить время начала и окончания каждого интервала, используя аргументы time1 = и time2 =.
bmt_td_model = coxph(
Surv(time = tstart, time2 = tstop, event = death) ~ agvhd,
data = td_dat
)
summary(bmt_td_model)Call:
coxph(formula = Surv(time = tstart, time2 = tstop, event = death) ~
agvhd, data = td_dat)
n= 163, number of events= 80
coef exp(coef) se(coef) z Pr(>|z|)
agvhd 0.3351 1.3980 0.2815 1.19 0.234
exp(coef) exp(-coef) lower .95 upper .95
agvhd 1.398 0.7153 0.8052 2.427
Concordance= 0.535 (se = 0.024 )
Likelihood ratio test= 1.33 on 1 df, p=0.2
Wald test = 1.42 on 1 df, p=0.2
Score (logrank) test = 1.43 on 1 df, p=0.2Опять же, мы визуализируем результаты нашей модели Кокса, используя функцию ggforest() из пакета survminer.:
Как видно из форест-графика, доверительного интервала и значения p, в контексте нашей простой модели не наблюдается сильной связи между смертью и острой реакцией трансплантат против хозяина.
27.7 Ресурсы
Анализ выживаемости Часть I: Основные концепции и первый анализ
Анализ выживаемости в исследованиях по инфекционным заболеваниям: Описание событий во времени
Глава по продвинутым моделям выживания от Принстона
Использование ковариат, зависящих от времени, в модели Кокса