3 R - les bases
Bienvenue !
Cette page passe en revue les éléments essentiels de R. Elle n’a pas pour but d’être un tutoriel complet, mais elle fournit les bases et peut être utile pour rafraîchir votre mémoire. La section Ressources pour l’apprentissage renvoie à des didacticiels plus complets.
Certaines parties de cette page ont été adaptées avec l’autorisation du projet R4Epis.
Voir la page sur la transition a R pour des conseils sur le passage de STATA, SAS ou Excel à R.
3.1 Pourquoi utiliser R ?
Comme indiqué sur le site Web du projet R, R est un langage de programmation et un environnement pour le calcul statistique et les graphiques. Il est très polyvalent, extensible et axé sur la communauté.
Coût
L’utilisation de R est gratuite ! Il existe une forte éthique dans la communauté du matériel gratuit et open-source.
Reproductibilité
La gestion et l’analyse de vos données par le biais d’un langage de programmation (par rapport à Excel ou à un autre outil essentiellement manuel) améliore la reproductibilité, facilite la détection des erreurs et allège votre charge de travail.
Communauté
La communauté des utilisateurs de R est énorme et collaborative. De nouveaux paquets et outils destinés à résoudre des problèmes concrets sont développés quotidiennement et approuvés par la communauté des utilisateurs. À titre d’exemple, R-Ladies est une organisation mondiale dont la mission est de promouvoir la diversité des genres dans la communauté R, et c’est l’une des plus grandes organisations d’utilisateurs de R. Elle a probablement un chapitre près de chez vous !
3.2 Termes clés
RStudio - RStudio est une interface utilisateur graphique (GUI) qui facilite l’utilisation de R. Pour en savoir plus, consultez la section RStudio.
Objets - Tout ce que vous stockez dans R - les jeu de données, les variables, une liste de noms de villages, un population total d’habitants, et même les résultats tels que les graphiques - sont des objets auxquels on attribue un nom et qui peuvent être référencés dans des commandes ultérieures. Pour en savoir plus, consultez la section Objets.
Fonctions - Une fonction est une opération de code qui accepte des entrées et renvoie une sortie transformée. Pour en savoir plus, consultez la section Fonctions.
Paquets - Un paquet R est un ensemble de fonctions partageables. Pour en savoir plus, consultez la section Packages.
Scripts - Un script est le fichier document qui contient vos commandes. Pour en savoir plus, consultez la section Scripts
3.3 Ressources pour l’apprentissage
Ressources au sein de RStudio
Documentation d’aide
Recherchez dans l’onglet “Aide” de RStudio la documentation sur les paquets R et les fonctions spécifiques. Cet onglet se trouve dans le volet qui contient également les fichiers, les graphiques et les paquets (généralement dans le volet inférieur à droit). Comme raccourci, vous pouvez également taper le nom d’un paquet ou d’une fonction dans la console R après un point d’interrogation pour ouvrir la page d’aide correspondante. N’incluez pas les parenthèses.
Par exemple : ?filter ou ?diagrammeR.
Tutoriels interactifs
Il existe plusieurs façons d’apprendre R de manière interactive dans RStudio.
RStudio lui-même offre un volet Tutoriel qui est alimenté par le paquet R learnr. Il suffit d’installer ce paquet et d’ouvrir un tutoriel via le nouvel onglet “Tutorial” dans le volet supérieur droit de RStudio (qui contient également les onglets Environnement et Historique).
Le paquet R swirl propose des cours interactifs dans la console R. Installez et chargez ce paquet, puis lancez la commande swirl() (parenthèses vides) dans la console R. Vous verrez apparaître des invites dans la console. Répondez en tapant dans la console. Elle vous guidera à travers un cours de votre choix.
Fiches d’aide-mémoire
Il existe de nombreuses fiches d’aide-mémoire au format PDF disponibles sur le site Web de RStudio, par exemple :
- Facteurs avec le paquet forcats
- Dates et heures avec le paquet lubridate
- Chaînes de caractères avec le paquet stringr
- Opérations itératives avec le paquet purrr
- Importation de données
- Aide-mémoire pour la transformation des données avec le paquet dplyr
- R Markdown (pour créer des documents comme PDF, Word, Powerpoint…)
- Shiny (pour créer des applications Web interactives)
- Visualisation de données avec le paquet ggplot2
- Cartographie (SIG)
- Paquet leaflet (cartes interactives)
- Python avec R (paquet reticulate)
Il existe également une ressource R en ligne spécialement destinée aux utilisateurs d’Excel.
R possède une communauté Twitter dynamique où vous pouvez apprendre des astuces, des raccourcis et des nouvelles - suivez ces comptes :
- Suivez-nous ! @epiRhandbook
- R Function A Day @rfuntionaday est une ressource incroyable
- R pour la science des données @rstats4ds
- RStudio @RStudio
- Conseils sur RStudio@rstudiotips
- R-Bloggers @Rbloggers
- R-ladies @RLadiesGlobal
- Hadley Wickham @hadleywickham
Aussi :
#epitwitter et #rstats
Ressources gratuites en ligne
Un texte définitif est le livre R for Data Science de Garrett Grolemund et Hadley Wickham.
Le site Web du projet R4Epis vise à “développer des outils standardisés de nettoyage, d’analyse et de rapport des données pour couvrir les types courants d’épidémies et d’enquêtes auprès de la population qui seraient menées dans le cadre d’une réponse d’urgence de MSF”. Vous y trouverez des supports de formation aux bases de R, des modèles de rapports RMarkdown sur les épidémies et les enquêtes, ainsi que des tutoriels pour vous aider à les configurer.
Langues autres que l’anglais
3.4 Installation
R et RStudio
Comment installer R
Visitez ce site Web https://www.r-project.org/ et téléchargez la dernière version de R adaptée à votre ordinateur.
Comment installer RStudio
Visitez ce site Web https://rstudio.com/products/rstudio/download/ et téléchargez la dernière version de bureau gratuite de RStudio adaptée à votre ordinateur.
Autorisations requises
Notez que vous devez installer R et RStudio sur un lecteur sur lequel vous avez des droits de lecture et d’écriture. Sinon, votre capacité à installer des paquets R (ce qui arrive fréquemment) sera affectée. Si vous rencontrez des problèmes, essayez d’ouvrir RStudio en faisant un clic droit sur l’icône et en sélectionnant “Exécuter en tant qu’administrateur”. Vous trouverez d’autres conseils sur la page R sur les lecteurs réseau.
Comment mettre à jour R et RStudio
Votre version de R est imprimée dans la Console R au démarrage. Vous pouvez également exécuter sessionInfo().
Pour mettre à jour R, allez sur le site web mentionné ci-dessus et réinstallez R. Alternativement, vous pouvez utiliser le paquet installr (sous Windows) en exécutant installr::updateR(). Cela ouvrira des boîtes de dialogue pour vous aider à télécharger la dernière version de R et à mettre à jour vos paquets vers la nouvelle version de R. Plus de détails peuvent être trouvés dans la documentation de installr.
Sachez que l’ancienne version de R existera toujours sur votre ordinateur. Vous pouvez temporairement exécuter une ancienne version (ancienne “installation”) de R en cliquant sur “Outils” -> “Options globales” dans RStudio et en choisissant une version de R. Cela peut être utile si vous voulez utiliser un paquet qui n’a pas été mis à jour pour fonctionner sur la version la plus récente de R.
Pour mettre à jour RStudio, vous pouvez aller sur le site Web ci-dessus et retélécharger RStudio. Une autre option consiste à cliquer sur “Aide” -> “Vérifier les mises à jour” dans RStudio, mais cela peut ne pas montrer les toutes dernières mises à jour.
Pour savoir quelles versions de R, RStudio ou des paquets ont été utilisées lors de la réalisation de ce manuel, consultez la page sur Notes techniques et choix éditoriaux.
Autres logiciels que vous pourriez avoir besoin d’installer
- TinyTeX (pour la compilation d’un document RMarkdown au format PDF)
- Pandoc (pour compiler des documents RMarkdown)
- RTools (pour construire des paquets pour R)
- phantomjs (pour enregistrer des images fixes de réseaux animés, tels que des chaînes de transmission)
TinyTex
TinyTex est une distribution LaTeX personnalisée, utile lorsqu’on essaie de produire des PDF à partir de R. Voir https://yihui.org/tinytex/ pour plus d’informations.
Pour installer TinyTex à partir de R :
install.packages('tinytex')
tinytex::install_tinytex()
# pour désinstaller TinyTeX, lancez tinytex::uninstall_tinytex()Pandoc
Pandoc est un convertisseur de document, un logiciel séparé de R. Il est fourni avec RStudio et ne devrait pas avoir besoin d’être téléchargé. Il aide le processus de conversion de documents Rmarkdown à des formats comme .pdf et ajoute des fonctionnalités complexes.
RTools
RTools est une collection de logiciels permettant de construire des paquets pour R.
Installer à partir de ce site web : https://cran.r-project.org/bin/windows/Rtools/
phantomjs
Cet outil est souvent utilisé pour faire des “captures d’écran” despages web. Par exemple, lorsque vous faites une chaîne de transmission avec le paquet epicontacts, un fichier HTML interactif et dynamique est produit. Si vous voulez une image statique, il peut être utile d’utiliser le paquet webshot pour automatiser ce processus. Cela nécessite le programme externe “phantomjs”. Vous pouvez installer phantomjs via le paquet webshot avec la commande webshot::install_phantomjs().
3.4.1 RStudio
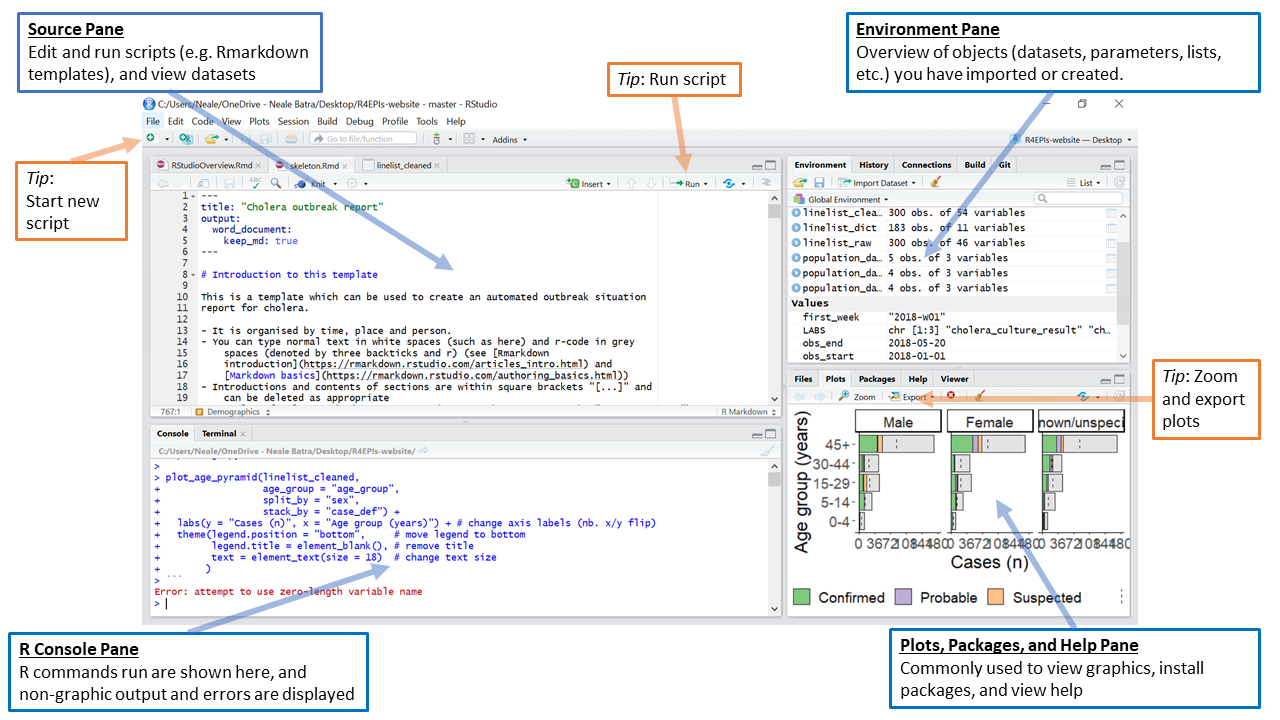
Orientation de RStudio
D’abord, ouvrez RStudio. Comme leurs icônes peuvent être très similaires, assurez-vous que vous ouvrez bien RStudio et non pas R.
Pour que RStudio fonctionne, vous devez également avoir R installé sur l’ordinateur (voir ci-dessus pour les instructions d’installation).
RStudio est une interface (GUI) pour une utilisation plus facile de R. Vous pouvez considérer R comme le moteur d’un véhicule, qui effectue le travail crucial, et RStudio comme le corps du véhicule (avec les sièges, les accessoires, etc.) qui vous aide à utiliser le moteur pour avancer ! Vous pouvez consulter la fiche technique complète de l’interface utilisateur de RStudio (PDF) ici
Par défaut, RStudio affiche quatre volets rectangulaires.
TIP: Si votre RStudio n’affiche qu’un seul volet gauche, c’est parce que vous n’avez pas encore de scripts ouverts.
Le volet source
Ce volet, par défaut en haut à gauche, est un espace pour éditer, exécuter et enregistrer vos scripts. Les scripts contiennent les commandes que vous souhaitez exécuter. Ce volet peut également afficher des ensembles de données (cadres de données) pour les visualiser.
Pour les utilisateurs de Stata, ce volet est similaire aux fenêtres Do-file et Data Editor.
Le volet Console R
La console R, qui est par défaut le volet gauche ou inférieur gauche de R Studio, est le siège du “moteur” R. C’est là que les commandes sont réellement exécutées et que les sorties non graphiques et les messages d’erreur/d’avertissement apparaissent. Vous pouvez saisir et exécuter directement des commandes dans la console R, mais sachez que ces commandes ne sont pas enregistrées comme c’est le cas lorsque vous exécutez des commandes à partir d’un script.
Si vous êtes familier avec Stata, la console R ressemble à la fenêtre de commande et à la fenêtre des résultats.
Le volet Environnement
Ce volet, situé par défaut en haut à droite, est le plus souvent utilisé pour afficher de brefs résumés des objets de l’environnement R dans la session en cours. Ces objets peuvent inclure des ensembles de données importés, modifiés ou créés, des paramètres que vous avez définis (par exemple, une semaine épi spécifique pour l’analyse), ou des vecteurs ou des listes que vous avez définis pendant l’analyse (par exemple, les noms des régions). Vous pouvez cliquer sur la flèche à côté du nom d’un cadre de données pour voir ses variables.
Dans Stata, cette fenêtre est très similaire à celle du gestionnaire de variables.
Ce volet contient également l’onglet “Historique” où vous pouvez voir les commandes que vous avez exécutées précédemment. Il comporte également un onglet “Tutoriel” où vous pouvez suivre des tutoriels R interactifs si vous avez installé le paquet learnr. En outre, il existe un volet “Connexions” pour les connexions aux bases de données externes. Si vous avez lié le répertoire actif à un dépôt sur Github, il y aura également un volet “Git”.
Volets Graphiques, visionneuse, paquets et aide
Le volet inférieur droit comprend plusieurs onglets importants. Les graphiques de tracé typiques, y compris les cartes, s’affichent dans le volet Tracé. Les sorties interactives ou HTML s’affichent dans le volet Visionneuse. Le volet Aide permet d’afficher la documentation et les fichiers d’aide. Le volet Fichiers est un navigateur qui peut être utilisé pour ouvrir ou supprimer des fichiers. Le volet Paquets vous permet de voir, d’installer, de mettre à jour, de supprimer, de charger/décharger des paquets R et de voir quelle version du paquet vous avez. Pour en savoir plus sur les paquets, consultez la section paquets ci-dessous.
Ce volet contient les équivalents Stata des fenêtres Plots Manager et Project Manager.
Paramètres RStudio
Modifiez les paramètres et l’apparence de RStudio dans le menu déroulant Outiles, en sélectionnant Options globales. Vous pouvez y modifier les paramètres par défaut, y compris l’apparence/couleur de fond.
Redémarrage
Si votre R se fige, vous pouvez redémarrer R en allant dans le menu Session et en cliquant sur “Redémarrer R”. Cela vous évite de devoir fermer et ouvrir RStudio. Tout ce qui se trouve dans votre environnement R sera supprimé lorsque vous ferez cela.
Raccourcis clavier
Vous trouverez ci-dessous quelques raccourcis clavier très utiles. Vous trouverez tous les raccourcis clavier pour Windows, Max et Linux sur la deuxième page de ce fichier technique par RStudio.
| Windows/Linux | Mac | Action |
|---|---|---|
| Esc | Esc | Interrompre la commande en cours (utile si vous avez accidentellement lancé une commande incomplète et que vous ne pouvez pas éviter de voir “+” dans la console R) |
| Ctrl+s | Cmd+s | Sauvegarder (script) |
| Tab | Tab | Autocomplétion |
| Ctrl + Enter | Cmd + Enter | Exécuter la ou les ligne(s) courante(s)/sélection(s) de code |
| Ctrl + Shift + C | Cmd + Shift + c | commenter/dé-commenter les lignes souslignées |
| Alt + - | Option + | Insérer <-
|
| Ctrl + Shift + m | Cmd + Shift + m | Insérer %>%
|
| Ctrl + l | Cmd + l | Effacer le contenu de la console R |
| Ctrl + Alt + b | Cmd + Option + b | Exécuter du début à la ligne courante |
| Ctrl + Alt + t | Cmd + Option + t | Exécuter la section de code actuelle (R Markdown) |
| Ctrl + Alt + i | Cmd + Shift + r | Insérer un morceau de code (en R Markdown) |
| Ctrl + Alt + c | Cmd + Option + c | Exécuter le morceau de code actuel (en R Markdown) |
| Flèches haut/bas dans la console R | Idem | Basculer entre les commandes récemment exécutées |
| Shift + flèches haut/bas dans le script | Idem | Sélectionner plusieurs lignes de code |
| Ctrl + f | Cmd + f | Rechercher et remplacer dans le script actuel |
| Ctrl + Shift + f | Cmd + Shift + f | Rechercher dans les dossiers (rechercher/remplacer dans plusieurs scripts) |
| Alt + l | Cmd + Option + l | Plier le code sélectionné |
| Shift + Alt + l | Cmd + Shift + Option+l | Déplier le code sélectionné |
TIP: Utilisez votre touche de tabulation lorsque vous tapez pour activer la fonctionnalité de complétion automatique de RStudio. Cela peut éviter les fautes d’orthographe. Appuyez sur la touche Tab pendant la saisie pour produire un menu déroulant de fonctions et d’objets probables, en fonction de ce que vous avez tapé jusqu’à présent.
3.5 Fonctions
Les fonctions sont au cœur de l’utilisation de R. Les fonctions vous permettent d’effectuer des tâches et des opérations. De nombreuses fonctions sont installées avec R, beaucoup d’autres sont disponibles à télécharger dans des paquets (expliqués dans la section paquets), et vous pouvez même écrire vos propres fonctions personnalisées !
Cette section de base sur les fonctions explique :
- Ce qu’est une fonction et comment elle fonctionne
- Ce que sont les paramètres des fonctions
- Comment obtenir de l’aide pour comprendre une fonction
Une note rapide sur la syntaxe : Dans ce manuel, les fonctions sont écrites en code-texte avec des parenthèses vides, comme ceci : filter(). Comme expliqué dans la section paquets, les fonctions sont téléchargées dans des paquets. Dans ce manuel, les noms de paquets sont écrits en gras, comme dplyr. Parfois, dans le code d’exemple, vous pouvez voir le nom de la fonction lié explicitement au nom de son paquet avec deux points de suspension (::) comme ceci : dplyr::filter(). Le but de ce lien est expliqué dans la section sur les paquets.
Fonctions simples
Une fonction est comme une machine qui reçoit des entrées, effectue une action avec ces entrées, et produit une sortie. La nature de la sortie dépend de la fonction.
Les fonctions opèrent généralement sur un objet placé entre les parenthèses de la fonction. Par exemple, la fonction sqrt() calcule la racine carrée d’un nombre :
sqrt(49)[1] 7L’objet fourni à une fonction peut également être une colonne dans un jeu de données (voir la section Objets pour plus de détails sur tous les types d’objets). Comme R peut stocker plusieurs jeux de données, vous devrez spécifier à la fois le jeu de données et la colonne. Une façon de le faire est d’utiliser la notation $ pour lier le nom du jeu de données et le nom de la colonne (dataset$column). Dans l’exemple ci-dessous, la fonction summary() est appliquée à la colonne numérique age du jeu de données linelist, et la sortie est un résumé des valeurs numériques et manquantes de la colonne.
# Imprimez les statistiques sommaires de la colonne 'age' dans le jeu de données 'linelist'.
summary(linelist$age) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.07 23.00 84.00 86 NOTE: En coulisses, une fonction représente un code supplémentaire complexe qui a été regroupé pour l’utilisateur dans une seule commande simple.
Fonctions à paramètres multiples
Les fonctions demandent souvent plusieurs entrées, appelées paramètres, situées entre les parenthèses de la fonction, généralement séparées par des virgules.
- Certains paramètres sont obligatoires pour que la fonction fonctionne correctement, d’autres sont facultatifs
- Les paramètres facultatifs ont des valeurs par défaut
- Les paramètres peuvent prendre des entrées de type caractère, numérique, logique (VRAI/FAUX) et autres.
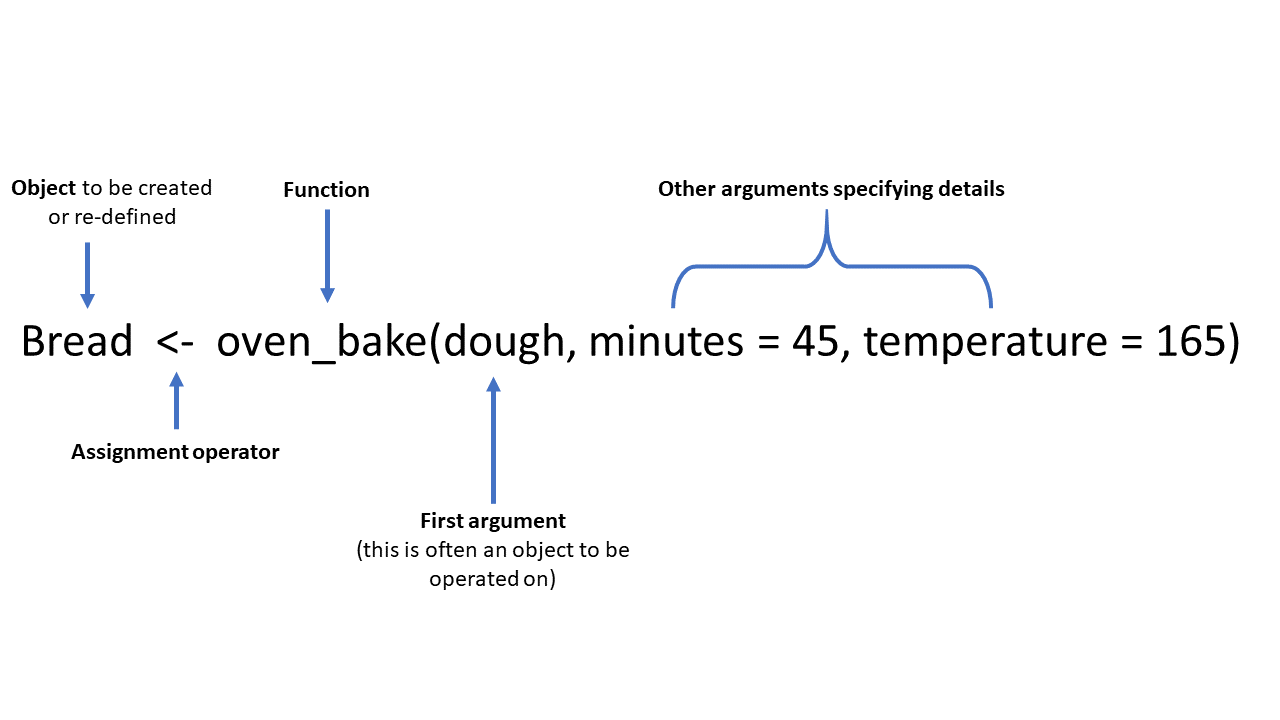
Voici une fonction fictive amusante, appelée oven_bake() (cuisson au four), comme exemple d’une fonction typique. Elle prend un objet comme entrée (par exemple un jeu de données, ou dans cet exemple “pâte”) et effectue des opérations sur celui-ci comme spécifié par des paramètres supplémentaires (minutes = et température =). La sortie peut être imprimée sur la console, ou sauvegardée comme un objet en utilisant l’opérateur d’affectation <-.
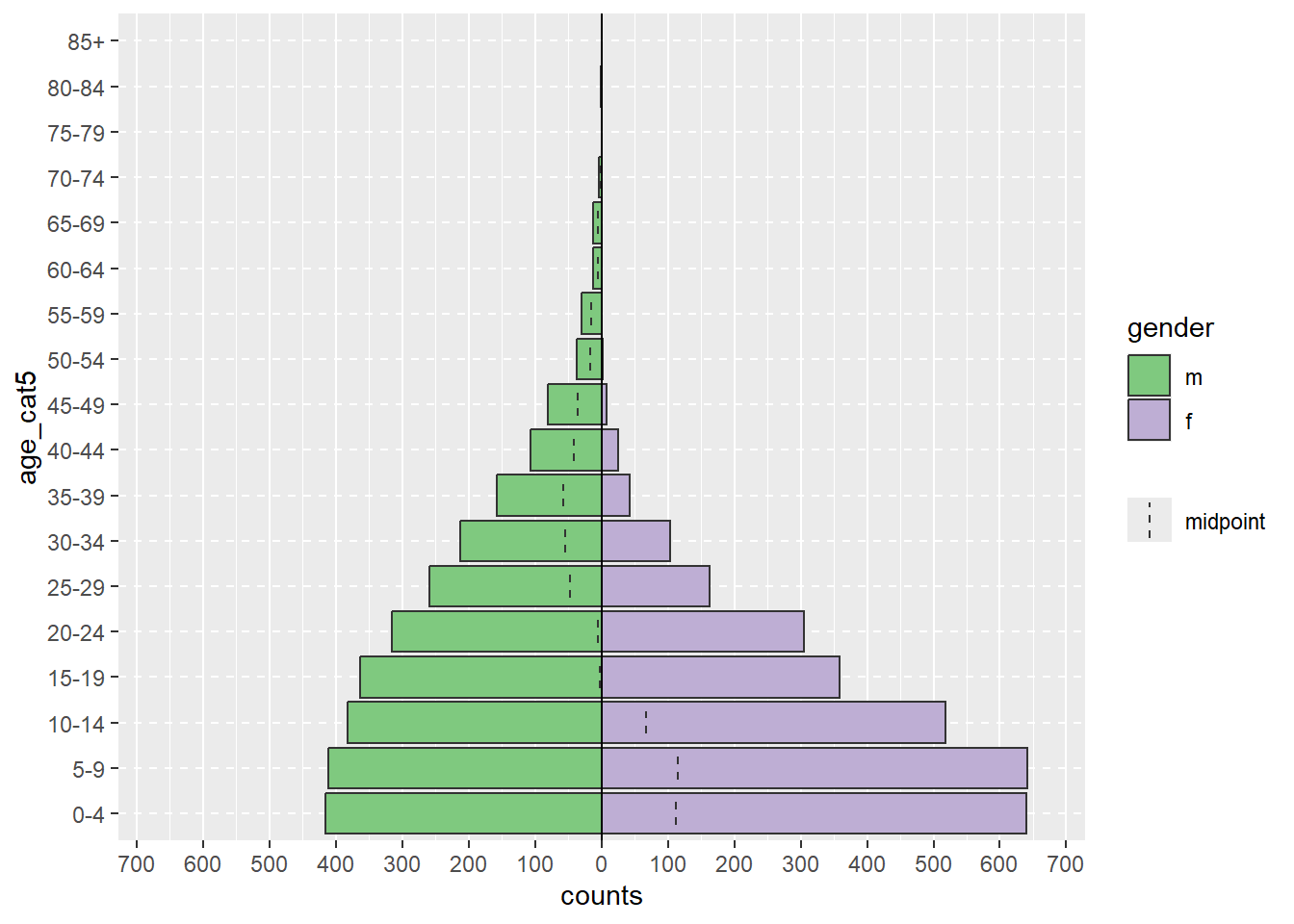
Dans un exemple plus réaliste, la commande age_pyramid() ci-dessous produit un graphique de pyramide des âges basé sur des groupes d’âge définis et une colonne de division binaire, comme le genre gender. La fonction reçoit trois paramètres entre parenthèses, séparés par des virgules. Les valeurs fournies aux paramètres établissent linelist comme le cadre de données à utiliser, age_cat5 comme la colonne à compter, et gender comme la colonne binaire à utiliser pour diviser la pyramide par couleur.
# Créer une pyramide des âges
age_pyramid(data = linelist, age_group = "age_cat5", split_by = "gender")La commande ci-dessus peut être écrite de manière équivalente comme ci-dessous, dans un style plus long avec une nouvelle ligne pour chaque argument. Ce style peut être plus facile à lire, et plus facile d’écrire des “commentaires” avec # pour expliquer chaque partie (commenter abondamment est une bonne pratique !). Pour exécuter cette commande plus longue, vous pouvez souligner la commande entière et cliquer sur “Run”, ou simplement placer votre curseur sur la première ligne et appuyer simultanément sur les touches Ctrl et Enter.
# Créer une pyramide des âges
age_pyramid(
data = linelist, # utiliser la liste linéaire des cas
age_group = "age_cat5", # fournir une colonne de groupe d'âge
split_by = "gender" # utiliser la colonne genre pour les deux côtés de la pyramide
)La première moitié d’une affectation de paramètre (par exemple data =) n’a pas besoin d’être spécifiée si les paramètres sont écrits dans un ordre spécifique (spécifié dans la documentation de la fonction). Le code ci-dessous produit exactement la même pyramide que ci-dessus, parce que la fonction attend l’ordre des paramètres : cadre de données, le variable age_group, puis le variable split_by.
# Cette commande produira exactement le même graphique que ci-dessus
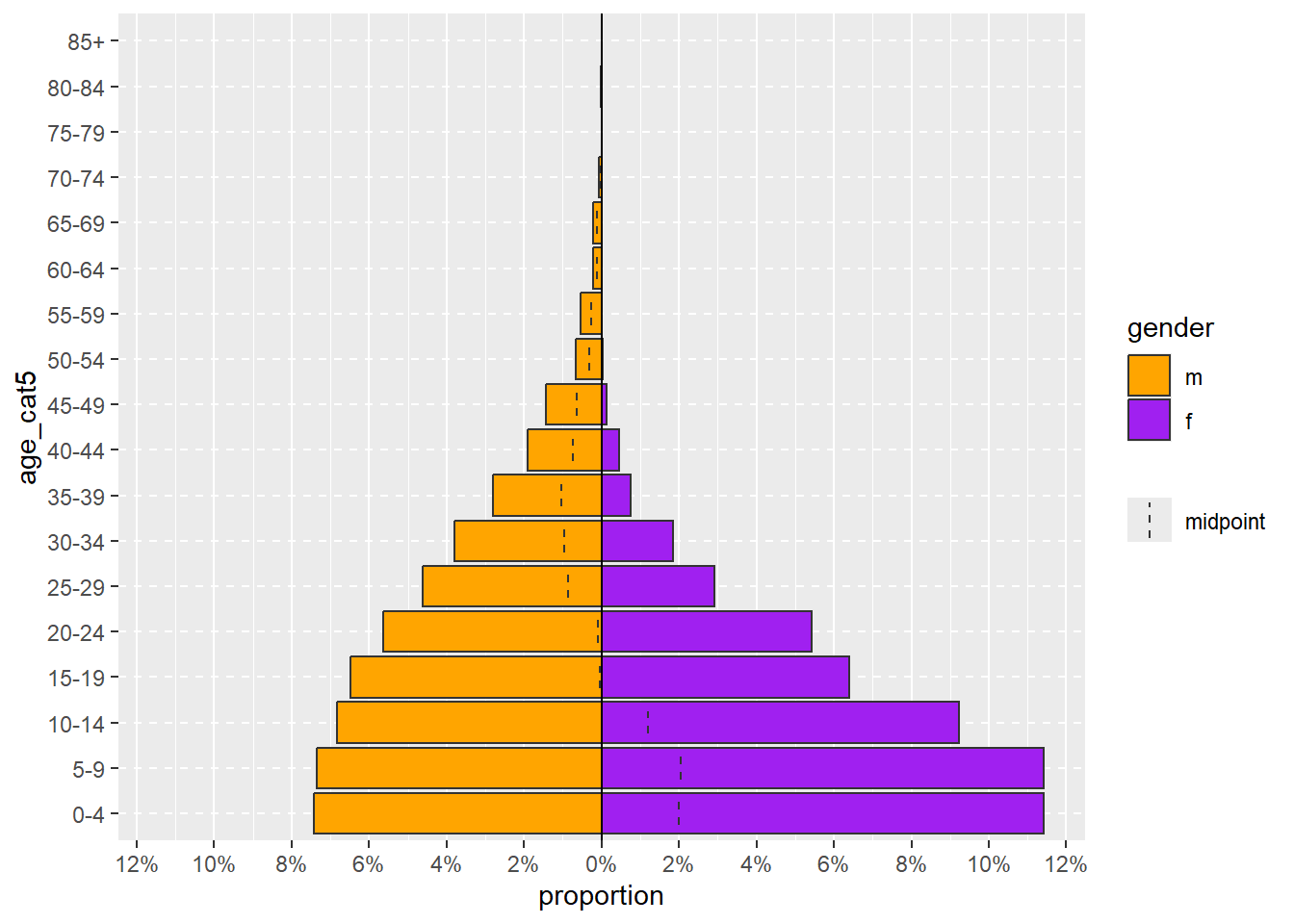
age_pyramid(linelist, "age_cat5", "gender")Une commande age_pyramid() plus complexe pourrait inclure les paramètres optionnels pour :
- Afficher les proportions au lieu des nombres (définissez
proportional = TRUE(vrai) quand la valeur par défaut estFALSE(faux)) - Spécifier les deux couleurs à utiliser (
pal =est l’abréviation de “palette” et est fourni avec un vecteur de deux noms de couleurs. Voir la page objets pour savoir comment la fonctionc()fabrique un vecteur).
NOTE: Pour les paramètres que vous spécifiez avec les deux parties du paramètre (par exemple proportional = TRUE), leur ordre parmi tous les paramètres n’a pas d’importance.
Ecrire des fonctions
R est un langage orienté autour des fonctions, vous devez donc vous sentir capable d’écrire vos propres fonctions. La création de fonctions présente plusieurs avantages :
- Faciliter la programmation modulaire - la séparation du code en morceaux indépendants et gérables
- Remplacer le copier-coller répétitif, qui peut être source d’erreurs
- Donner des noms mémorisables aux morceaux de code
L’écriture d’une fonction est traitée en détail à la page Écriture de fonctions.
3.6 Paquets
Les paquets contiennent des fonctions.
Un paquet en R est un ensemble partageable de code et de documentation qui contient des fonctions prédéfinies. Les utilisateurs de la communauté R développent en permanence des packages répondant à des problèmes spécifiques; donc il est probable que l’un d’entre eux puisse vous aider dans votre travail ! Vous allez installer et utiliser des centaines de paquets dans votre utilisation de R.
À l’installation, R contient des paquets et des fonctions “de base” qui effectuent des tâches élémentaires communes. Mais de nombreux utilisateurs de R créent des fonctions spécialisées, qui sont vérifiées par la communauté R et que vous pouvez télécharger en tant que paquet pour votre propre usage. Dans ce manuel, les noms des paquets sont écrits en gras. L’un des aspects les plus difficiles de R est qu’il existe souvent de nombreuses fonctions ou paquets parmi lesquels on peut choisir pour effectuer une tâche donnée.
Installer et charger
Les fonctions sont contenues dans des paquets qui peuvent être téléchargés (“installés”) sur votre ordinateur à partir d’Internet. Une fois qu’un paquet est téléchargé, il est stocké dans votre “bibliothèque”. Vous pouvez alors accéder aux fonctions qu’il contient pendant votre séance R actuelle en “chargeant” le paquet.
Pensez à R comme votre bibliothèque personnelle : Lorsque vous téléchargez un paquet, votre bibliothèque gagne un nouveau livre de fonctions, mais chaque fois que vous voulez utiliser une fonction de ce livre, vous devez emprunter (“charger”) ce livre dans votre bibliothèque.
En résumé : pour utiliser les fonctions disponibles dans un paquet R, deux étapes doivent être mises en œuvre :
- Le paquet doit être installé (une fois), et
- Le paquet doit être chargé (à chaque séance R)
Votre bibliothèque
Votre “bibliothèque” est en fait un dossier sur votre ordinateur, contenant un dossier pour chaque paquet qui a été installé. Déterminez où R est installé sur votre ordinateur, et cherchez un dossier appelé “win-library”. Par exemple : R\win-library\4.0 (4.0 est la version de R). Notez que vous aurez une bibliothèque différente pour chaque version de R que vous avez téléchargée.
Vous pouvez imprimer le chemin d’accès à votre bibliothèque en entrant.libPaths() (parenthèses vides). Ceci devient particulièrement important si vous travaillez avec R sur des lecteurs réseau.
Installer à partir du CRAN
Le plus souvent, les utilisateurs de R téléchargent des paquets depuis CRAN. CRAN (Comprehensive R Archive Network) est un entrepôt public en ligne de paquets R qui ont été publiés par des membres de la communauté R.
Vous vous inquiétez des virus et de la sécurité lorsque vous téléchargez un paquet depuis CRAN ? Lisez cet article à ce sujet.
Syntaxe du code
Pour plus de clarté dans ce manuel, les fonctions sont parfois précédées du nom de leur paquet en utilisant le symbole :: de la manière suivante : nom_du_paquet::nom_de_la_fonction().
Une fois qu’un paquet est chargé pour une séance, ce style explicite n’est plus nécessaire. On peut simplement utiliser nom_de_la_fonction(). Cependant, écrire le nom du paquet est utile lorsqu’un nom de fonction est commun et peut exister dans plusieurs paquets (par exemple, plot()). L’écriture du nom du paquet chargera également le paquet s’il n’est pas déjà chargé.
# Cette commande utilise le paquet "rio" et sa fonction "import()" pour importer un jeu de données
linelist <- rio::import("linelist.xlsx", which = "Sheet1")Aide sur les fonctions
Pour en savoir plus sur une fonction, vous pouvez la rechercher dans l’onglet Aide du RStudio en bas à droite. Vous pouvez également lancer une commande comme ?thefunctionname (mettez le nom de la fonction après un point d’interrogation) et la page d’aide apparaîtra dans le volet d’aide. Enfin, essayez de rechercher des ressources en ligne.
Mettre à jour les paquets
Vous pouvez mettre à jour les paquets en les réinstallant. Vous pouvez également cliquer sur le bouton vert “Update” dans votre panneau “RStudio Packages” pour voir quels paquets ont de nouvelles versions à installer. Sachez que votre ancien code peut avoir besoin d’être mis à jour s’il y a une révision majeure du fonctionnement d’une fonction !
Supprimer des paquets
Utilisez p_delete() de pacman, ou remove.packages() de base R. Alternativement, allez chercher le dossier qui contient votre bibliothèque et supprimez manuellement le dossier.
Dépendances
Les paquets dépendent souvent d’autres paquets pour fonctionner. Ceux-ci sont appelés dépendances. Si une dépendance ne s’installe pas, le paquet qui en dépend peut également ne pas s’installer.
Voir les dépendances d’un paquet avec p_depends(), et voir quels paquets en dépendent avec p_depends_reverse().
Fonctions masquées
Il n’est pas rare que deux paquets ou plus contiennent le même nom de fonction. Par exemple, le paquet dplyr possède une fonction filter(), mais le paquet stats aussi. La fonction filter() par défaut dépend de l’ordre dans lequel ces paquets sont chargés pour la première fois dans la séance R - le dernier sera la fonction par défaut de la commande filter().
Vous pouvez vérifier l’ordre dans votre panneau Environnement de R Studio - cliquez sur la liste déroulante pour “Global Environment” et voyez l’ordre des paquets. Les fonctions des paquets inférieurs dans cette liste déroulante masqueront les fonctions du même nom dans les paquets qui apparaissent plus haut dans la liste déroulante. Lors du premier chargement d’un paquet, R vous avertira dans la console si le masquage se produit, mais il est facile de ne pas le voir.
Voici comment vous pouvez corriger le masquage :
- Spécifiez le nom du paquet dans la commande. Par exemple, utilisez
dplyr::filter() - Réorganisez l’ordre dans lequel les paquets sont chargés (par exemple, dans
p_load()), et démarrez une nouvelle séance R.
Détacher / décharger
Pour détacher (décharger) un paquet, utilisez cette commande, avec le nom correct du paquet et un seul deux-points. Notez que cela peut ne pas résoudre le masquage.
detach(package:NOM_DU_PAQUET_ICI, unload = TRUE)Installer une ancienne version
Consultez ce guide pour installer une ancienne version d’un paquet particulier.
Paquets suggérés
Voir la page Paquets suggérés pour une liste de paquets que nous recommandons pour l’épidémiologie quotidienne.
3.7 Scripts
Les scripts sont une partie fondamentale de la programmation. Ce sont des documents qui contiennent vos commandes (par exemple, des fonctions pour créer et modifier des jeux de données, imprimer des visualisations, etc). Vous pouvez sauvegarder un script et l’exécuter à nouveau ultérieurement. Le stockage et l’exécution de vos commandes à partir d’un script présentent de nombreux avantages (par rapport à la saisie des commandes une par une dans la “ligne de commande” de la console R) :
- Portabilité : vous pouvez partager votre travail avec d’autres personnes en leur envoyant vos scripts
- Reproductibilité : pour que vous et les autres sachiez exactement ce que vous avez fait
- Contrôle de version : pour que vous puissiez suivre les modifications apportées par vous-même ou par vos collègues
- Commentaire/annotation : pour expliquer à vos collègues ce que vous avez fait
Commentaire
Dans un script, vous pouvez également annoter (“commenter”) votre code R. Les commentaires sont utiles pour expliquer à vous-même et aux autres lecteurs ce que vous faites. Vous pouvez ajouter un commentaire en tapant le symbole dièse (#) et en écrivant votre commentaire après. Le texte commenté apparaîtra dans une couleur différente de celle du code R.
Tout code écrit après le # ne sera pas exécuté. Par conséquent, placer un # avant le code est également un moyen utile de bloquer temporairement une ligne de code (“commenter”) si vous ne souhaitez pas la supprimer). Vous pouvez mettre en commentaire plusieurs lignes à la fois en les soulignant et en appuyant sur Ctrl+Shift+c (Cmd+Shift+c sur Mac).
# Un commentaire peut être sur une ligne par lui-même, ex.:
# Importer des données:
linelist <- import("linelist_raw.xlsx") %>% # un commentaire peut aussi venir après le code
# filter(age > 50)
# Il peut aussi être utilisé pour désactiver une ligne de code
count()Vous trouverez ci-dessous quelques conseils essentiels pour commenter et annoter votre code :
- Commentez ce que vous faites et pourquoi vous le faites
- Découpez votre code en sections logiques
- Accompagnez votre code d’une description textuelle étape par étape de ce que vous faites (par exemple, des étapes numérotées).
Style
Il est important d’être conscient de votre style de codage, surtout si vous travaillez en équipe. Nous préconisons le tidyverse guide de style. Il existe également des paquets tels que styler et lintr qui vous aident à vous conformer à ce style.
Quelques points très basiques pour rendre votre code lisible pour les autres:
- Lorsque vous nommez des objets, n’utilisez que des lettres minuscules, des chiffres et des traits de soulignement
_, par exemplemes_donnees - Utilisez fréquemment des espaces, y compris autour des opérateurs, par exemple
n = 1etage_nouveau <- age_vieillesse + 3.
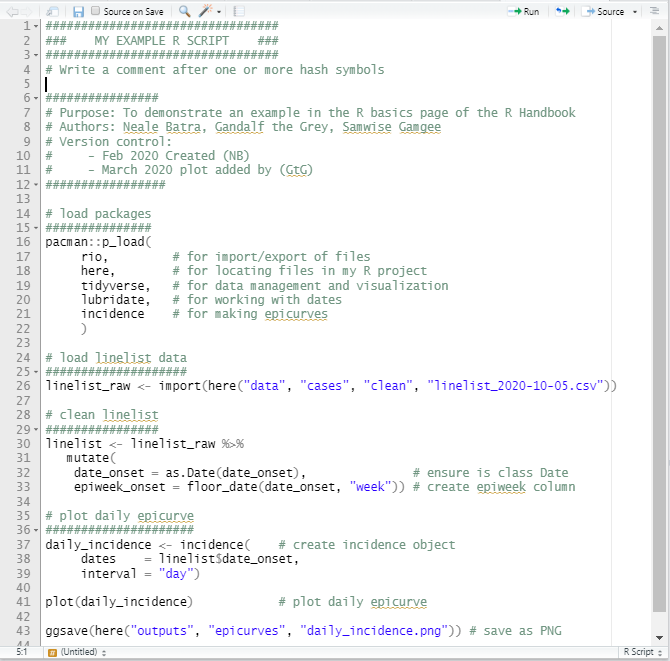
Exemple de script
Vous trouverez ci-dessous un exemple d’un court script R. N’oubliez pas que plus vous expliquerez succinctement votre code dans les commentaires, plus vos collègues vous apprécieront !
R markdown
Un script R markdown est un type de script R dans lequel le script lui-même devient un document de sortie (PDF, Word, HTML, Powerpoint, etc.). Ce sont des outils incroyablement utiles et polyvalents, souvent utilisés pour créer des rapports dynamiques et automatisés. Même ce site Web et ce manuel sont produits à l’aide de scripts R markdown !
Il convient de noter que les utilisateurs débutants de R peuvent également utiliser R Markdown - ne vous laissez pas intimider !Pour en savoir plus, consultez la page du manuel consacrée aux rapports avec R Markdown.
Carnets de notes R
Il n’y a pas de différence entre écrire dans un Rmarkdown et un R notebook. Cependant, l’exécution du document diffère légèrement. Voir ce site pour plus de détails.
Shiny
Les applications/sites web Shiny sont contenus dans un script, qui doit être nommé app.R. Ce fichier comporte trois éléments :
- Une interface utilisateur (ui)
- Une fonction serveur
- Un appel à la fonction
shinyApp
Consultez la page du manuel sur les teableaux de bord avec Shiny, ou ce tutoriel en ligne : Tutoriel Shiny
Auparavant, le fichier ci-dessus était divisé en deux fichiers (ui.R et server.R).
Repli du code
Vous pouvez replier des portions de code pour rendre votre script plus facile à lire.
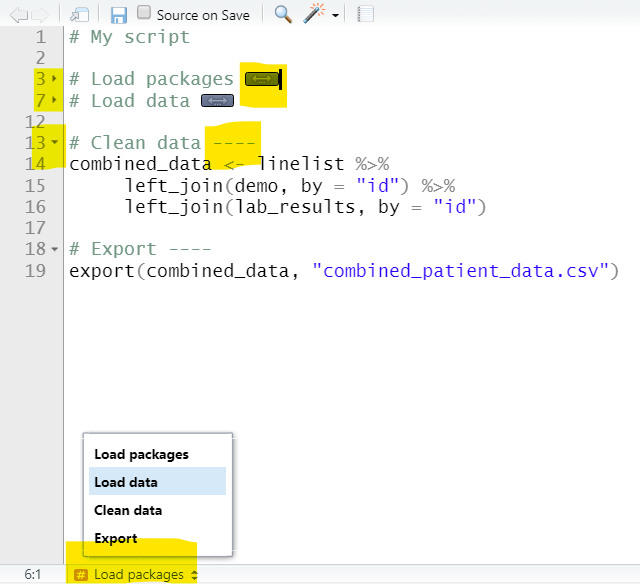
Pour ce faire, créez un en-tête de texte avec #, écrivez votre en-tête, et faites-le suivre d’au moins 4 tirets (-), hachages (#) ou égaux (=). Lorsque vous aurez fait cela, une petite flèche apparaîtra dans la “gouttière” à gauche (près du numéro de ligne). Vous pouvez cliquer sur cette flèche et sur le code situé en dessous jusqu’à ce que l’en-tête suivant se réduise et qu’une icône à double flèche apparaisse à sa place.
Pour développer le code, cliquez à nouveau sur la flèche dans la gouttière ou sur l’icône à double flèche. Il existe également des raccourcis clavier, comme expliqué dans la section RStudio de cette page.
En créant des en-têtes avec #, vous activerez également la table des matières au bas de votre script (voir ci-dessous) que vous pouvez utiliser pour naviguer dans votre script. Vous pouvez créer des sous-titres en ajoutant d’autres symboles, par exemple # pour les titres primaires, ## pour les titres secondaires et ### pour les titres tertiaires.
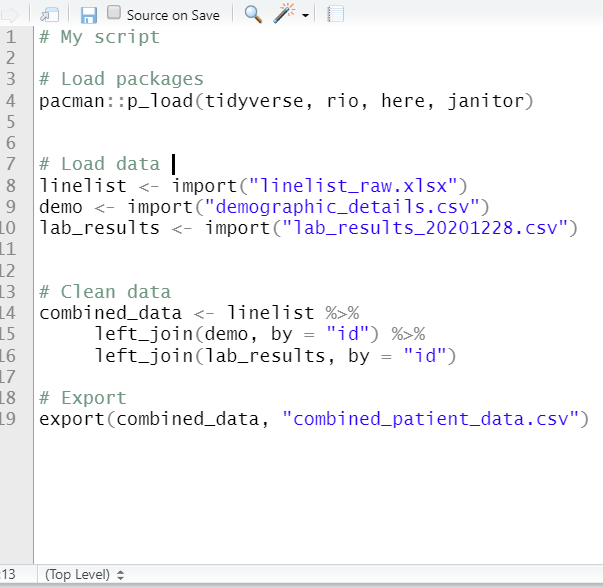
Vous trouverez ci-dessous deux versions d’un exemple de script. À gauche, l’original avec des en-têtes commentés. À droite, quatre tirets ont été écrits après chaque en-tête, les rendant ainsi repliables. Deux d’entre eux ont été réduits, et vous pouvez voir que la table des matières en bas de page affiche maintenant chaque section.
D’autres zones de code qui sont automatiquement éligibles pour le pliage incluent les régions “accolées” avec des parenthèses { } telles que les définitions de fonctions ou les blocs conditionnels (instructions “if else”). Vous pouvez en savoir plus sur le pliage du code sur le site RStudio.
3.8 Répertoire de travail
Le répertoire de travail est l’emplacement du dossier racine utilisé par R pour votre travail - où R recherche et enregistre les fichiers par défaut. Par défaut, il enregistrera de nouveaux fichiers et sorties à cet emplacement et recherchera ici des fichiers (par exemple, des ensembles de données).
Le répertoire de travail apparaît dans le texte gris en haut du volet de la console RStudio. Vous pouvez également imprimer le répertoire de travail actuel en exécutant getwd() (laissez les parenthèses vides).
Approche recommandée
Voir la page sur projets R pour plus de détails sur notre approche recommandée pour gérer votre répertoire de travail.
Un moyen commun, efficace et sans problème de gérer votre répertoire de travail et vos chemins de fichier consiste à combiner ces trois éléments dans un flux de travail du projets R orienté comme expliqué ci-dessous:
- Un projet R pour stocker tous vos fichiers (voir page sur projets R)
- Le paquet here pour localiser les fichiers (voir page sur importer et exporter)
- Le paquet rio pour importer ou exporter des fichiers (voir page sur importer et exporter)
Définir le répertoire de travail par commande
Jusqu’à récemment, de nombreuses personnes apprenant R ont appris à commencer leurs scripts avec une commande setwd(). Veuillez plutôt envisager d’utiliser un flux de travail orienté par projets R et lire les raisons de ne pas utiliser setwd().
En bref, votre travail devient spécifique à votre ordinateur, les chemins de fichier utilisés pour importer et exporter des fichiers deviennent “cassants”, ce qui entrave gravement la collaboration et l’utilisation de votre code sur tout autre ordinateur. Heureusement il existe des alternatives faciles!
Comme indiqué ci-dessus, bien que nous ne recommandons pas cette approche dans la plupart des cas, vous pouvez utiliser la commande setwd() avec le chemin du fichier de dossier souhaité dans les citations, par exemple:
setwd("C:/Documents/R Files/My analysis")DANGER: Définition d’un répertoire de travail avec setwd() peut être “cassant” si le chemin de fichier est spécifique à un ordinateur. Au lieu de cela, utilisez des chemins de fichier par rapport à un répertoire racine du projet R (avec le paquet here).
Définir manuellement le répertoire de travail
Pour définir le répertoire de travail manuellement (l’équivalent graphique du setwd()), cliquez sur le menu déroulant “Session” et accédez à “Set Working Directory”, puis “Choose Directory”. Cela définira le répertoire de travail pour cette scéance spécifique de R. Remarque: Si vous utilisez cette approche, vous devrez le faire manuellement chaque fois que vous ouvrez Rstudio.
Définir le répertoire de travail dans un projet R
Si vous utilisez un projet R, le répertoire de travail sera par défaut dans le dossier racine du projet R qui contient le fichier .rproj. Cela s’appliquera si vous ouvrez RStudio en cliquant sur le projet R (le fichier avec l’extension .rproj).
3.8.1 Répertoire de travail dans un script R Markdown
Dans un script R Markdown, le répertoire de travail par défaut est le dossier ou le fichier RMarkdown (.rmd) est enregistré. Si vous utilisez un projet R et le paquet here, cela ne s’applique pas et le répertoire de travail sera here(), comme expliqué dans la page projets R.
Si vous souhaitez modifier le répertoire de travail d’une dossier RMarkdown autonome (qui ne fait pas partie d’un projet R), et vous utilisez setwd(), cela ne s’appliquera qu’à ce morceau de code spécifique. Pour modifier tous les morceaux de code dans une dossier RMarkdown, modifiez le morceau de configuration pour ajouter le paramètre root.dir =, comme ci-dessous:
knitr::opts_knit$set(root.dir = 'desired/directorypath')Il est beaucoup plus facile d’utiliser simplement le script RMarkdown dans un projet R et d’utiliser le paquet here.
Fournir des chemins de fichier
La source de frustration la plus commune pour un débutant R (au moins sur un ordinateur avec Windows) est de saisir un chemin de fichier pour importer ou exporter des données. Il existe une explication approfondie sur la meilleure façon de saisir les chemins de fichier de saisie dans la page importer et exporter, mais voici quelques points clés:
Chemins cassés
Vous trouverez ci-dessous un exemple de chemin de fichier “absolute” avec un “adresse complète”. Ceux-ci se casseront probablement s’ils sont utilisés par un autre ordinateur. Une exception est si vous utilisez un dossier sur un réseau partagé.
C:/Utilisateurs/Nom/Document/Logiciels analytiques/R/Projets/Analyse2019/data/mars2019.csvDirection de la barre oblique
Si vous saisissez un chemin de fichier, soyez conscient de la direction des barres obliques. Utilisez des barres obliques vers l’avant (/) pour séparer les composants, par exemple Data/Provincial.csv. Le défaut pour les ordinateurs avec Windows est de séparer les composants du chemin avec des barres obliques en arrière (\\). Vous devrez donc modifier la direction de chaque barre oblique. Si vous utilisez le paquet here comme décrit dans la page projets R, la direction des barres obliques n’est pas un problème.
Chemins de fichiers relatifs
Nous recommandons généralement de utiliser des fichiers avec chemins “relatifs” - c’est-à-dire le chemin par rapport à la racine de votre projet R. Vous pouvez le faire en utilisant le paquet here comme expliqué dans la page projets R. Un chemin de fichiers relatif peut ressembler à ceci:
# Importer csv Linelist à partir de données/listes linéare/propres/sous-dossiers d'un projet R
linelist <- import(here("data", "clean", "linelists", "marin_country.csv"))Même si vous utilisez des chemins de fichiers relatifs dans un projet R, vous pouvez toujours utiliser des chemins absolus pour importer/exporter des données en dehors de votre projet R.
3.9 Objets
Tout dans R est un objet, et R est une langue “orienté sur l’objet”. Les sections suivantes expliquent:
- Comment créer des objets (
<-) - Types d’objets (par exemple, trames de données, vecteurs ..)
- Comment accéder à des sous-parties d’objets (par exemple, des variables dans un jeu de données)
- Classes d’objets (ex. numérique, logique, nombres entieres, double, caractère, facteur)
Tout est un objet
Cette section est adaptée du projet R4Epis.Tout ce que vous stockez dans R - des ensembles de données, des variables, une liste de noms de villages, un nombre total de population, même des sorties telles que des graphiques - sont des objets qui sont attribués à un nom et peuvent être référencés dans les commandes ultérieures.
Un objet existe lorsque vous lui avez attribué une valeur (voir la section d’attribution ci-dessous). Lorsqu’une valeur lui est attribuée, l’objet apparaît dans l’environnement (voir le volet supérieur droit de RStudio). Il peut alors être exploité, manipulé, modifié et redéfini.
Définir des objets (<-)
Créez des objets en leur attribuant une valeur avec l’opérateur <-. Vous pouvez considérer l’opérateur d’affectation<- comme les mots “est défini comme”. Les commandes d’affectation suivent généralement un ordre standard:
nom_objet <- valeur (ou processus/calcul qui produit une valeur)
Par exemple, vous souhaiterez peut-être enregistrer la semaine de rapport épidémiologique en cours en tant qu’objet de référence dans le code ultérieur. Dans cet exemple, l’objet semaine_en_cours est créé lorsqu’il reçoit la valeur "2018-W10" (les guillemets en font une valeur de caractère). L’objet semaine_en_cours apparaîtra alors dans le volet Environnement de RStudio (en haut à droite) et pourra être référencé dans les commandes ultérieures.
Voir les commandes R et leur sortie dans les cases ci-dessous.
# Créer l'objet semaine_en_cours en lui attribuant une valeur:
semaine_en_cours <- "2018-W10"
# Imprime la valeur actuelle de l'objet semaine_en_cours dans la console:
semaine_en_cours[1] "2018-W10"NOTE: Notez que le [1] dans la sortie de la console R indique simplement que vous visualisez le premier élément de la sortie
ATTENTION: La valeur d’un objet peut être écrasée à tout moment en exécutant une commande d’affectation pour redéfinir sa valeur. Ainsi, l’ordre d’exécution des commandes est très important.
La commande suivante redéfinira la valeur de semaine_en_cours:
# Attribuer une NOUVELLE valeur à l'objet semaine_en_cours:
semaine_en_cours <- "2018-W51"
# Afficher la valeur actuelle de semaine_en_cours dans la console:
semaine_en_cours[1] "2018-W51"Signe égal =
Vous verrez également des signes égal dans le code R:
- Un double signe égal
==entre deux objets ou valeurs pose une question logique: “est-ce égal à cela?”. - Vous verrez également des signes égal dans les fonctions utilisées pour spécifier les valeurs des arguments d’un fonction (lisez-les dans les sections ci-dessous), par exemple
max(age, na.rm = TRUE). - Vous pouvez utiliser un seul signe égal
=à la place de<-pour créer et définir des objets, mais cela est déconseillé. Vous pouvez lire pourquoi cela est déconseillé ici.
Ensembles de données
Les ensembles de données sont également des objets (généralement des «dataframes») et doivent recevoir des noms lors de leur importation. Dans le code ci-dessous, l’objet linelist est créé et reçoit la valeur d’un fichier CSV importé avec le paquet rio et sa fonction import().
# <<linelist>> est créée et reçoit la valeur du fichier CSV importé:
linelist <- import("my_linelist.csv")Vous pouvez en savoir plus sur l’importation et l’exportation d’ensembles de données dans la section sur importer et exporter.
ATTENTION: Une note rapide sur la dénomination des objets:
- Les noms d’objets ne doivent pas contenir d’espaces, mais vous devez utiliser un trait de soulignement (_) ou un point (.) au lieu d’un espace.
- Les noms d’objets sont sensibles à la casse (lettres majuscules et minuscules; ce qui signifie que Dataset_A est différent de dataset_A).
- Les noms d’objets doivent commencer par une lettre (ne peuvent pas commencer par un chiffre comme 1, 2 ou 3).
Les sorties
Les sorties telles que les tableaux et les tracés fournissent un exemple de la façon dont les sorties peuvent être enregistrées en tant qu’objets ou simplement imprimées sans être enregistrées. Un tableau croisé du sexe et du résultat à l’aide de la fonction R base table() peut être imprimé directement sur la console R (sans être enregistré).
# Imprimé sur la console R uniquement:
table(linelist$gender, linelist$outcome)
Death Recover
f 1227 953
m 1228 950La même table peut également être enregistrée en tant qu’objet nommé. Ensuite, éventuellement, il peut être imprimé.
# Enregistrer:
gen_out_table <- table(linelist$gender, linelist$outcome)
# Imprimer:
gen_out_table
Death Recover
f 1227 953
m 1228 950Colonnes
Les colonnes d’un ensemble de données sont également des objets et peuvent être définies, écrasées et créées comme décrit ci-dessous dansla section sur les colonnes.
Vous pouvez utiliser l’opérateur d’affectation de base R pour créer une nouvelle colonne. Ci-dessous, la nouvelle colonne bmi (indice de masse corporelle) est créée, et pour chaque ligne la nouvelle valeur est le résultat d’une opération mathématique sur la valeur de la ligne dans les colonnes wt_kg et ht_cm.
# Créer une nouvelle colonne "bmi" en utilisant la syntaxe de base R:
linelist$bmi <- linelist$wt_kg / (linelist$ht_cm/100)^2Cependant, dans ce manuel, nous mettons l’accent sur une approche différente de la définition des colonnes, qui utilise la fonction mutate() du package dplyr et piping avec l’opérateur pipe (%>%). La syntaxe est plus facile à lire et il y a d’autres avantages expliqués dans la page nettoyage de donnees et fonctions essentielles.
Vous pouvez lire plus sur piping dans la section “Piping” ci-dessous.
# Créer une nouvelle colonne "bmi" en utilisant la syntaxe dplyr:
linelist <- linelist %>%
mutate(bmi = wt_kg / (ht_cm/100)^2)Structure d’objet
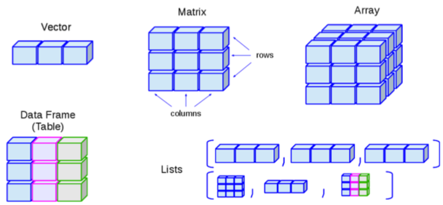
Les objets peuvent être une seule donnée (par exemple, “mon_numéro <-24”), ou ils peuvent être constitués de données structurées.
Le graphique ci-dessous est emprunté à ce tutoriel R en ligne. Il montre certaines structures de données courantes et leurs noms. Les données spatiales ne sont pas incluses dans cette image, qui sont abordées dans la page bases de GIS.
En épidémiologie (et en particulier en épidémiologie de terrain), vous rencontrerez le plus souvent des trames de données et des vecteurs:
| Structure commune | Explication | Exemple |
|---|---|---|
| Vecteurs | Un conteneur pour une séquence d’objets singuliers, tous de la même classe (par exemple numérique, caractère). |
Les “variables” (colonnes) dans les blocs de données sont des vecteurs (par exemple, la colonne age_years). |
| Trames de données | Vecteurs (par exemple, des colonnes) qui sont liés ensemble et qui ont tous le même nombre de lignes. |
linelist est une trame de données. |
Notez que pour créer un vecteur “autonome” (ne faisant pas partie d’un bloc de données), la fonction c() est utilisée pour combiner les différents éléments. Par exemple, si vous créez un vecteur de couleurs à appliquer à l’échelle de couleurs d’un tracé:
vector_of_colors <- c("blue", "red2", "orange", "grey")
Classes d’objets
Tous les objets stockés dans R ont une classe qui indique à R comment gérer l’objet. Il existe de nombreuses classes possibles, mais les plus courantes incluent:
| Classe | Explication | Exemples |
| Caractère | Ce sont des textes/mots/phrases “entre guillemets”. Les mathématiques ne peuvent pas être effectuées sur ces objets. | “Les objets caractères sont entre guillemets” |
| Entier | Nombres entiers uniquement (pas de décimales) | -5, 14 ou 2000 |
| Numérique | Ce sont des nombres et peuvent inclure des décimales. S’ils sont entre guillemets, ils seront considérés comme une classe de caractères. | 23.1 ou 14 |
| Facteur | Ce sont des vecteurs qui ont un ordre spécifié ou une hiérarchie de valeurs | Une variable de statut économique à valeurs ordonnées |
| Des dates | Une fois que R est informé que certaines données sont des dates, ces données peuvent être manipulées et affichées de manière spéciale. Voir la page sur manipuler les dates pour plus d’informations. | 2018-04-12 15/3/1954 mer, 4 janv, 1980 |
| Logique | Les valeurs doivent être l’une des deux valeurs spéciales TRUE ou FALSE (notez qu’elles ne sont pas “TRUE” et “FALSE” entre guillemets) |
TRUE ou FALSE |
| data.frame a.frame | Une trame de données est la façon dont R stocke un ensemble de données typiques. Il se compose de vecteurs (colonnes) de données liés entre eux, qui ont tous le même nombre d’observations (lignes). | L’exemple de jeu de données AJS nommé linelist_raw contient 68 variables avec 300 observations (lignes) chaque. |
| tibble | Les tibbles sont une variante du cadre de données; la principale différence opérationnelle étant qu’ils s’impriment mieux sur la console (affichent les 10 premières lignes et uniquement les colonnes qui tiennent sur l’écran) | Tout cadre de données, liste ou matrice peut être converti en tibble avec as_tibble()
|
| liste | Une liste est comme un vecteur, mais contient d’autres objets qui peuvent être d’autres classes différentes |
Vous pouvez tester la classe d’un objet en fournissant son nom à la fonction class(). Remarque : vous pouvez référencer une colonne spécifique dans un jeu de données en utilisant la notation «$» pour séparer le nom du jeu de données et le nom de la colonne.
# La classe doit être une trame de données ou un tibble:
class(linelist) [1] "data.frame"# La classe doit être numérique:
class(linelist$age)[1] "numeric"# La classe doit être caractère:
class(linelist$gender)[1] "character"Parfois, une colonne sera automatiquement convertie dans une classe différente par R. Attention à cela ! Par exemple, si vous avez un vecteur ou une colonne de nombres, mais qu’une valeur de caractère est insérée; toute la colonne deviendra un caractère de classe.
# Définir le vecteur avec des numéros:
num_vector <- c(1,2,3,4,5)
# Le vecteur est de classe "numérique":
class(num_vector) [1] "numeric"# Convertir le troisième élément en caractère:
num_vector[3] <- "three"
# Le vecteur est maintenant de classe "caractère"
class(num_vector) [1] "character"Un exemple courant de ceci est lors de la manipulation d’un bloc de données afin d’imprimer un tableau. Si vous faites une ligne totale et essayez de coller/coller ensemble des pourcentages dans la même cellule que des nombres (par exemple 23 (40%)), le toute la colonne numérique ci-dessus sera convertie en caractère et ne pourra plus être utilisée pour des calculs mathématiques. Parfois, vous devrez convertir des objets ou des colonnes dans une autre classe.
| Fonction | Action |
as.character() |
Convertit en classe “caractère” |
as.numeric() |
Convertit en classe “numérique” |
as.integer() |
Convertit en classe “entière” |
as.Date() |
Convertit en classe “Date” Remarque: voir le chapitre sur les dates pour plus de détails |
factor() |
Convertit en classe “facteur” Remarque: la redéfinition de l’ordre des niveaux de valeur nécessite des arguments supplémentaires |
De même, il existe des fonctions base R pour vérifier si un objet EST d’une classe spécifique, comme is.numeric(), is.character(), is.double(), is .facteur(), is.integer()
Voici plus de matériel en ligne sur les classes et les structures de données dans R.
Colonnes/Variables ($)
Une colonne dans un bloc de données est techniquement un “vecteur” (voir tableau ci-dessus) - une série de valeurs qui doivent toutes être de la même classe (caractère, numérique, logique, etc.).
Un vecteur peut exister indépendamment d’un bloc de données, par exemple un vecteur de noms de colonnes que vous souhaitez inclure en tant que variables explicatives dans un modèle. Pour créer un vecteur “autonome”, utilisez la fonction c() comme ci-dessous:
# Définir le vecteur autonome des valeurs de classe caractère:
var_explicatives <- c("gender", "fever", "chills", "cough", "aches", "vomit")
# Affiche les valeurs dans ce vecteur nommé:
var_explicatives[1] "gender" "fever" "chills" "cough" "aches" "vomit" Les colonnes d’un bloc de données sont également des vecteurs et peuvent être appelées, référencées, extraites ou créées à l’aide du symbole $. Le symbole $ relie le nom de la colonne au nom de son bloc de données. Dans ce manuel, nous essayons d’utiliser le mot “colonne” au lieu de “variable”.
# Récupérer la longueur du vecteur age:
length(linelist$age) # (l'âge est une colonne dans le bloc de données nomé "linelist")En tapant le nom de la trame de données suivi de $, vous verrez également un menu déroulant de toutes les colonnes de la trame de données. Vous pouvez les faire défiler à l’aide de votre touche fléchée, en sélectionner une avec votre touche Entrée et éviter les fautes d’orthographe !
CONSEIL AVANCÉ: Certains objets plus complexes (par exemple, une liste ou un objet epicontacts) peuvent avoir plusieurs niveaux accessibles via plusieurs signes dollar. Par exemple epicontacts$linelist$date_onset
Accès/index avec crochets ([ ])
Vous devrez peut-être afficher des parties d’objets, également appelées “indexation”, ce qui se fait souvent à l’aide des crochets [ ]. L’utilisation de $ sur une trame de données pour accéder à une colonne est également un type d’indexation.
# Définir le vecteur:
mon_vecteur <- c("a", "b", "c", "d", "e", "f")
# Imprimer le 5ème élément:
mon_vecteur[5][1] "e"Les crochets fonctionnent également pour renvoyer des parties spécifiques d’une sortie renvoyée, comme la sortie d’une fonction summary():
# Tout le résumé
summary(linelist$age) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.07 23.00 84.00 86 # Juste le deuxième élément du résumé, avec le nom (en utilisant uniquement des crochets simples)
summary(linelist$age)[2]1st Qu.
6 # Juste le deuxième élément, sans nom (en utilisant des doubles crochets)
summary(linelist$age)[[2]][1] 6# Extraire un élément par son nom, sans afficher le nom
summary(linelist$age)[["Median"]][1] 13Les crochets fonctionnent également sur les blocs de données pour afficher des lignes et des colonnes spécifiques. Vous pouvez le faire en utilisant la syntaxe dataframe[lignes, colonnes]:
# Afficher une ligne spécifique (2) du jeu de données, avec toutes les colonnes
# (n'oubliez pas la virgule!)
linelist[2,]
# Afficher toutes les lignes, mais une seule colonne:
linelist[, "date_onset"]
# Afficher les valeurs de la ligne 2 et des colonnes 5 à 10:
linelist[2, 5:10]
# Afficher les valeurs de la ligne 2 et des colonnes 5 à 10 et 18:
linelist[2, c(5:10, 18)]
# Afficher les lignes 2 à 20 et des colonnes spécifiques:
linelist[2:20, c("date_onset", "outcome", "age")]
# Afficher les lignes et les colonnes en fonction de critères
# *** Notez que le dataframe doit toujours être nommé dans les critères!
linelist[linelist$age > 25 , c("date_onset", "outcome", "age")]
# Utilisez View() pour voir les sorties dans le volet RStudio Viewer (plus facile à lire)
# *** Notez le "V" majuscule dans la fonction View()
View(linelist[2:20, "date_onset"])
# Enregistrer en tant que nouvel objet:
new_table <- linelist[2:20, c("date_onset")]Notez que vous pouvez également réaliser l’indexation des lignes/colonnes ci-dessus sur les blocs de données et les tibbles en utilisant la syntaxe dplyr (fonctions filter() pour les lignes et select() pour les colonnes). Pour en savoir plus sur ces fonctions principales, consultez la page sur le nettoyage de deonnees et fonctions essentielles.
Pour filtrer en fonction du “numéro de ligne”, vous pouvez utiliser la fonction dplyr row_number() avec des parenthèses ouvertes dans le cadre d’une instruction de filtrage logique. Vous utiliserez souvent l’opérateur %in% et une plage de nombres dans le cadre de cette instruction logique, comme indiqué ci-dessous. Pour voir les premières N lignes, vous pouvez également utiliser la fonction spéciale dplyr head().
# Afficher les 100 premières lignes:
linelist %>%
head(100)
# Afficher la ligne 5 uniquement:
linelist %>%
filter(row_number() == 5)
# Afficher les lignes 2 à 20 et trois colonnes spécifiques
# (notez qu'aucun guillemet n'est nécessaire sur les noms de colonne)
linelist %>%
filter(row_number() %in% 2:20) %>%
select(date_onset, issue, age)Lors de l’indexation d’un objet de classe list, les crochets simplesretournent toujours avec la classe list, même si un seul objet est retourné. Les crochets doubles, cependant, peuvent être utilisés pour accéder à un seul élément et renvoyer une classe différente de la liste.Les parenthèses peuvent également être écrites les unes après les autres, comme illustré ci-dessous.
Cette explication visuelle de l’indexation des listes, avec des poivrières est humoristique et utile.
# définir la liste des démos
ma_liste <- list(
# Le premier élément de la liste est un vecteur de caractères:
hopitaux = c("Central", "Empire", "Santa Anna"),
# Le deuxième élément de la liste est une trame de données d'adresses:
adresses = data.frame(
rue = c("145 Medical Way", "1048 Brown Ave", "999 El Camino"),
ville = c("Andover", "Hamilton", "El Paso")
)
)Voici à quoi ressemble la liste lorsqu’elle est imprimée sur la console. Voyez comment il y a deux éléments nommés:
-
hôpitaux, un vecteur de caractères -
adresses, une trame de données d’adresses
ma_liste$hopitaux
[1] "Central" "Empire" "Santa Anna"
$adresses
rue ville
1 145 Medical Way Andover
2 1048 Brown Ave Hamilton
3 999 El Camino El PasoMaintenant, nous extrayons, en utilisant diverses méthodes:
# Cela renvoie l'élément dans la classe "list" - le nom de l'élément est toujours affiché:
ma_liste[1] $hopitaux
[1] "Central" "Empire" "Santa Anna"# Cela ne renvoie que le vecteur de caractères (sans nom):
ma_liste[[1]][1] "Central" "Empire" "Santa Anna"# Vous pouvez également indexer par le nom de l'élément de la liste:
ma_liste[["hopitaux"]][1] "Central" "Empire" "Santa Anna"# Cela renvoie le troisième élément du vecteur de caractères "hôpitaux":
ma_liste[[1]][3] [1] "Santa Anna"# Cela renvoie la première colonne ("rue") de la trame de données d'adresse:
ma_liste[[2]][1] rue
1 145 Medical Way
2 1048 Brown Ave
3 999 El CaminoSupprimer des objets
Vous pouvez supprimer des objets individuels de votre environnement R en mettant le nom dans la fonction rm() (sans guillemets):
rm(nom_objet)Vous pouvez supprimer tous les objets (vider votre espace de travail) en exécutant:
rm(list = ls(all = TRUE))
3.10 Tuyauterie / “Piping” (%>%)
Deux approches générales pour travailler avec des objets sont:
- Pipes/tidyverse - les tuyaux envoient un objet d’une fonction à l’autre - l’accent est mis sur l’action, pas sur l’objet
- Définir les objets intermédiaires - un objet est redéfini encore et encore - l’accent est mis sur l’objet
Tuyaux / Pipes
Expliqué simplement, l’opérateur pipe (%>%) passe une sortie intermédiaire d’une fonction à la suivante. Vous pouvez penser que cela signifie “alors”. De nombreuses fonctions peuvent être liées avec %>%.
- Le tuyau met l’accent sur une séquence d’actions, et non sur l’objet sur lequel les actions sont effectuées
- Les tuyaux sont plus efficaces lorsqu’une séquence d’actions doit être effectuée sur un objet
- Les tuyaux proviennent du paquet magrittr, qui est automatiquement inclus dans les paquets dplyr et tidyverse
- Les tuyaux peuvent rendre le code plus propre et plus facile à lire, plus intuitif
En savoir plus sur cette approche dans le tidyverse guide de style
Voici un faux exemple de comparaison, utilisant des fonctions fictives pour “faire un gâteau”. Tout d’abord, la méthode du tuyau:
# Un faux exemple de comment faire cuire un gâteau en utilisant la syntaxe de tuyauterie:
gateau <- farine %>% # pour définir le gâteau, commencez par la farine, puis...
# ajouter des oeufs
add(oeufs) %>%
# ajouter de l'huile
add(huile) %>%
# ajouter de l'eau
add(eau) %>%
# mélanger ensemble avec cuillère pour 2 minutes:
mix_together(
ustensil = "spoon",
minutes = 2) %>%
# cuire à 200 degrés centigrade pour 35 minutes:
bake(
degrees = 200,
system = "centigrade",
minute = 35) %>%
# laissez-le refroidir
let_cool() Voici un autre lien décrivant l’utilitaire de tuyaux.
La tuyauterie n’est pas une fonction de base en R. Pour utiliser la tuyauterie, le paquet magrittr doit être installé et chargé (cela se fait généralement en chargeant le paquet tidyverse ou dplyr qui l’inclut). Vous pouvez en savoir plus sur la tuyauterie dans la documentation de magrittr.
Notez que, tout comme les autres commandes R, les tuyaux peuvent être utilisés pour afficher simplement le résultat ou pour enregistrer/réenregistrer un objet, selon que l’opérateur d’affectation <- est impliqué ou non. Voir les deux exemplaires ci-dessous:
# Créer ou écraser un objet, en le définissant sous
# forme de nombres agrégés par catégorie d'âge (non imprimé)
linelist_summary <- linelist %>%
count(age_cat)# Imprimez le tableau des comptes dans la console, mais ne l'enregistrez pas:
linelist %>%
count(age_cat) age_cat n
1 0-4 1095
2 5-9 1095
3 10-14 941
4 15-19 743
5 20-29 1073
6 30-49 754
7 50-69 95
8 70+ 6
9 <NA> 86%<>% Il s’agit d’un “tuyau d’affectation” du paquet magrittr, qui transmet un objet en avant et redéfinit également l’objet. Il doit être le premier opérateur pipe de la chaîne. C’est un raccourci. Les deux commandes ci-dessous sont équivalentes:
# Utilisez l'opérateur d'affectation:
linelist <- linelist %>%
filter(age > 50)
# Utilisez le tuyau d'affectation:
linelist %<>% filter(age > 50)Définir les objets intermédiaires
Cette approche de modification des objets ou trammes de données peut être meilleure si:
- Vous devez manipuler plusieurs objets
- Il y a des étapes intermédiaires qui sont significatives et méritent des noms d’objets séparés
Des risques:
- Créer de nouveaux objets pour chaque étape signifie créer beaucoup d’objets. Si vous utilisez le mauvais, vous ne vous en rendrez peut-être pas compte!
- Nommer tous les objets peut prêter à confusion
- Les erreurs peuvent ne pas être facilement détectables
Soit nommer chaque objet intermédiaire, soit écraser l’original, soit combiner toutes les fonctions ensemble. Tous viennent avec leurs propres risques.
Vous trouverez ci-dessous le même exemple de faux “gâteau” que ci-dessus, mais en utilisant ce style:
# un faux exemple de comment faire un gâteau en utilisant cette méthode
# (définissant des objets intermédiaires):
# Ajouter le farine et les oeufs:
pate_1 <- left_join(farine, oeufs)
# Ajouter l'huile:
pate_2 <- left_join (pate_1, huile)
# Ajouter l'eau:
pate_3 <- left_join(pate_2, eau)
# Melange tous ensemble:
pate_4 <- mix_together(object = pate_3,
ustensil = "spoon",
minutes = 2)
# Cuire le gâteau dans le four:
gateau <-bake(object = pate_4,
degrees = 200,
system = "centigrade",
minutes = 35)
# Laissez-le à refroidir:
gateau <- laisse_refroidir(gateau)Combinez toutes les fonctions ensemble - c’est difficile à lire :
# Un exemple de combinaison/imbrication de plusieurs fonctions - difficile à lire:
gateau <- let_cool(bake(mix_together(pate_3,
utensil = "spoon",
minutes = 2),
degrees = 200,
system = "centigrade",
minutes = 35))3.11 Opérateurs clés et fonctions
Cette section détaille les opérateurs dans R, tels que:
- Opérateurs définitionnels
- Opérateurs relationnels (inférieur à, égal aussi..)
- Opérateurs logiques (et, ou…)
- Gestion des valeurs manquantes
- Opérateurs et fonctions mathématiques (+/-, >, sum(), median(), …)
- L’opérateur
%in%
Opérateurs d’affectation
<-
L’opérateur d’affectation de base dans R est <-. Tel que nom_objet <- valeur. Cet opérateur d’affectation peut également être écrit comme =. Nous vous conseillons d’utiliser <- pour une utilisation générale de R.Nous conseillons également d’entourer ces opérateurs d’espaces, pour plus de lisibilité.
<<-
Si Fonctions d’écriture, ou si vous utilisez R de manière interactive avec des scripts sourcés, vous devrez peut-être utiliser cet opérateur d’affectation <<- (de base R). Cet opérateur est utilisé pour définir un objet dans un environnement R « parent » supérieur. Voir ceci référence en ligne.
%<>%
Il s’agit d’un “tuyau d’affectation” du paquet magrittr, qui dirige un objet vers l’avant et redéfinit également l’objet. Il doit être le premier opérateur pipe de la chaîne. Il s’agit d’un raccourci.
%<+%
Ceci est utilisé pour ajouter des données aux arbres phylogénétiques avec le package ggtree. Voir la page sur les arbres phylogénétiques ou ce livre de ressources en ligne.
Opérateurs relationnels et logiques
Les opérateurs relationnels comparent les valeurs et sont souvent utilisés lors de la définition de nouvelles variables et de sous-ensembles des blocs de données. Voici les opérateurs relationnels courants dans R:
| Sens | Opérateur | Exemple | Exemple de résultat | ||
| Égal à | == |
" A" = = "a" |
Notez que |
||
| Non égal à | != |
2 != 0 |
TRUE |
||
| Supérieur a | > |
4 > 2 |
TRUE |
||
| Moins de | < |
4 < 2 |
FALSE |
||
| Supérieur ou égal à | >= |
6 >= 4 |
TRUE |
||
| Inférieur ou égal à | <= |
6 <= 4 |
FALSE |
||
| Valeur manquante | is.na() |
is.na(7) |
voir page sur valeur manquante |
||
Valeur ne | !is.na() manque pas | |
!is.na(7) | TRUE | |
Les opérateurs logiques, tels que ET et OU, sont souvent utilisés pour connecter des opérateurs relationnels et créer des critères plus complexes. Les instructions complexes peuvent nécessiter des parenthèses ( ) pour le regroupement et l’ordre d’application.
| Sens | Opérateur |
|---|---|
| ET | & |
| OU |
| (barre verticale) |
| Parenthèses |
( ) Utilisé pour regrouper les critères et clarifier l’ordre des opérations |
Par exemple, ci-dessous, nous avons une liste linéaire avec deux variables que nous voulons utiliser pour créer notre définition de cas, resultat_tdr, un résultat d’un test rapide, et autres_cas_menage, qui nous dira s’il y a d’autres cas dans le ménage. La commande ci-dessous utilise la fonction case_when() pour créer la nouvelle variable case_def telle que:
linelist_propre <- linelist %>%
mutate(case_def = case_when(
is.na(resultat_tdr) & is.na(autres_cas_menage) ~ NA_character_,
resultat_tdr == "Positive" ~ "Confirmé",
resultat_tdr != "Positive" & other_cases_in_home == "Oui" ~ "Probable",
TRUE ~ "Suspect"
))| Critères dans l’exemple ci-dessus | Valeur dans “case_def” |
|---|---|
Si la valeur des variables resultat_tdr et autres_cas_menage est manquante |
NA (manquante) |
Si la valeur dans resultat_tdr est “Positive” |
“Confirmé” |
Si la valeur dans resultat_tdr n’est pas “Positive” ET la valeur dans autres_cas_menage est “Oui” |
“Probable” |
| Si l’un des critères ci-dessus n’est pas rempli | “Suspect” |
Notez que R est sensible à la casse, donc “Positif” est différent de “positif”…
Valeurs manquantes
Dans R, les valeurs manquantes sont représentées par la valeur spéciale NA (une valeur “réservée”) (lettres majuscules N et A - pas entre guillemets). Si vous importez des données qui enregistrent des données manquantes d’une autre manière (par exemple, 99, “Missing” ou .), vous pouvez recoder ces valeurs en “NA”. La procédure à suivre est expliquée dans la page importer et exporter.
Pour tester si une valeur est NA, utilisez la fonction spéciale is.na(), qui renvoie TRUE ou FALSE.
# 2 cas positives, un suspect et un inconnu:
resultat_tdr <- c("Positive", "Suspect", "Positive", NA)
# Verifier si il y' a des valeurs manquantes:
is.na(resultat_tdr)[1] FALSE FALSE FALSE TRUEEn savoir plus sur les valeurs manquantes, infinies, NULL et impossibles dans la page sur les valeur manquantes. Découvrez comment convertir les valeurs manquantes lors de l’importation de données dans la page sur importer et exporter.
Mathématiques et statistiques
Tous les opérateurs et fonctions de cette page sont automatiquement disponibles en utilisant base R.
Opérateurs mathématiques
Ceux-ci sont souvent utilisés pour effectuer des additions, des divisions, pour créer de nouvelles colonnes, etc. Vous trouverez ci-dessous des opérateurs mathématiques courants dans R. Que vous mettiez des espaces autour des opérateurs n’est pas important.
| Objectif | Exemple en R |
|---|---|
| addition | 2 + 3 |
| soustraction | 2 - 3 |
| multiplication | 2 * 3 |
| division | 30 / 5 |
| exposant | 2^3 |
| ordre des opérations | ( ) |
Fonctions mathématiques
| Objectif | Fonction |
|---|---|
| arrondir | round(x, digits = n) |
| arrondir | janitor::round_half_up(x, digits = n) |
| plafond (arrondi) | ceiling(x) |
| étage (arrondir à l’inférieur) | floor(x) |
| valeur absolue | abs(x) |
| racine carrée | sqrt(x) |
| exposant | exponent(x) |
| un algorithme naturel | log(x) |
| log à la base 10 | log10(x) |
| log à la base 2 | log2(x) |
Remarque: pour round(), les digits = spécifient le nombre de décimales placées. Utilisez signif() pour arrondir à un nombre de chiffres significatifs.
Notation scientifique
La probabilité d’utilisation de la notation scientifique dépend de la valeur de l’option “scipen”.
D’après la documentation de ?options: scipen est une pénalité à appliquer lors de la décision d’imprimer des valeurs numériques en notation fixe ou exponentielle. Les valeurs positives tendent vers la notation fixe et négatives vers la notation scientifique: la notation fixe sera préférée à moins qu’elle ne soit plus large de plus de ‘scipen’.
S’il est réglé sur un nombre faible (par exemple 0), il sera toujours “allumé”. Pour “désactiver” la notation scientifique dans votre session R, définissez-la sur un nombre très élevé, par exemple:
# Désactiver la notation scientifique
options(scipen = 999)Arrondi
DANGER: round() utilise “l’arrondi du banquier” qui arrondit à partir de 0,5 uniquement si le nombre supérieur est pair. Utilisez round_half_up() de janitor pour arrondir systématiquement les moitiés au nombre entier le plus proche. Voir cette explication
# Fonction d'arrondi avec R de base:
round(c(2.5, 3.5))[1] 2 4# Fonction d'arrondi du paquet "janitor":
janitor::round_half_up(c(2.5, 3.5))[1] 3 4Fonctions statistiques
ATTENTION: Les fonctions ci-dessous incluront par défaut les valeurs manquantes dans les calculs. Les valeurs manquantes entraîneront une sortie de NA, sauf si l’argument na.rm = TRUE est spécifié. Cela peut être écrit en raccourci comme na.rm = T.
| Objective | Fonction |
|---|---|
| moyen | mean(x, na.rm=T) |
| médian | median(x, na.rm=T) |
| écart-type | sd(x, na.rm=T) |
| quantiles | quantile(x, probs) |
| somme | sum(x, na.rm=T) |
| valeur minimum | min(x, na.rm=T) |
| valeur maximum | max(x, na.rm=T) |
| plage de valeurs numériques | range(x, na.rm=T) |
| sommaire | summary(x) |
Remarques:
-
*quantile():xest le vecteur numérique à examiner etprobs =est un vecteur numérique avec des probabilités comprises entre 0 et 1,0, par exemplec(0,5, 0,8, 0,85) -
**summary(): donne un résumé sur un vecteur numérique comprenant la moyenne, la médiane et les centiles communs
DANGER: Si vous fournissez un vecteur de nombres à l’une des fonctions ci-dessus, assurez-vous d’envelopper les nombres dans c().
# Si vous fournissez des nombres bruts à une fonction,
# enveloppez-les dans c():
# !!! ERREUR !!!
mean(1, 6, 12, 10, 5, 0) [1] 1# CORRECT
mean(c(1, 6, 12, 10, 5, 0)) [1] 5.666667Autres fonctions utiles
| Objectif | Fonction | Exemple |
|---|---|---|
| créer une séquence | seq(from, to, by) | seq(1, 10, 2) |
| répéter x, n fois | rep(x, ntimes) |
rep(1:3, 2) or rep(c("a", "b", "c"), 3)
|
| subdiviser un vecteur numérique | cut(x, n) | cut(linelist$age, 5) |
| prendre un échantillon au hasard | sample(x, size) | sample(linelist$i d , size = 5, replace = TRUE) |
%in%
Un opérateur très utile pour faire correspondre les valeurs et pour évaluer rapidement si une valeur se trouve dans un vecteur ou une trame de données:
mon_vecteur <- c("a", "b", "c", "d")"a" %in% mon_vecteur[1] TRUE"h" %in% mon_vecteur[1] FALSEPour demander si une valeur n’est pas %in% un vecteur, placez un point d’exclamation (!) devant l’instruction logique:
# Pour nier, mettre une exclamation devant:
!"a" %in% mon_vecteur[1] FALSE!"h" %in% mon_vecteur[1] TRUE%in% est très utile lors de l’utilisation de la fonction dplyr case_when(). Vous pouvez définir un vecteur précédemment, puis le référencer ultérieurement. Par exemple:
affirmative <- c("1", "Yes", "YES", "yes", "y", "Y", "oui", "Oui", "Si")
linelist <- linelist %>%
mutate(enfant_hospitalise = case_when(
hospitalise %in% affirmative & age < 18 ~ "Hospitalized Child",
TRUE ~ "Not"))Remarque: Si vous souhaitez détecter une chaîne partielle, en utilisant peut-être str_detect() de stringr, il n’acceptera pas un vecteur de caractères tel que c("1", "Oui", "oui", "y "). Au lieu de cela, il doit recevoir une expression régulière - une chaîne condensée avec des barres OU, telle que “1|Oui|oui|y”. Par exemple, str_detect(hospitalisé, "1|Oui|oui|y"). Voir la page sur les caractères et les chaîne de caractères pour plus d’informations.
Vous pouvez convertir un vecteur de caractères en une expression régulière nommée avec cette commande:
affirmative <- c("1", "Yes", "YES", "yes", "y", "Y", "oui", "Oui", "Si")
affirmative[1] "1" "Yes" "YES" "yes" "y" "Y" "oui" "Oui" "Si" # Condenser à:
affirmative_str_search <- paste0(affirmative, collapse = "|") # option avec R de base
affirmative_str_search <- str_c(affirmative, collapse = "|") # option avec le paquet stringr
affirmative_str_search[1] "1|Yes|YES|yes|y|Y|oui|Oui|Si"3.12 Erreurs et avertissements
Cette section explique :
- La différence entre les erreurs et les avertissements
- Conseils généraux de syntaxe pour l’écriture de code R
- Aides au code
Les erreurs et avertissements courants ainsi que des conseils de dépannage sont disponibles sur la page erreurs frequentes.
Erreur contre avertissement
Lorsqu’une commande est exécutée, la console R peut afficher des messages d’avertissement ou d’erreur en texte rouge.
Un avertissement signifie que R a terminé votre commande, mais a dû prendre des mesures supplémentaires ou a produit une sortie inhabituelle dont vous devez être conscient.
Une erreur signifie que R n’a pas pu terminer votre commande.
Cherchez des indices:
Le message d’erreur/d’avertissement inclura souvent un numéro de ligne pour le problème.
Si un objet “est inconnu” ou “introuvable”, vous l’avez peut-être mal orthographié, vous avez oublié d’appeler un package avec library() ou vous avez oublié de relancer votre script après avoir apporté des modifications.
Si tout le reste échoue, copiez le message d’erreur dans Google avec quelques termes clés - il y a de fortes chances que quelqu’un d’autre ait déjà travaillé dessus!
Conseils généraux sur la syntaxe
Quelques points à retenir lors de l’écriture de commandes dans R, pour éviter les erreurs et les avertissements:
- Fermez toujours les parenthèses - astuce: comptez le nombre de “(” et de parenthèses fermantes “)” pour chaque bloc de code
- Évitez les espaces dans les noms de colonnes et d’objets. Utilisez le trait de soulignement ( _ ) ou les points ( . ) à la place
- Gardez une trace et n’oubliez pas de séparer les arguments d’une fonction par des virgules
- R est sensible à la casse, ce qui signifie que
Variable_Aest différent deVariable_a
Aides au code
N’importe quel script (RMarkdown ou autre) donnera des indices lorsque vous avez fait une erreur. Par exemple, si vous avez oublié d’écrire une virgule là où c’est nécessaire, ou de fermer une parenthèse, RStudio lèvera un drapeau sur cette ligne, sur le côté droit du script, pour vous avertir.



Comment installer et charger
Dans ce manuel, nous suggérons d’utiliser le paquet pacman (abréviation de “package manager” en anglais). Il offre une fonction pratique
p_load()qui installera un paquet si nécessaire et le chargera pour l’utiliser dans la séance R actuelle.La syntaxe est assez simple. Il suffit de lister les noms des paquets entre les parenthèses de
p_load(), séparés par des virgules.La commande ci-dessous installera les paquets rio, tidyverse, et here s’ils ne sont pas encore installés, et les chargera pour les utiliser. Cela rend l’approche
p_load()pratique et concise si vous partagez des scripts avec d’autres personnes. Notez que les noms des paquets sont sensibles à la casse.Notez que nous avons utilisé la syntaxe
pacman::p_load()qui écrit explicitement le nom du paquet (pacman) avant le nom de la fonction (p_load()), reliés par deux deux points::. Cette syntaxe est utile car elle charge également le paquet pacman (en supposant qu’il soit déjà installé).Il existe d’autres fonctions R de base que vous verrez souvent. La fonction R de base pour installer un paquet est
install.packages(). Le nom du paquet à installer doit être fourni entre les parenthèses et entre guillemets. Si vous voulez installer plusieurs paquets en une seule commande, ils doivent être listés dans un vecteur de caractèresc().Remarque : cette commande installe un paquet, mais ne le charge pas pour l’utiliser dans la séance en cours.
L’installation peut également être effectuée par pointer-cliquer en allant dans le panneau “Packages” de RStudio, en cliquant sur “Installer” et en recherchant le nom du paquet souhaité.
La fonction base de R pour charger un paquet à utiliser (après qu’il ait été installé) est
library(). Elle ne peut charger qu’un seul paquet à la fois (une autre raison d’utiliserp_load()). Vous pouvez fournir le nom du paquet avec ou sans guillemets.Pour vérifier si un paquet est installé et/ou chargé, vous pouvez afficher le panneau des paquets dans RStudio. Si le paquet est installé, il est affiché avec son numéro de version. Si sa case est cochée, il est chargé pour la séance en cours.
Installation depuis Github
Parfois, vous avez besoin d’installer un paquet qui n’est pas encore disponible sur CRAN. Ou peut-être que le paquet est disponible sur CRAN mais que vous voulez la version de développement avec de nouvelles fonctionnalités qui ne sont pas encore proposées dans la version CRAN publiée, plus stable. Ces versions sont souvent hébergées sur le site Web github.com dans un “dépôt” de code libre et public. Pour en savoir plus sur Github, consultez la page du manuel intitulée Version control et collaboration avec GitHub.
Pour télécharger des paquets R depuis Github, vous pouvez utiliser la fonction
p_load_gh()de pacman, qui installera le paquet si nécessaire, et le chargera pour l’utiliser dans votre séance R actuelle. Les alternatives à l’installation incluent l’utilisation des paquets remotes ou devtools. Pour en savoir plus sur toutes les fonctions de pacman, consultez la documentation du paquet.Pour installer à partir de Github, vous devez fournir plus d’informations. Vous devez fournir :
Dans les exemples ci-dessous, le premier mot entre guillemets est l’ID Github du propriétaire du dépôt. Après la barre oblique est le nom du dépôt (typiquement le nom du paquet).
Si vous voulez installer à partir d’une “branche” (version) autre que la branche principale, ajoutez le nom de la branche après un “@”, après le nom du dépôt.
S’il n’y a pas de différence entre la version Github et la version sur votre ordinateur, aucune action ne sera entreprise. Vous pouvez “forcer” une réinstallation en utilisant
p_load_current_gh()avec le paramètreupdate = TRUE. Lisez plus sur pacman dans cette vignette en ligneInstallation à partir d’un ZIP ou d’un TAR
Vous pouvez installer le paquet à partir d’une URL :
Ou bien, le télécharger sur votre ordinateur dans un fichier zippé :
Option 1 : utiliser
install_local()du paquet remotes.Option 2 : en utilisant
install.packages()du R de base, en fournissant le chemin d’accès au fichier ZIP et en définissanttype = "source"etrepos = NULL.