36 Analyse des combinaisons
Cette analyse représente la fréquence des différentes combinaisons de valeurs/réponses. Dans cet exemple, nous traçons la fréquence à laquelle les cas ont présenté diverses combinaisons de symptômes.
Cette analyse est aussi souvent appelée :
-
“Analyse des réponses multiples”
-
“Analyse des ensembles”
- “Analyse des combinaisons”
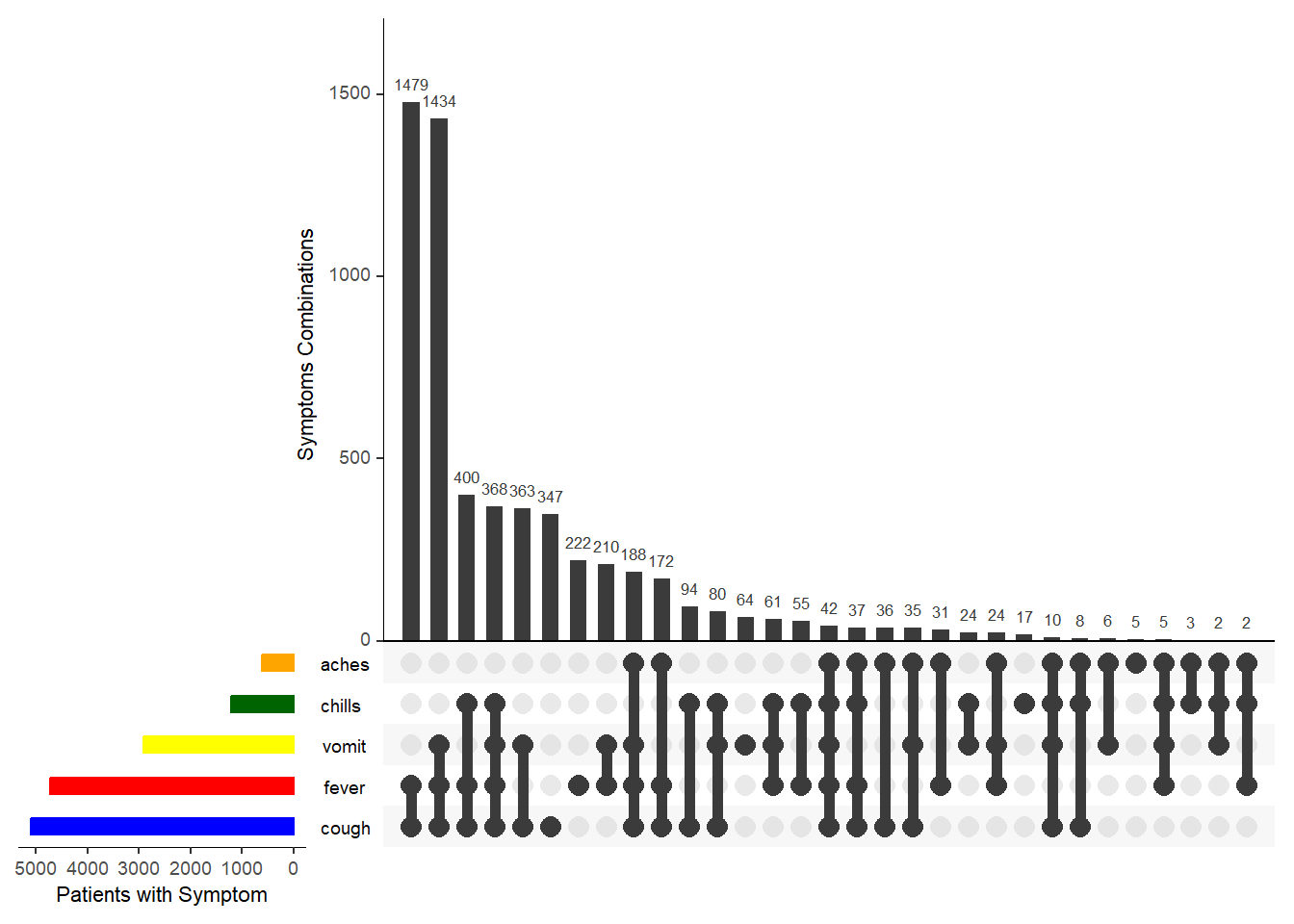
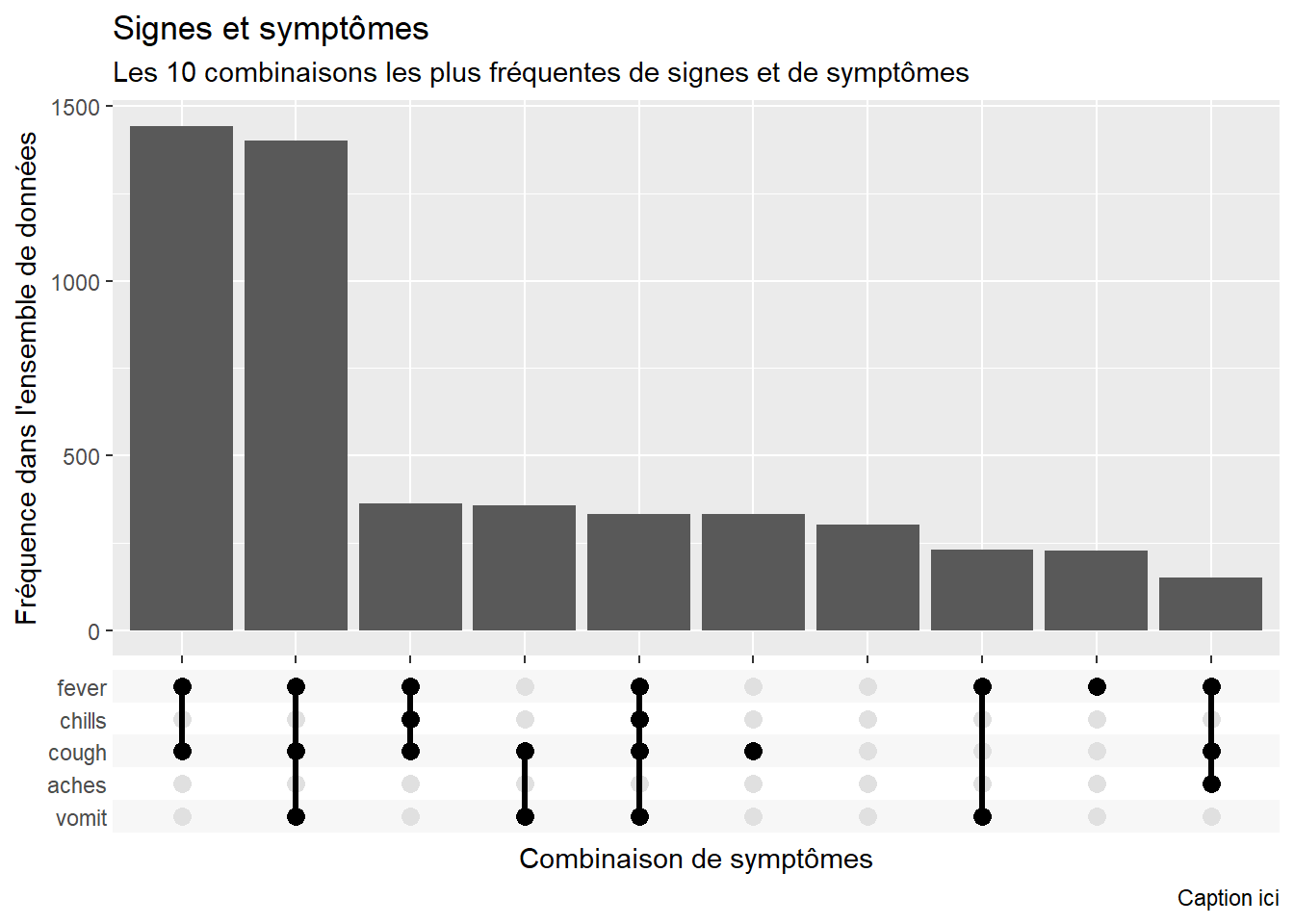
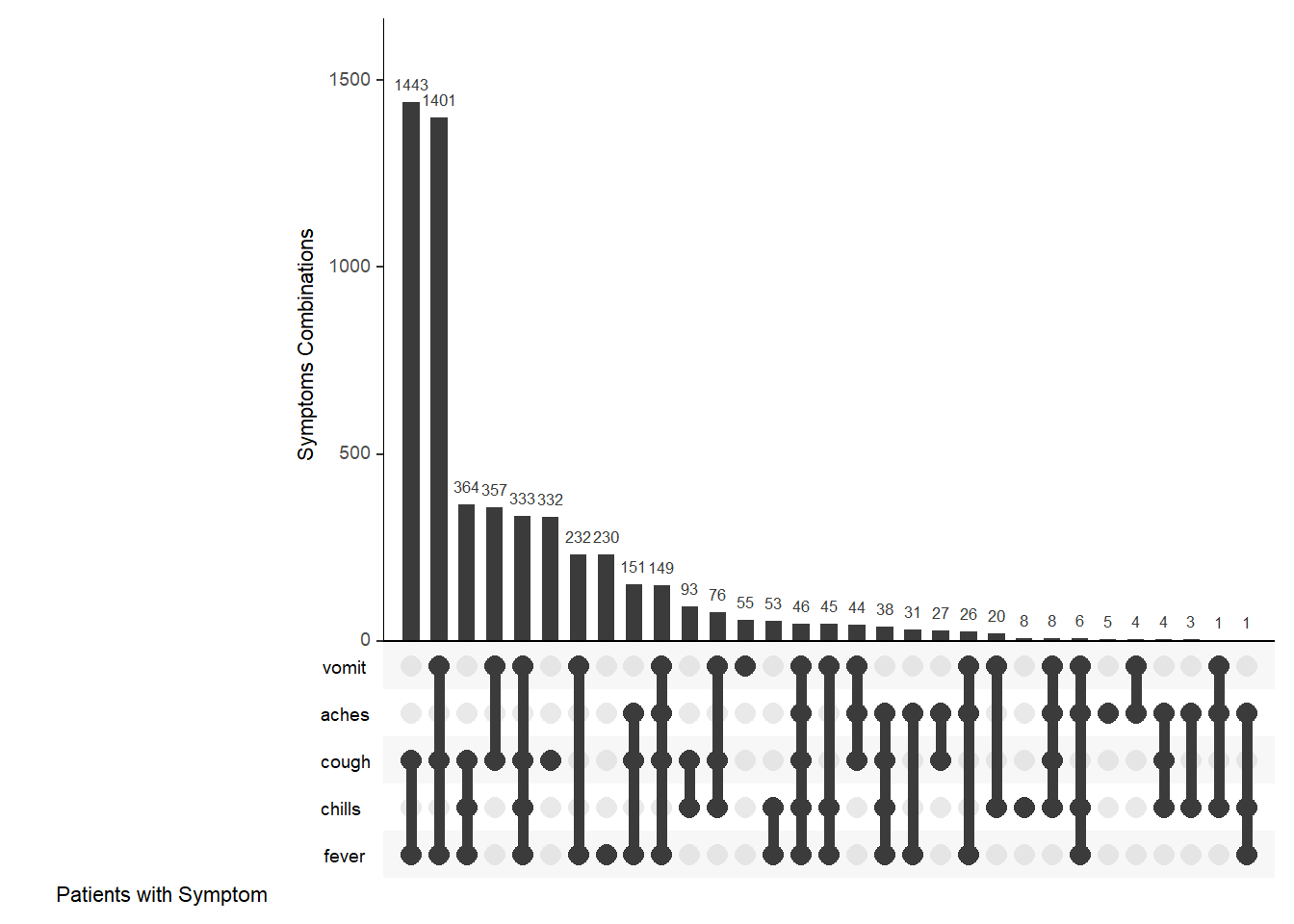
Dans l’exemple de graphique ci-dessus, cinq symptômes sont représentés. Sous chaque barre verticale se trouve une ligne et des points indiquant la combinaison de symptômes reflétée par la barre ci-dessus. À droite, des barres horizontales reflètent la fréquence de chaque symptôme individuel.
La première méthode que nous montrons utilise le paquet ggupset, et la seconde utilise le paquet UpSetR.
36.1 Préparation
Chargement des paquets
Ce chunk de code montre le chargement des paquets nécessaires aux analyses. Dans ce manuel, nous mettons l’accent sur p_load() de pacman, qui installe le paquet si nécessaire et le charge pour l’utiliser. Vous pouvez également charger les paquets installés avec library() de base R. Voir la page sur R - les bases pour plus d’informations sur les paquets R.
pacman::p_load(
tidyverse, # gestion et visualisation de données
UpSetR, # paquet spécial pour les graphiques combinés
ggupset) # paquet spécial pour les tracés combinésImporter les données
Pour commencer, nous importons la linelist nettoyée des cas d’une épidémie d’Ebola simulée. Si vous voulez suivre, cliquez pour télécharger la linelist “propre” (en tant que fichier .rds). Importez des données avec la fonction import() du paquet rio (elle gère de nombreux types de fichiers comme .xlsx, .csv, .rds - voir la page Importer et exporter des données pour plus de détails).
# importer la liste de cas linelist
linelist_sym <- import("linelist_cleaned.rds")Cette linelist comprend cinq variables “oui/non” sur les symptômes déclarés. Nous devrons transformer un peu ces variables pour utiliser le paquet ggupset afin de réaliser notre tracé. Visualisez les données (faites défiler vers la droite pour voir les variables de symptômes).
Re-formatage des valeurs
Pour s’aligner sur le format attendu par ggupset, nous convertissons les “yes” (“oui”) et “no” (“non”) en nom de symptôme réel, en utilisant case_when() de dplyr. Si “non”, nous mettons la valeur en blanc, donc les valeurs sont soit NA ou le symptôme.
# crée une colonne avec les symptômes nommés, séparés par des points-virgules
linelist_sym_1 <- linelist_sym %>%
# convertissez les valeurs "oui" et "non" dans le nom du symptôme lui-même
mutate(fever = ifelse(fever == "yes", "fever", NA),
chills = ifelse(chills == "yes", "chills", NA),
cough = ifelse(cough == "yes", "cough", NA),
aches = ifelse(aches == "yes", "aches", NA),
vomit = ifelse(vomit == "yes", "vomit", NA))Maintenant, nous faisons deux dernières colonnes :
- Concaténation (collage) de tous les symptômes du patient (une colonne de caractères)
- Convertir la colonne ci-dessus en classe list, afin qu’elle puisse être acceptée par ggupset pour faire le graphe.
Voir la page sur Caractères et chaînes de caractères pour en savoir plus sur la fonction unite() de stringr.
linelist_sym_1 <- linelist_sym_1 %>%
unite(col = "all_symptoms",
c(fever, chills, cough, aches, vomit),
sep = " ; ",
remove = TRUE,
na.rm = TRUE) %>%
mutate(
# Faites une copie de la colonne all_symptoms, mais de la classe "list" (qui est nécessaire pour utiliser ggupset() à l'étape suivante).
all_symptoms_list = as.list(strsplit(all_symptoms, " ; "))
)Visualisez les nouvelles données. Notez les deux colonnes vers l’extrémité droite - les valeurs combinées collées, et la liste
36.2 ggupset
Charger le paquet
pacman::p_load(ggupset)Créez le graphique. Nous commençons par un ggplot() et un geom_bar(), mais ensuite nous ajoutons la fonction spéciale scale_x_upset() du ggupset.
ggplot(
data = linelist_sym_1,
mapping = aes(x = all_symptoms_list)) +
geom_bar() +
scale_x_upset(
reverse = FALSE,
n_intersections = 10,
sets = c("fever", "chills", "cough", "aches", "vomit"))+
labs(
title = "Signes et symptômes",
subtitle = "Les 10 combinaisons les plus fréquentes de signes et de symptômes",
caption = "Caption ici",
x = "Combinaison de symptômes",
y = "Fréquence dans l'ensemble de données")De plus amples informations sur ggupset peuvent être trouvées en ligne ou hors ligne dans la documentation du paquet dans votre onglet d’aide RStudio ?ggupset.
36.3 UpSetR
Le paquet UpSetR permet de personnaliser davantage le tracé, mais il peut être plus difficile à exécuter :
Chargez le paquet
pacman::p_load(UpSetR)Nettoyage des données
Nous devons convertir les valeurs des symptômes de la linelist en 1 / 0.
linelist_sym_2 <- linelist_sym %>%
# convertissez les valeurs "oui" et "non" dans le nom du symptôme lui-même
mutate(fever = ifelse(fever == "yes", 1, 0),
chills = ifelse(chills == "yes", 1, 0),
cough = ifelse(cough == "yes", 1, 0),
aches = ifelse(aches == "yes", 1, 0),
vomit = ifelse(vomit == "yes", 1, 0))Si vous êtes intéressé par une commande plus efficace, vous pouvez profiter de la fonction +(), qui convertit les 1 et les 0 en fonction d’une déclaration logique. Cette commande utilise la fonction across() pour modifier plusieurs colonnes à la fois (pour en savoir plus, lisez la section Nettoyage des données et des fonctions de base).
# convertissez "oui" et "non" a 1 et 0
linelist_sym_2 <- linelist_sym %>%
mutate(across(c(fever, chills, cough, aches, vomit), .fns = ~+(.x == "yes")))Maintenant, faites le graphique en utilisant la fonction personnalisée upset() - en utilisant seulement les colonnes de symptômes. Vous devez désigner les “ensembles” à comparer (les noms des colonnes de symptômes). Vous pouvez aussi utiliser nsets = et order.by = "freq" pour n’afficher que les X combinaisons les plus importantes.
# Créer le graphique
UpSetR::upset(
select(linelist_sym_2, fever, chills, cough, aches, vomit),
sets = c("fever", "chills", "cough", "aches", "vomit"),
order.by = "freq",
sets.bar.color = c("blue", "red", "yellow", "darkgreen", "orange"), # couleurs optionnelles
empty.intersections = "on",
# nsets = 3,
number.angles = 0,
point.size = 3.5,
line.size = 2,
mainbar.y.label = "Symptoms Combinations",
sets.x.label = "Patients with Symptom")36.4 Ressources
Une version Shiny App - vous pouvez télécharger vos propres données