32 Curvas epidêmicas
Uma curva epidêmica (também conhecida como “epicurva”) é uma ferramenta essencial tipicamente utilizada para visualizar padrões temporais do início de doenças entre um conjunto ou epidemia de casos.
A análise da epicurva pode revelar tendências temporais, anomalias/valores discrepantes (“outliers”), a magnitude de um surto, o período mais provável de exposição, intervalos temporais entre picos de casos, e pode até ajudar a identificar o modo de transmissão de uma doença não identificada (ex.: fonte pontual, fonte comum contínua, propagação pessoa-pessoa). Uma aula online sobre a interpretação de epicurvas pode ser encontrada no website do CDC americano.
Nesta página, nós demonstraremos duas abordagens para a produção de curvas epidêmicas no R:
- O pacote incidence2, que pode produzir uma epicurva com comandos simples
- O pacote ggplot2, que permite customizações avançadas através de comandos mais complexos
Também serão abordados casos de uso específico:
- Criar gráficos de contagem agregada
- Mostrar ou produzir múltiplos-pequenos
- Aplicar médias móveis
- Mostrar quais dados são “preliminares” ou sujeitos a atrasos na notificação
- Sobreposição da incidência acumulada de casos usando um segundo eixo do gráfico
32.1 Preparação
Carregue os pacotes necessários
O código abaixo realiza o carregamento dos pacotes necessários para a análise dos dados. Neste manual, enfatizamos o uso da função p_load(), do pacman, que instala os pacotes, caso não estejam instalados, e os carrega no R para utilização. Também é possível carregar pacotes instalados utilizando a função library(), do R base. Para mais informações sobre os pacotes do R, veja a página Introdução ao R.
pacman::p_load(
rio, # importar/exportar arquivos
here, # caminhos de arquivos relativos
lubridate, # trabalhando com datas/semanas epidemiológicas
aweek, # pacote alternativo para trabalhar com datas/semanas epidemiológicas
incidence2, # epicurvas de dados em uma linelist
i2extras, # suplemento para o pacote incidence2
stringr, # procure e manipule strings de caracteres
forcats, # trabalhando com fators
RColorBrewer, # paleta de cores do colorbrewer2.org
tidyverse # gerenciamento de dados + gráficos no ggplot2
) Importe os dados
Dois exemplos de conjuntos de dados são utilizados nesta seção:
- Lista de linhas (linelist) com casos individuais de uma simulação de epidemia
- Contagens agregadas por hospital da mesma epidemia simulada
Os conjuntos de dados são importados utilizando a função import() do pacote rio. Veja a página sobre Importar e exportar para conhecer as diferentes formas de importar dados.
Lista de linhas de casos
Nós importamos o conjunto de dados de casos de uma simulação de epidemia de Ebola. Se você quiser baixar os dados para acompanhar etapa-por-etapa, veja instruções na página sobre Download do manual e dados. Aqui, nós assumimos que o arquivo está no diretório de trabalho. Logo, nenhuma sub-pasta é especificada no endereço do arquivo.
linelist <- import("linelist_cleaned.xlsx")As primeiras 50 linhas são mostradas abaixo.
Casos agregados por hospital
Para atingir os propósitos deste manual, o conjunto de dados de contagens semanais de casos agregados por hospital é criado a partir da linelist, com o seguinte código.
# cria os dados de contagem no R
count_data <- linelist %>%
group_by(hospital, date_hospitalisation) %>%
summarize(n_cases = dplyr::n()) %>%
filter(date_hospitalisation > as.Date("2013-06-01")) %>%
ungroup()As primeiras 50 linhas são mostradas abaixo:
Ajuste os parâmetros
Para produzir um relatório, você pode querer ajustar parâmetros modificáveis, como a data para a qual os dados são atuais (a “data dos dados”). Você pode, então, referenciar o objeto data_date em seu código ao aplicar filtros ou em legendas dinâmicas.
## ajuste a data do relatório para o relatório
## nota: pode ser ajustada para Sys.Date() para a data atual
data_date <- as.Date("2015-05-15")Verifique as datas
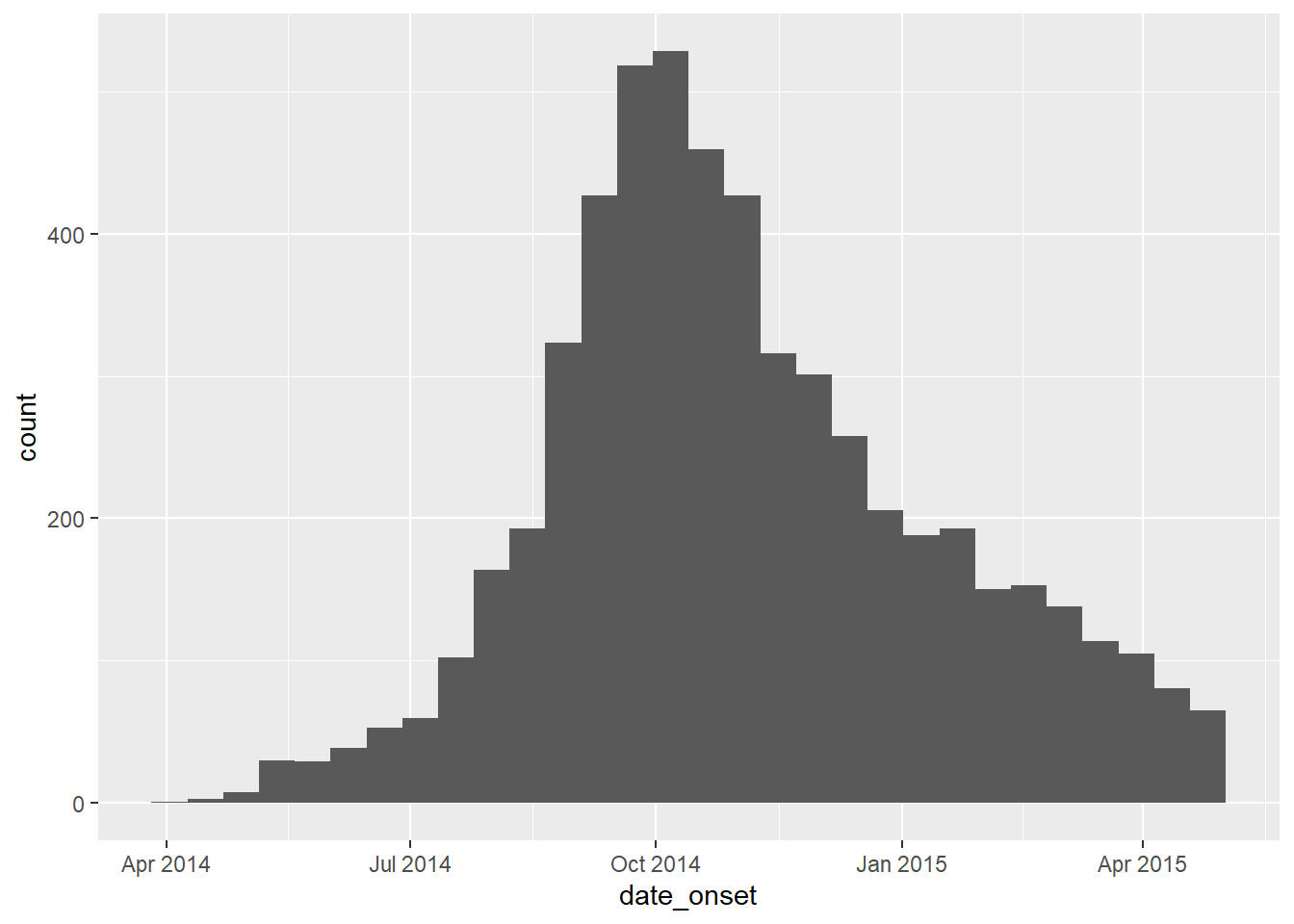
Perceba que cada coluna de datas relevantes é da classe Date e tem um intervalo apropriado de valores. Você pode fazer isso simplesmente utilizando hist(), para histograma, ou range() com na.rm=TRUE, ou com o ggplot(), como mostrado abaixo.
32.2 Epicurvas no ggplot2
Utilizar o ggplot() para criar sua epicurva permite mais flexibilidade e customização. Entretanto, é necessário mais esforço e entendimento sobre como o ggplot() funciona.
Diferente do pacote incidence2, no ggplot você precisa controlar manualmente a agregação dos casos pelo tempo (em semanas, meses, etc) e os intervalos dos rótulos no eixo de data. Isto precisa ser cuidadosamente gerenciado.
Os exemplos abaixo utilizam um subconjunto do banco de dados linelist - apenas os casos do Hospital Central.
central_data <- linelist %>%
filter(hospital == "Central Hospital")Para produzir uma epicurva com o ggplot(), existem três elementos principais:
- Um histograma, com os casos da linelist agregados em “classes” (Nota do tradutor: tradução livre de ´bins´, que em inglês significa caixa, ou container) separados por pontos “de quebra” específicos
- Escalas para os eixos e seus rótulos
- Temas para a aparência do gráfico, incluindo títulos, rótulos, legendas, etc.
Especifique as classes de casos
Aqui nós mostramos como especificar a maneira em que os casos serão agregados nas classes do histogramas (“barras”). É importante reconhecer que o agrupamento dos casos nessas classes do histograma não será necessariamente nos mesmos intervalos das datas que irão aparecer no eixo x.
A seguir é mostrado, provavelmente, o código mais simples para produzir epicurvas diárias ou semanais.
No comando global ggplot(), o conjunto de dados é fornecido em data =. A partir disto, a geometria do histrograma é adicionada com um +. Dentro de geom_histogram(), nós mapeamos a aparência do gráfico de forma que a coluna date_onset é mapeada para o eixo x. Também dentro de geom_histogram(), mas não dentro de aes(), nós ajustamos o binwidth = dos containers do histobrama, em dias. Se essa sintaxe do ggplot2 é confusa, revise a página sobre básico do ggplot.
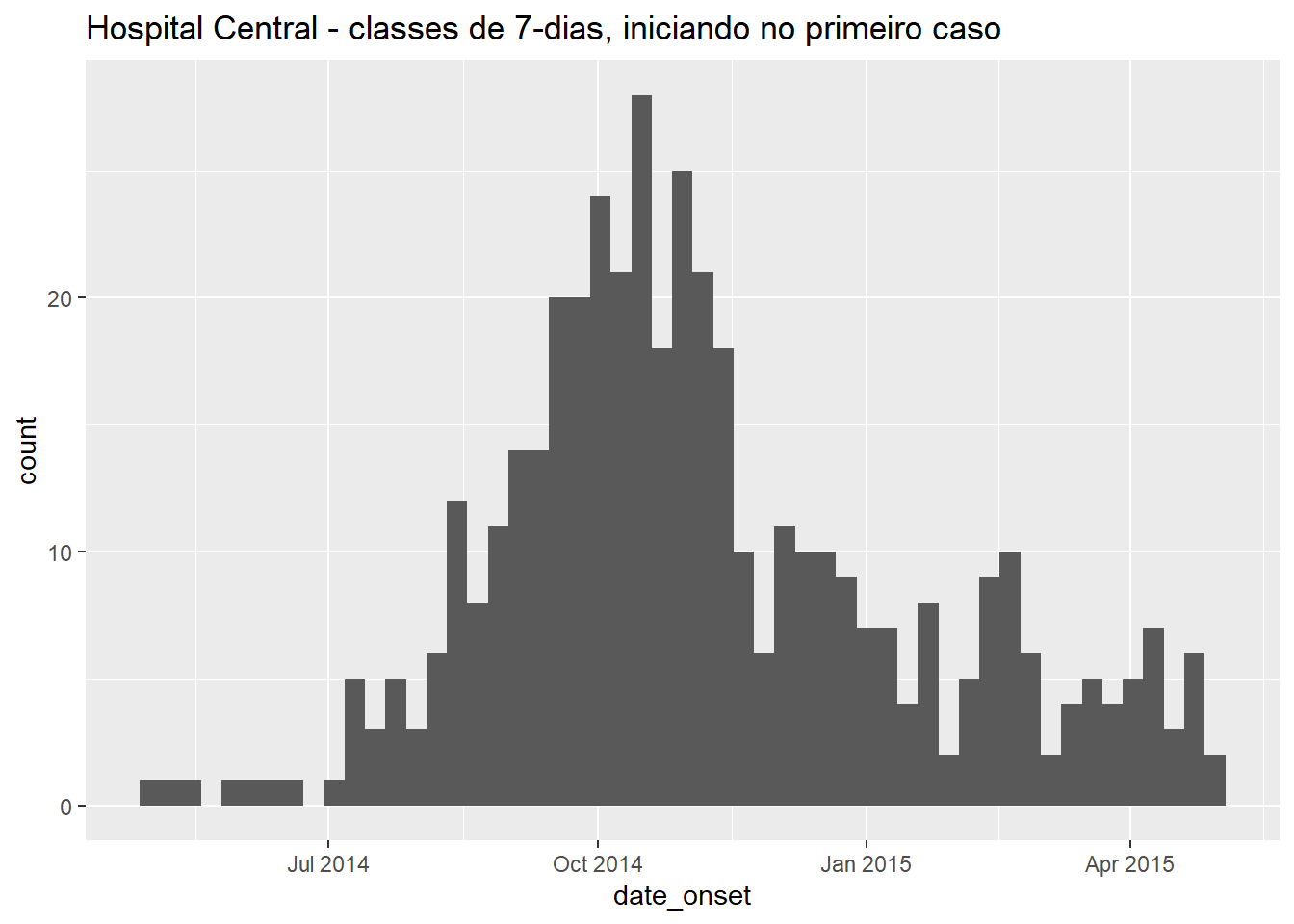
CUIDADO: Criar um gráfico de casos semanas ao utilizar binwidth = 7 inicia o primeiro container de 7-dias no primeiro caso, que pode ser em qualquer dia da semana! Para criar semanas específicas, veja a seção abaixo.
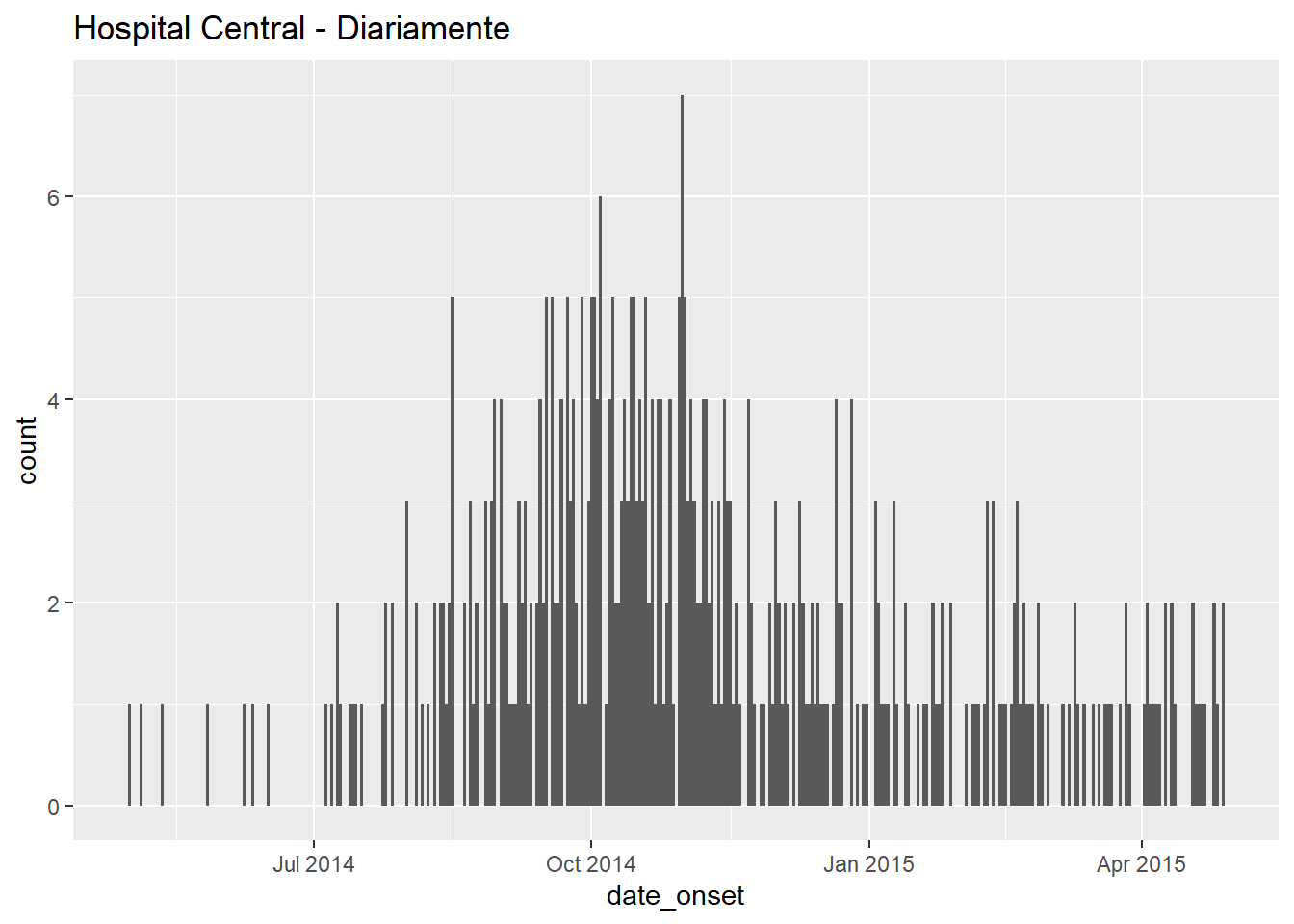
# diariamente
ggplot(data = central_data) + # escolha os dados
geom_histogram( # adicione o histograma
mapping = aes(x = date_onset), # mapeia a coluna de data para o eixo x
binwidth = 1)+ # casos unidos por 1 dia
labs(title = "Hospital Central - Diariamente") # título
# semanal
ggplot(data = central_data) + # escolha os dados
geom_histogram( # adicione o histograma
mapping = aes(x = date_onset), # mapeia a coluna de data para o eixo x
binwidth = 7)+ # casos unidos a cada 7 dias, iniciando no primeiro caso (!)
labs(title = "Hospital Central - classes de 7-dias, iniciando no primeiro caso") # títuloDeixe-nos observar que o primeiro caso neste conjunto de dados do Hospital Central teve o início dos sintomas em:
format(min(central_data$date_onset, na.rm=T), "%A %d %b, %Y")[1] "Thursday 01 May, 2014"Para especificar manualmente as quebras das classes do histograma, não utilize o argumento binwidth =, e, em vez disso, forneça um vetor de datas para breaks =.
Crie o vetor de datas com a função seq.Date() do R base. Esta função precisa dos argumentos to =, from =, e by =. Por exemplo, o comando abaixo retorna datas mensais começando em 15 de janeiro e terminando em 28 de junho.
monthly_breaks <- seq.Date(from = as.Date("2014-02-01"),
to = as.Date("2015-07-15"),
by = "months")
monthly_breaks # exporta para o console [1] "2014-02-01" "2014-03-01" "2014-04-01" "2014-05-01" "2014-06-01"
[6] "2014-07-01" "2014-08-01" "2014-09-01" "2014-10-01" "2014-11-01"
[11] "2014-12-01" "2015-01-01" "2015-02-01" "2015-03-01" "2015-04-01"
[16] "2015-05-01" "2015-06-01" "2015-07-01"Este vetor pode ser fornecido para geom_histogram() como breaks =:
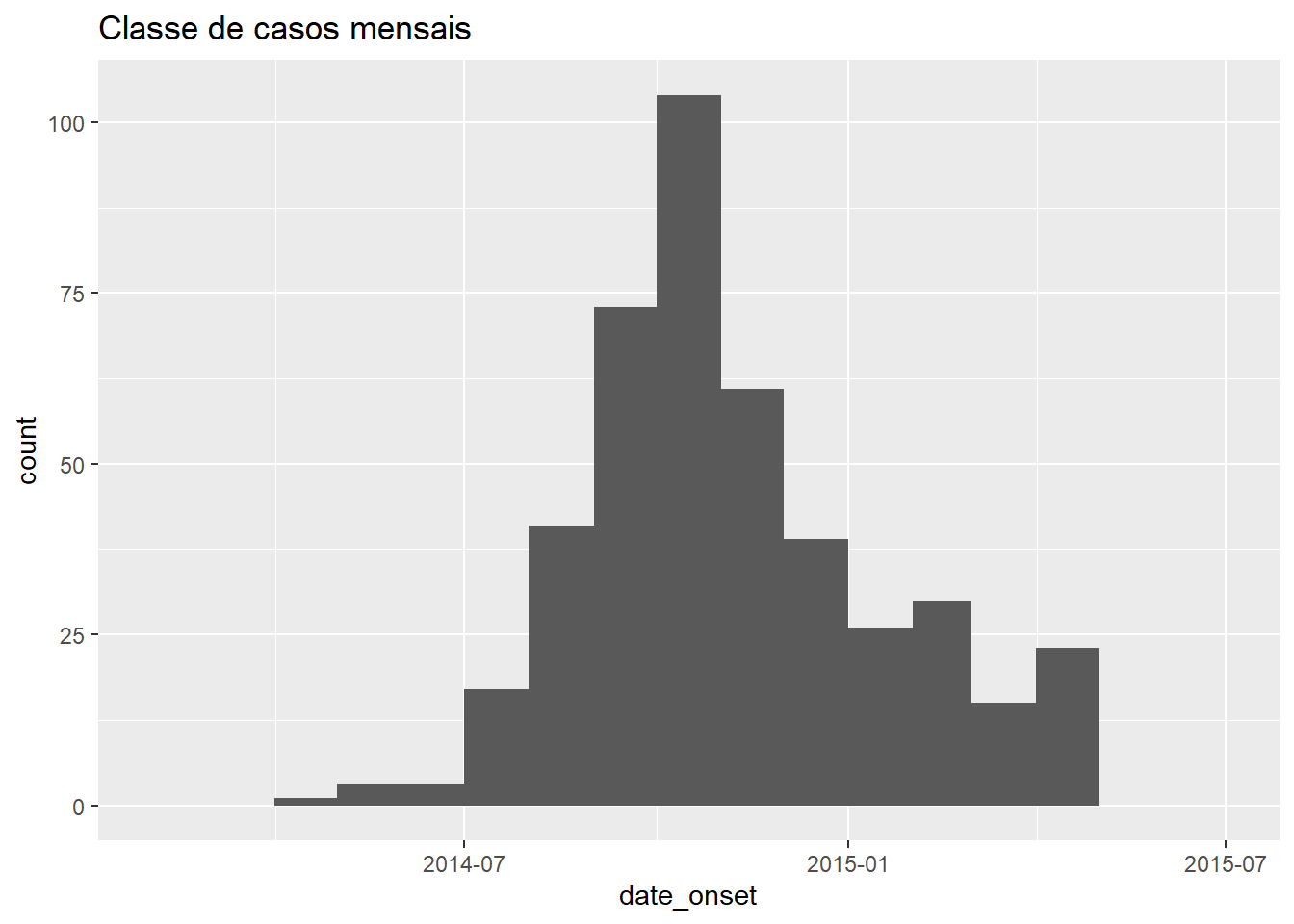
# mensalmente
ggplot(data = central_data) +
geom_histogram(
mapping = aes(x = date_onset),
breaks = monthly_breaks)+ # forneça um vetor pré-definido de quebras
labs(title = "Classe de casos mensais") # títuloUma sequência semanal simples pode ser obtida ao ajustar by = "week". Por exemplo:
weekly_breaks <- seq.Date(from = as.Date("2014-02-01"),
to = as.Date("2015-07-15"),
by = "week")Uma alternativa para o fornecimento de datas específicas de início e fim, é escrever um código dinâmico de forma que classes semanais iniciem na segunda antes do primeiro caso. Nós iremos utilizar estes vetores de data nos exemplos abaixo.
# Sequência de datas semanais iniciando nas Segundas para o HOSPITAL CENTRAL
weekly_breaks_central <- seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 1), # segunda anterior
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 1), # segunda após o último caso
by = "week")Vamos destrinchar o código complicado mostrado acima:
- O valor “from” (data mais antiga da sequência) é criado da seguinte forma: a menor data (
min()comna.rm=TRUE) na colunadate_onseté atribuída afloor_date(), do pacote lubridate.floor_date()é ajustada para gerar “semanas” a partir da data de início dos casos da “semana” em questão, considerando que o dia de início de cada semana é segunda (week_start = 1).
- Da mesma forma, o valor “to” (data final da sequência) é criado utilizando a função inversa
ceiling_date()para retornar a segunda após o último caso. - O argumento “by” de
seq.Date()pode ser ajustado para qualquer número de dias, semanas, ou meses. - Utilize
week_start = 7para semanas que iniciem no Domingo
Como iremos utilizar estes vetores de data nesta página, nós também definimos um vetor para o surto inteiro (o vetor acima é apenas para os casos do Hospital Central).
# Sequência para o surto inteiro
weekly_breaks_all <- seq.Date(
from = floor_date(min(linelist$date_onset, na.rm=T), "week", week_start = 1), # segunda antes do primeiro caso
to = ceiling_date(max(linelist$date_onset, na.rm=T), "week", week_start = 1), # segunda após o último caso
by = "week")Estas saídas do seq.Date() podem ser usadas para criar quebras de classes do histogramas, assim como as quebras para os rótulos de data, que podem ser independentes das primeiras. Leia mais sobre os rótulos de datas em seções posteriores.
DICA: Para um comando ggplot() mais simples, salve as quebras das classes e as quebras dos rótulos de datas como vetores nomeados no começo, e simplesmente os forneça em breaks =.
Exemplo de epicurva semanal
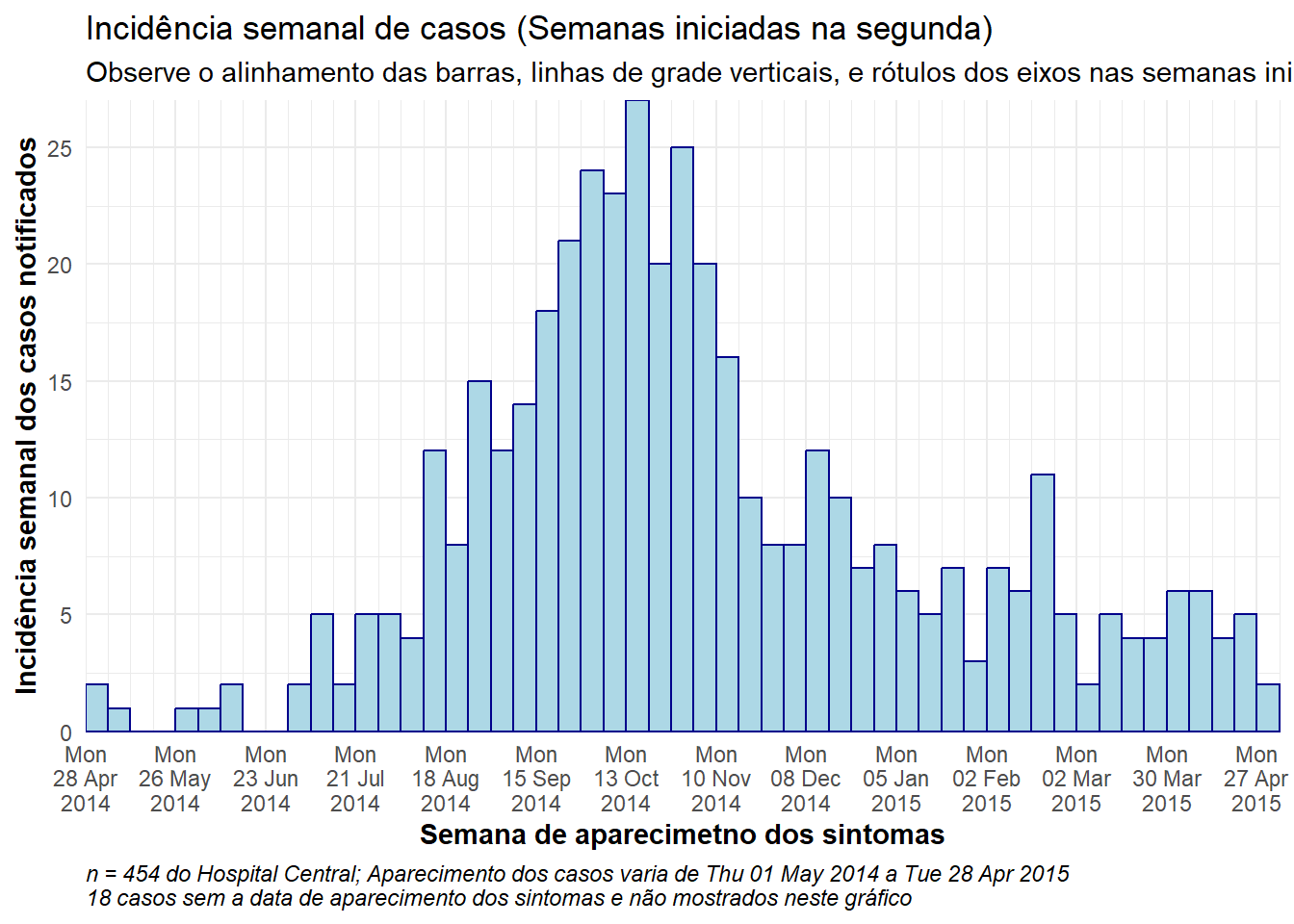
Abaixo esta detalhado um exemplo de código utilizado para produzir epicurvas semanais para semanas que iniciam nas segundas, com barras alinhadas, rótulos de data, e linhas de grade verticais. Esta seção é para o usuário que precisa rapidamente de um código. Para entender cada aspecto (temas, rótulos de datas, etc.) profundamente, continue para as seções subsequentes. De nota:
- As quebras dos containers do histograma são definidas com
seq.Date(), como explicado acima, para iniciar na segunda anterior ao primeiro caso, e para terminar na segunda posterior ao último caso - O intervalo dos rótulos de data é especificado por
date_breaks =dentro descale_x_date()
- O intervalo de linhas da grade vertical menores entre os rótulos de data é especificado em
date_minor_breaks =
-
expand = c(0,0)nas escalas de x e y remove o excesso de espaço em cada lado dos eixos, o que também garante que os rótulos de data iniciem a partir da primeira barra.
# ALINHAMENTO TOTAL A PARTIR DAS SEGUNDAS-FEIRAS
#############################
# Defina a sequência de quebras semanais
weekly_breaks_central <- seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 1), # Segunda antes do primeiro caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 1), # Segunda após último caso
by = "week") # containers são de 7-dias
ggplot(data = central_data) +
# crie o histograma: especifique os pontos de quebra do container: inicie na segunda antes do primeiro caso, termine na segunda após o último caso
geom_histogram(
# mapeando a estética do gráfico
mapping = aes(x = date_onset), # coluna de data mapeada para o eixo x
# quebras da classe do histograma
breaks = weekly_breaks_central, # quebras do container do histograma definidas anteriormente
# barras
color = "darkblue", # cor das linhas ao redor das barras
fill = "lightblue" # cor do preenchimento das barras
)+
# rótulos do eixo x
scale_x_date(
expand = c(0,0), # remove o espaço em excesso do eixo x antes e após as barras de casos
date_breaks = "4 weeks", # rótulos de data e principais linhas de grade verticais aparecem a cada 3 semanas iniciando nas segundas
date_minor_breaks = "week", # linhas de grade menores aparecem a cada semana iniciando na segunda
date_labels = "%a\n%d %b\n%Y")+ # formato dos rótulos de data
# eixo y
scale_y_continuous(
expand = c(0,0))+ # remove o excesso de espaço do eixo y abaixo de 0 (alinha o histograma nivelado com o eixo x)
# temas estéticos
theme_minimal()+ # simplifique o fundo do gráfico
theme(
plot.caption = element_text(hjust = 0, # legenda no lado esquerdo
face = "italic"), # legenda em itálico
axis.title = element_text(face = "bold"))+ # título dos eixos em negrito
# rótulos incluindo legendas dinâmicas
labs(
title = "Incidência semanal de casos (Semanas iniciadas na segunda)",
subtitle = "Observe o alinhamento das barras, linhas de grade verticais, e rótulos dos eixos nas semanas iniciadas na segunda",
x = "Semana de aparecimetno dos sintomas",
y = "Incidência semanal dos casos notificados",
caption = stringr::str_glue("n = {nrow(central_data)} do Hospital Central; Aparecimento dos casos varia de {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} a {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} casos sem a data de aparecimento dos sintomas e não mostrados neste gráfico"))Semanas iniciando no domingo
Para obter o gráfico acima para semanas que iniciam no domingo, algumas poucas modificações são necessárias, uma vez que o date_breaks = "weeks" funciona apenas para semanas iniciando às segundas.
- Os pontos de quebra das classes do histograma precisam serem ajustados para os domingos (
week_start = 7)
- Dentro de
scale_x_date(), as datas de quebra similares devem ser fornecidas parabreaks =eminor_breaks =, visando garantir que os rótulos de data e linhas de grade verticais alinhem nos domingos.
Por exemplo, o comando scale_x_date(), para semanas iniciando nos domingos, pode ser semelhante ao seguinte:
scale_x_date(
expand = c(0,0),
# especifique o intervalo do rótulo de datas e das linhas de grade principais
breaks = seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7), # Domingo antes do primeiro caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7), # Domingo após o último caso
by = "4 weeks"),
# especifique o intervalo das linhas de grade vertical secundárias
minor_breaks = seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7), # Domingo antes do primeiro caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7), # Domingo após o último caso
by = "week"),
# formato do rótulo de data
#date_labels = "%a\n%d %b\n%Y")+ # dia, mês acima abreviado, sobre o ano com 2-dígitos
label = scales::label_date_short())Grupo/cor por valor
As barras do histograma podem ser coloridas por grupo e podem ser “empilhadas” (o inglês stacked). Para designar a coluna de agrupamento, faça as seguintes modificações. Veja a página sobre básico do ggplot para mais detalhes.
- Dentro do mapeamento estético do histograma,
aes(), mapeie o nome da coluna para os argumentosgroup =efill =
- Remova qualquer argumento de
fill =fora deaes(), uma vez que irá sobrepor-se ao argumentos que estão dentro - Argumentos dentro de
aes()serão aplicados por grupo, enquanto qualquer argumento fora será aplicado para todas as barras (ex.: você pode querercolor =fora, de forma que cada barra tenha a mesma borda)
Aqui é mostrado como o comando aes() pode ser utilizado para agrupar e colorir as barras de acordo com o gênero:
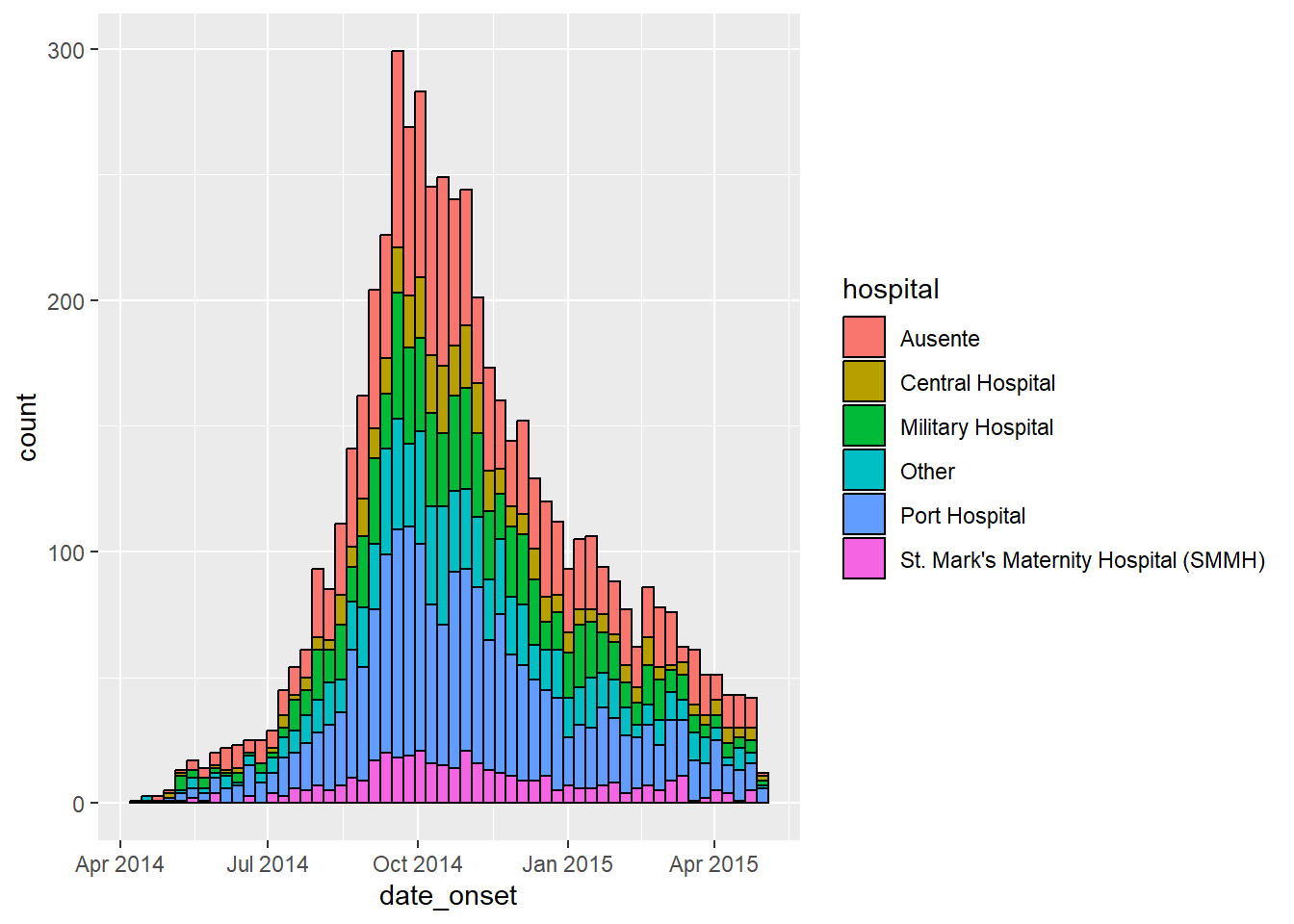
aes(x = date_onset, group = gender, fill = gender)A seguir, ele sendo aplicado:
ggplot(data = linelist) + # comece com o linelist (muitos hospitais)
# crie o histograma: especifique os pontos de ruptura dos containers: começe na segunda anterior ao primeiro caso, e finalize na segunda após o último caso
geom_histogram(
mapping = aes(
x = date_onset,
group = hospital, # ajuste os dados para serem agrupados por hospital
fill = hospital), # preenchimento da barra (cor de dentro) de acordo com o hospital
# quebras dos contentores são nas semanas iniciadas nas segundas
breaks = weekly_breaks_all, # sequência de quebras semanais iniciadas nas segundas para o surto inteiro, como definido em código anterior
# Cor ao redor das barras
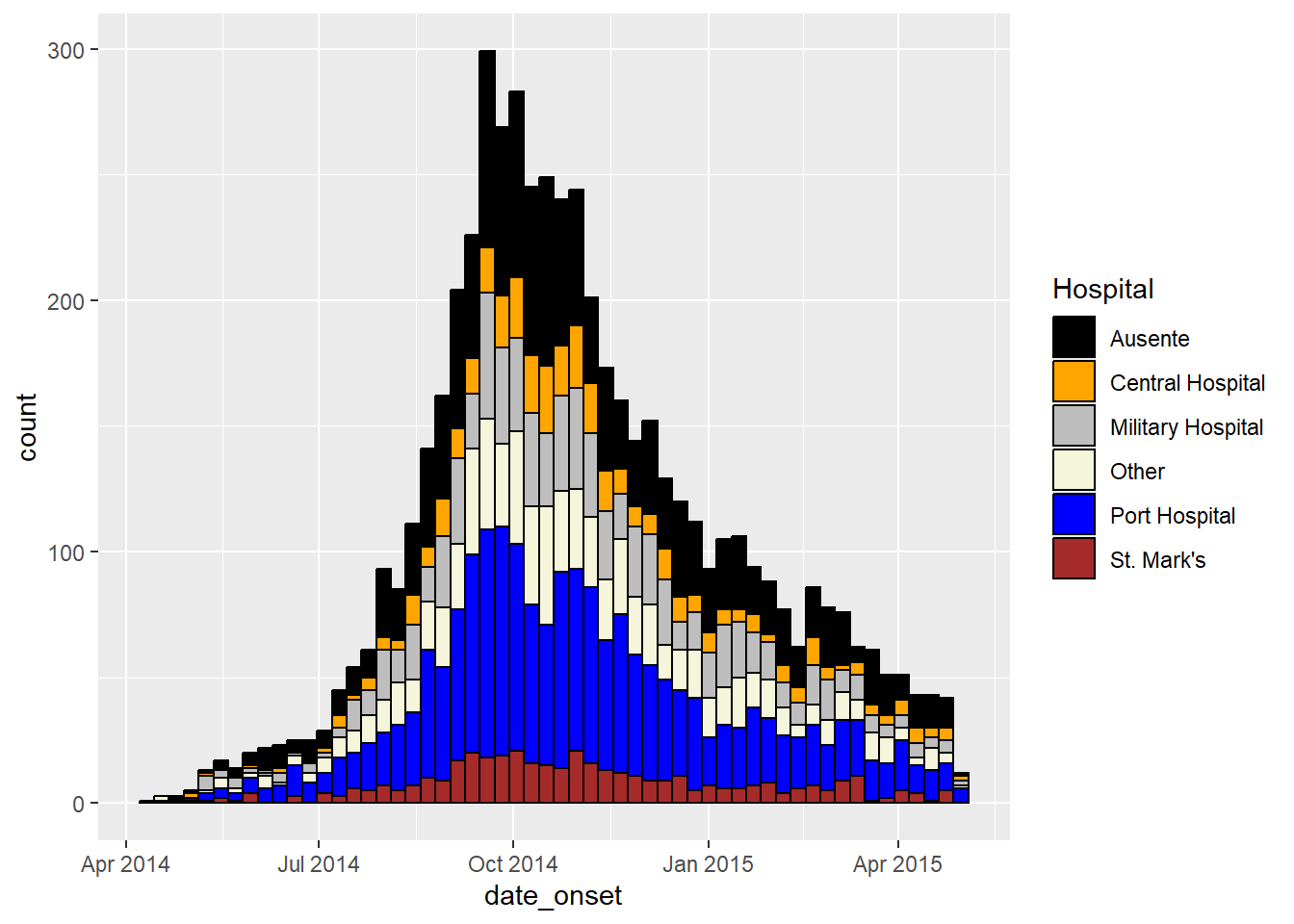
color = "black")Ajuste as cores
- Para manualmente ajustar o preenchimento de cada grupo, utilize
scale_fill_manual()(nota:scale_color_manual()é diferente!).- Use o argumetno
values =para aplicar um vetor de cores. - Use o argumetno
na.value =para especificar uma cor para os valoresNA.
- Use o argumento
labels =para mudar o texto dos itens da legenda. Por segurança, forneça como um vetor nomeado, comoc("old" = "new", "old" = "new")ou ajuste os valores nos próprios dados. - Use
name =para dar um título adequado à legenda
- Use o argumetno
- Para mais dicas sobre escalas de cor e paletas, veja a página sobre básico do ggplot.
ggplot(data = linelist)+ # inicie com a linelist (muitos hospitais)
# crie um histograma
geom_histogram(
mapping = aes(x = date_onset,
group = hospital, # casos agrupados por hospital
fill = hospital), # barras preenchidas por hospital
# quebra dos containers
breaks = weekly_breaks_all, # sequência de quebras semanais iniciadas nas segundas, como definido em código anterior
# Cor ao redor das barras
color = "black")+ # cor da borda para cada barra
# especificação manual das cores
scale_fill_manual(
values = c("black", "orange", "grey", "beige", "blue", "brown"),
labels = c("St. Mark's Maternity Hospital (SMMH)" = "St. Mark's"),
name = "Hospital") # especifique as cores de preenchimento ("values") - atenção à ordem!Ajuste a ordem dos níveis
A ordem em que as barras agrupadas estão empilhadas é melhor ajustada ao se classificar a coluna de agrupamento como sendo da classe fator (Factor). Você poderá, então, designar a ordem dos níveis desse fator (e os rótulos mostrados). Veja a página sobre Fatores ou dicas do ggplot para mais detalhes.
Antes de criar o gráfico, utilize a função fct_relevel() do pacote forcats para converter a coluna de agrupamento para a classe factor, e manualmente ajustar a ordem dos níveis, como detalhado na página sobre Fatores.
# carregue o pacote forcats para trabalhar com fators
pacman::p_load(forcats)
# Defina um novo conjunto de dados com o hospital como fator
plot_data <- linelist %>%
mutate(hospital = fct_relevel(hospital, c("Missing", "Other"))) # Converta para fator e ajuste "Missing" e "Other" como níveis do topo para aparecerem no topo da epicurvaWarning: There was 1 warning in `mutate()`.
ℹ In argument: `hospital = fct_relevel(hospital, c("Missing", "Other"))`.
Caused by warning:
! 1 unknown level in `f`: Missinglevels(plot_data$hospital) # gere os níveis em ordem[1] "Other"
[2] "Ausente"
[3] "Central Hospital"
[4] "Military Hospital"
[5] "Port Hospital"
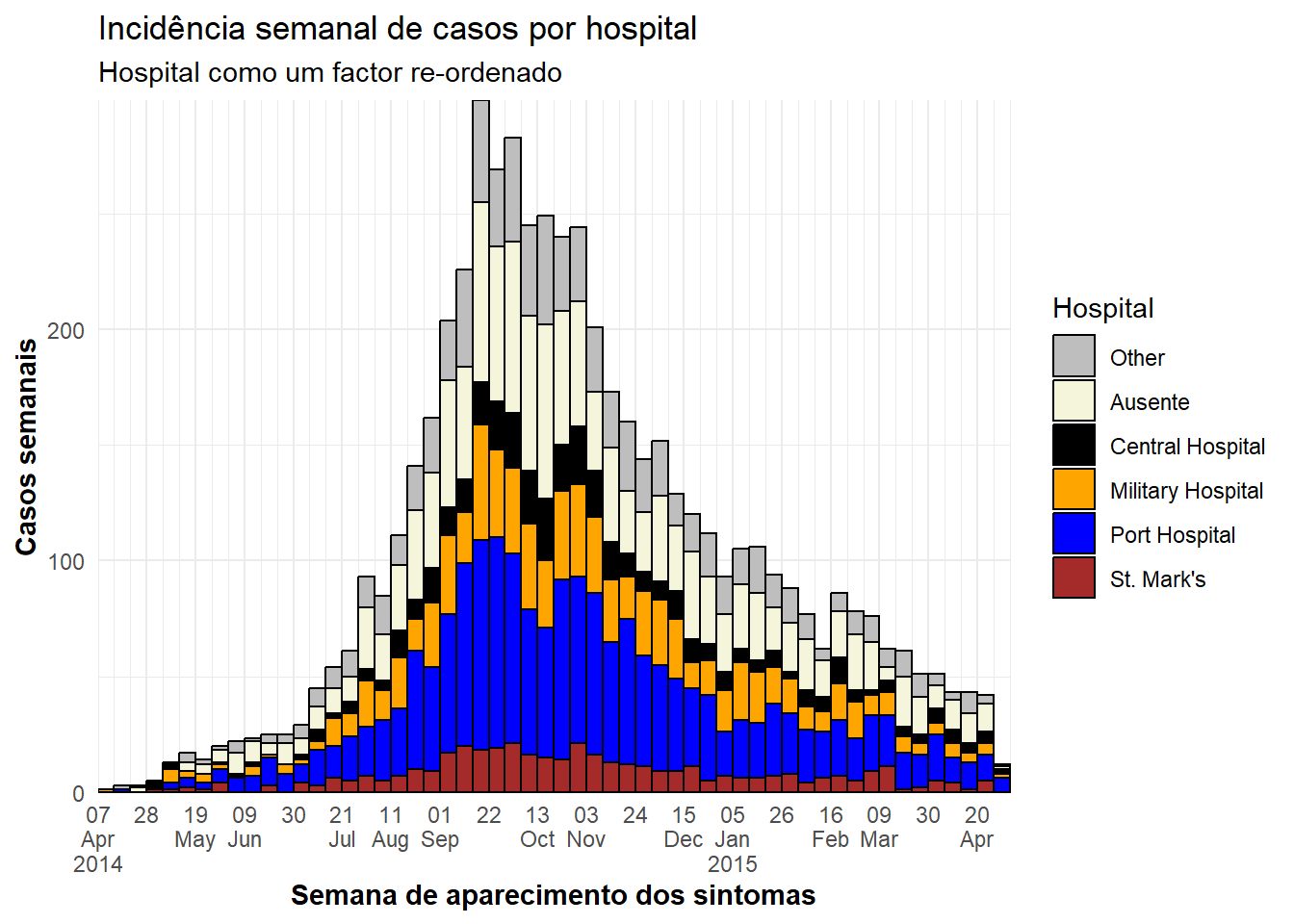
[6] "St. Mark's Maternity Hospital (SMMH)"No gráfico abaixo, a única diferença do gráfico anterior é que a coluna hospital foi consolidada como mostrado acima, e nós utilizamos guides() para reverter a ordem da legenda, de forma que “Missing” está no fim da legenda.
ggplot(plot_data) + # Utilize um NOVO conjunto de dados com hospital reordenado como fator
# crie o histograma
geom_histogram(
mapping = aes(x = date_onset,
group = hospital, # casos agrupados por hospital
fill = hospital), # preenchimento da barra (cor) por hospital
breaks = weekly_breaks_all, # sequência de quebras semanais iniciadas nas segundas para o surto inteiro, como definido no topo da seção sobre ggplot
color = "black")+ # cor da borda ao redor de cada barra
# rótulos do eixo x
scale_x_date(
expand = c(0,0), # remova o excesso de espaço do eixo x antes e após as barras de casos
date_breaks = "3 weeks", # rótulos aparecem a cada 3 semanas que iniciam nas segundas
date_minor_breaks = "week", # linhas verticais aparecem a cada semana iniciada na segunda
label = scales::label_date_short()) + # efficient date label
# eixo y
scale_y_continuous(
expand = c(0,0))+ # remova o espaço em excesso do eixo y abaixo de 0
# especificação manual das cores, atenção para a ordem!
scale_fill_manual(
values = c("grey", "beige", "black", "orange", "blue", "brown"),
labels = c("St. Mark's Maternity Hospital (SMMH)" = "St. Mark's"),
name = "Hospital")+
# temas estéticos
theme_minimal()+ # simplifique o fundo do gráfico
theme(
plot.caption = element_text(face = "italic", # legenda no lado esquerdo em itálico
hjust = 0),
axis.title = element_text(face = "bold"))+ # títulos dos eixos em negrito
# rótulos
labs(
title = "Incidência semanal de casos por hospital",
subtitle = "Hospital como um factor re-ordenado",
x = "Semana de aparecimento dos sintomas",
y = "Casos semanais")DICA: Para reverter apenas a ordem da legenda, adicione o seguinte no comando ggplot2: guides(fill = guide_legend(reverse = TRUE)).
Ajuste a legenda
Leia mais sobre legendas e escalas no página sobre dicas do ggplot. Aqui estão alguns destaques:
- Edite o título da legenda por meio da função de escala ou com
labs(fill = "Legend title")(se você estiver estilizando comcolor =, utilizelabs(color = ""))
-
theme(legend.title = element_blank())para não ter título de legenda -
theme(legend.position = "top")para a legenda acima do gráfico. Pode-se também escolher também “bottom” , “left”, “right” ou “none” para as posições embaixo, à esqueda, à direita ou para remover a legenda, respectivamente. -
theme(legend.direction = "horizontal")legenda horizontal -
guides(fill = guide_legend(reverse = TRUE))para reverter a ordem da legenda
Barras lado-a-lado
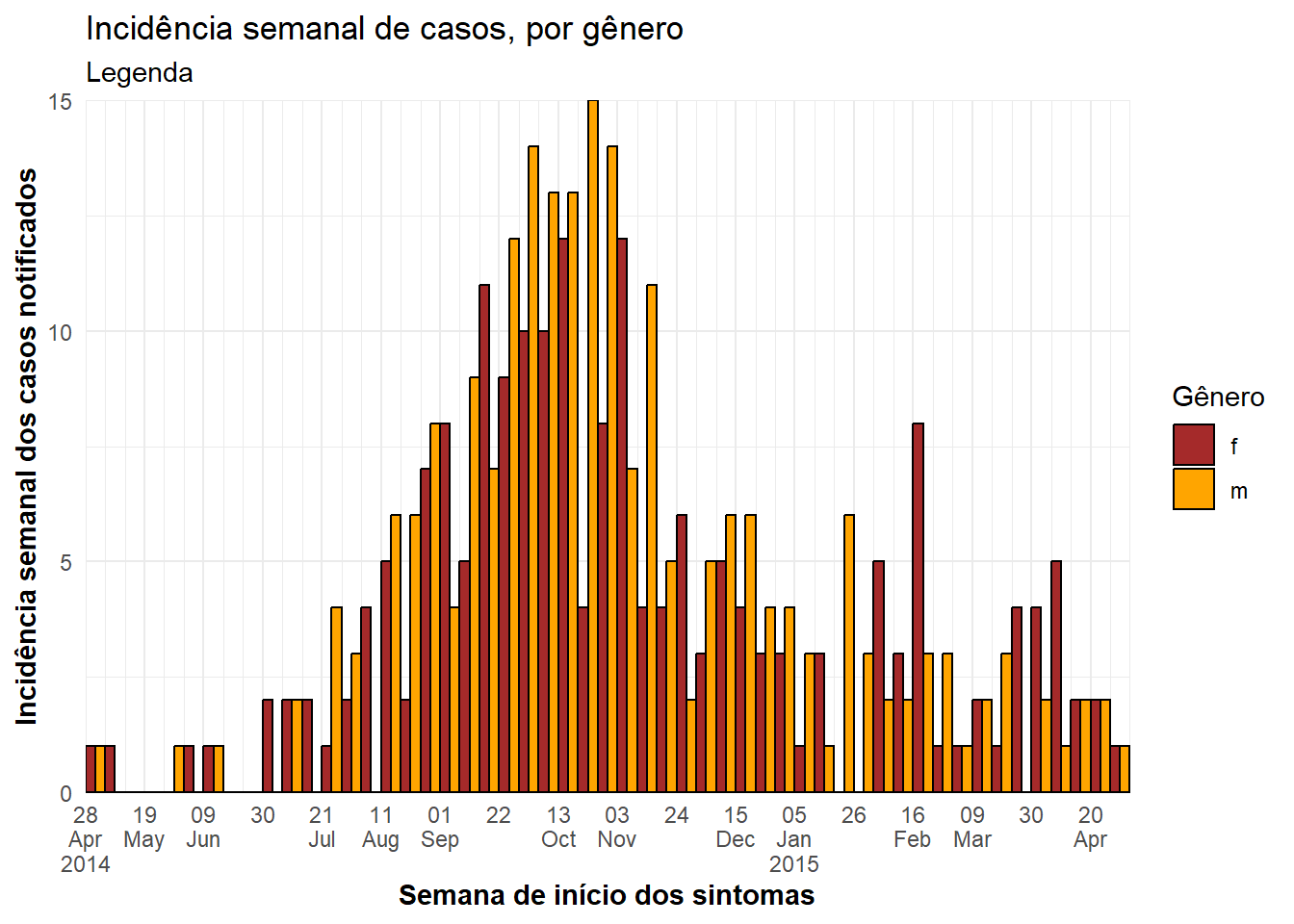
A visualização do grupo de barras lado-a-lado (oposto à posição de barras empilhadas) é especificado dentro de geom_histogram(), com o argumento position = "dodge" fora de aes()(Nota do tradutor: dodge vem do inglês “esquivar”).
Se existirem mais de dois grupos de valores, estes podem ser difíceis de ler. Em vez disso, considere utilizar um gráfico facetado (pequenos múltiplos). Para melhorar a visualização do gráfico neste exemplo, campos sem a informação do gênero foram removidos.
ggplot(central_data %>% drop_na(gender))+ # inicie com casos do Hospital Central, excluindo as linhas sem dados do gênero
geom_histogram(
mapping = aes(
x = date_onset,
group = gender, # casos agrupados por gênero
fill = gender), # barras preenchidas de acordo com o gênero
# quebras dos containers do histrograma
breaks = weekly_breaks_central, # sequência de datas semanais para o surto no Central - definido no topo da seção sobre ggplot
color = "black", # cor do contorno das barras
position = "dodge")+ # barras LADO-A-LADO
# Os rótulos no eixo x
scale_x_date(expand = c(0,0), # remova os espaços em excesso abaixo do eixo x e após as barras de casos
date_breaks = "3 weeks", # rótulos aparecem a cada 3 semanas iniciadas nas segundas
date_minor_breaks = "week", # linhas verticais aparecem a cada semana iniciada nas segundas
label = scales::label_date_short()) + # efficient label formatting
# eixo y
scale_y_continuous(expand = c(0,0))+ # remove o espaço extra nos eixos y entre a base das barras e os rótulos
# escala de cores e rótulos de legendas
scale_fill_manual(values = c("brown", "orange"), # especifique as cores de preenchimento ("values") - atenção na ordem!
na.value = "grey" )+
# temas estéticos
theme_minimal()+ # um conjunto de temas para simplificar o gráfico
theme(plot.caption = element_text(face = "italic", hjust = 0), # título no lado esquerdo em itálico
axis.title = element_text(face = "bold"))+ # título dos eixos em negrito
# rótulos
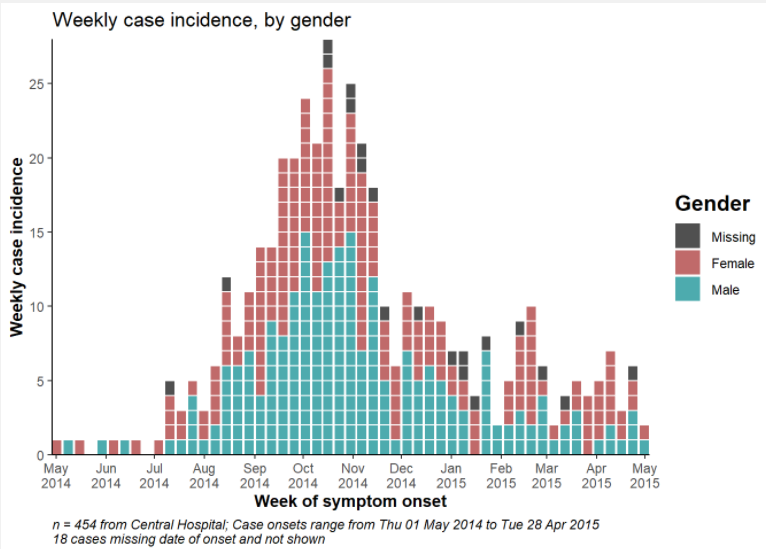
labs(title = "Incidência semanal de casos, por gênero",
subtitle = "Legenda",
fill = "Gênero", # forneça novos títulos para os eixos
x = "Semana de início dos sintomas",
y = "Incidência semanal dos casos notificados")Limite dos eixos
Existem duas formas de limitar a extensão dos valores dos eixos.
Geralmente, o método indicado é utilizar o comando coord_cartesian(), que aceita xlim = c(min, max) e ylim = c(min, max) (onde você fornece os valores mínimos e máximos). Esta ferramenta age como um “zoom” sem, na realidade, remover qualquer dado, o que é importante para estatísticas e resumos das medidas.
Alternativamente, você pode ajustar os valores mínimos e máximos das datas utilizando limits = c() dentro de scale_x_date(). Por exemplo:
scale_x_date(limits = c(as.Date("2014-04-01"), NA)) # escolhe uma data mínima mas deixa a data máxima em aberto.Da mesma forma, se você quiser que o eixo x se estenda até uma data específica (ex.: data atual), mesmo que novos casos não sejam notificados, você pode utilizar:
scale_x_date(limits = c(NA, Sys.Date()) # garante que o eixo da data se estenda até a data atualPERIGO: Tenha cuidado ao ajustar as quebras ou limites da escala do eixo y (ex.: 0 a 30 por 5: seq(0, 30, 5)). Estes números estáticos podem cortar o seu gráfico casos seus dados mudem e ultrapassem os limites!
Rótulos/linha de grade do eixo de data
DICA: Lembre que os rótulos do eixo das datas são independentes da agregação das datas em barras, mas visualmente podem ser importantes para alinhar as classes, rótulos de data, e linhas de grade verticais.
Para modificar os rótulos de data e as linhas de grade, utilize a função scale_x_date() em uma das seguintes formas:
-
Se as suas classes do histograma são dias, semanas iniciadas em segundas, meses, ou anos:
- Utilize
date_breaks =para especificar o intervalo de rótulos e linhas de grade principais (ex.: “day” (dia), “week” (semana), “3 weeks” (3 semanas), “month” (mês), ou “year” (ano)) - Utilize
date_minor_breaks =para especificar o intervalo das linhas de grade verticais secundárias (entre os rótulos) - Adicione
expand = c(0,0)para iniciar os rótulos na primeira barra - Utilize
date_labels =para especificar o formato dos rótulos de data - veja a página sobre Datas para dicas (utilize\npara uma nova linha)
- Utilize
-
Se as suas classes do histograma são semanas iniciadas nos domingos:
- Utilize
breaks =eminor_breaks =ao fornecer uma sequência de quebras de datas para cada um - Você ainda pode utilizar
date_labels =eexpand =para formatação, como decrito acima
- Utilize
Algumas notas:
- Veja a seção de abertura do ggplot para instruções sobre como criar uma sequência de datas utilizando
seq.Date().
- Veja esta página ou a página Trabalhando com datas para dicas sobre como criar rótulos de data.
Demonstrações
Abaixo está uma demonstração de gráficos onde as cásses e os rótulos/linhas de grade do gráfico estão alinhados e desalinhados:
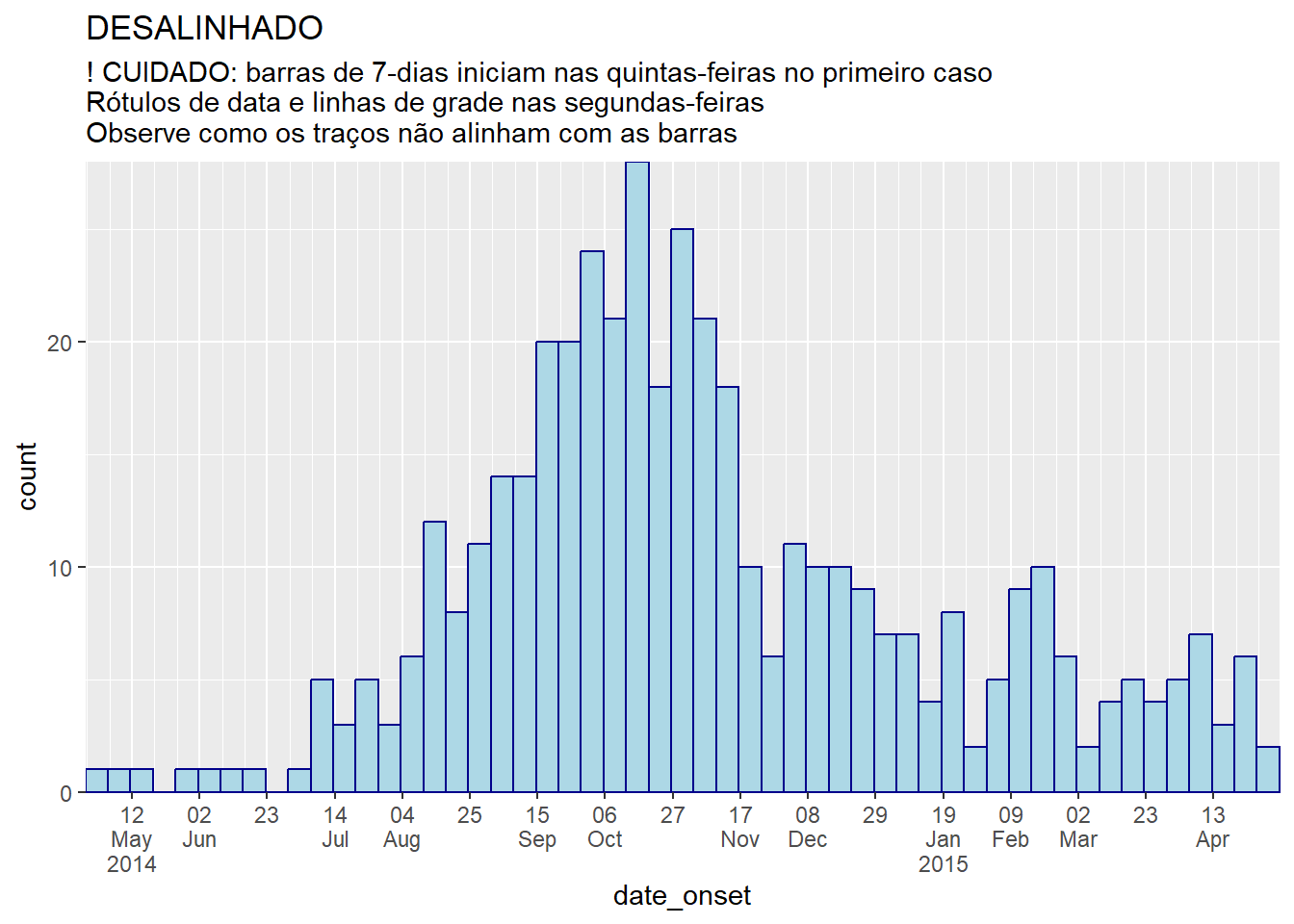
# classes de 7-dias + rótulos de Segunda
#############################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
binwidth = 7, # classes de 7-dias com início no primeiro caso
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # remova o excesso de espaço do eixo x abaixo e após as barras de caso
date_breaks = "3 weeks", # Segunda a cada 3 semanas
date_minor_breaks = "week", # Semanas iniciadas na segunda
label = scales::label_date_short())+ # automatic label formatting
scale_y_continuous(
expand = c(0,0))+ # remova o espaço em excesso abaixo do eixo x, fazendo um nivelamento
labs(
title = "DESALINHADO",
subtitle = "! CUIDADO: barras de 7-dias iniciam nas quintas-feiras no primeiro caso\nRótulos de data e linhas de grade nas segundas-feiras\nObserve como os traços não alinham com as barras")
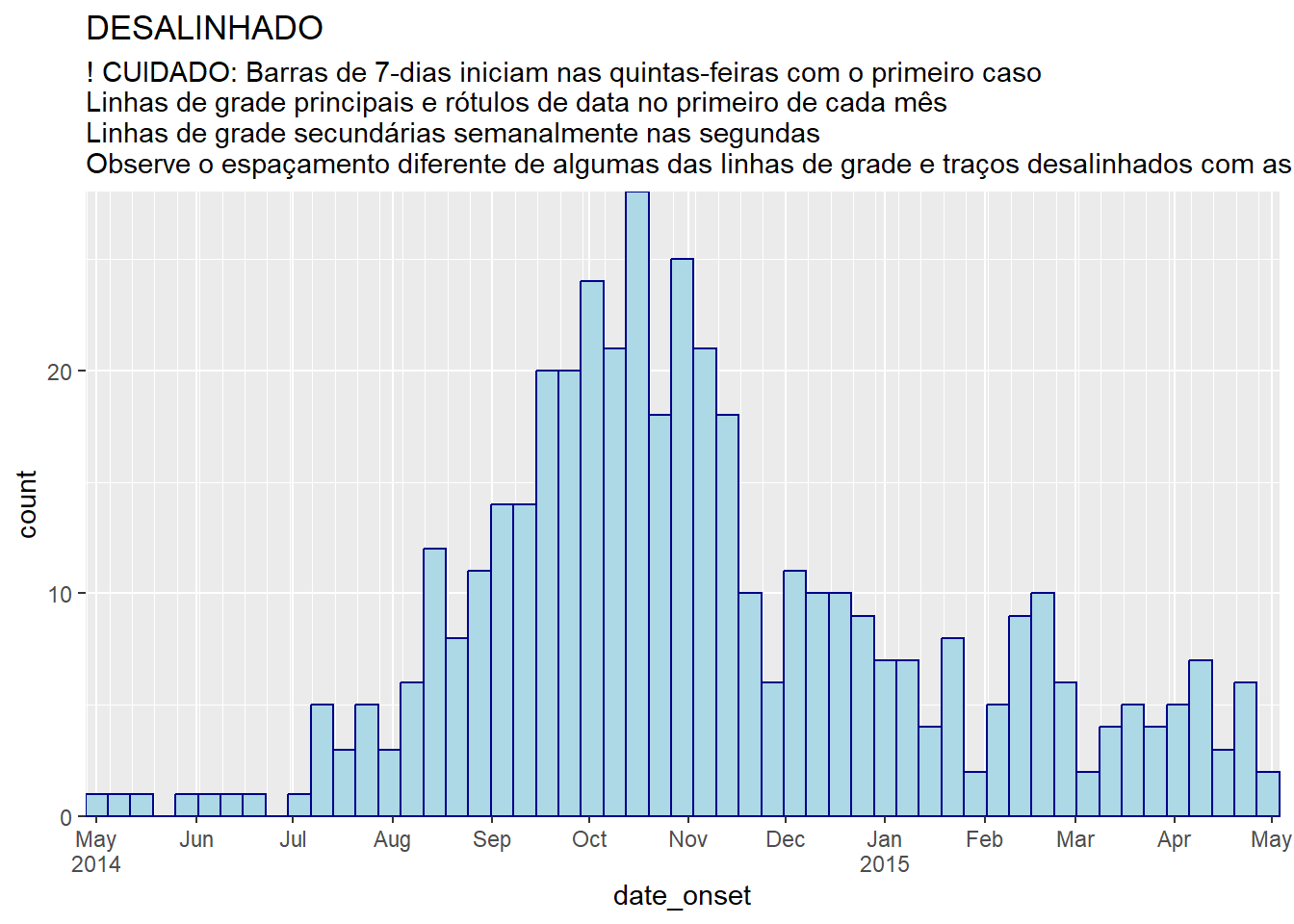
# classes de 7-dias + meses
#####################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
binwidth = 7,
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # remova o espaço em excesso abaixo e após as barras de casos
date_breaks = "months", # primeiro do mês
date_minor_breaks = "week", # semanas iniciadas nas segundas
label = scales::label_date_short())+ # automatic label formatting
scale_y_continuous(
expand = c(0,0))+ # remova o espaço em excesso abaixo do eixo x, faça um nivelamento
labs(
title = "DESALINHADO",
subtitle = "! CUIDADO: Barras de 7-dias iniciam nas quintas-feiras com o primeiro caso\nLinhas de grade principais e rótulos de data no primeiro de cada mês\nLinhas de grade secundárias semanalmente nas segundas\nObserve o espaçamento diferente de algumas das linhas de grade e traços desalinhados com as barras")
# ALINHAMENTO TOTAL NAS SEGUNDAS: especifique manualmente as quebras das classes para serem nas segundas
#################################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# quebras do histograma ajustadas para 7 dias iniciando na Segunda antes do primeiro caso
breaks = weekly_breaks_central, # definido anteriormente nesta página
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # remova o excesso de espaço do eixo x abaixo e após as barras de caso
date_breaks = "4 weeks", # Segunda-feira a cada 4 semanas
date_minor_breaks = "week", # Semanas iniciadas na segunda
label = scales::label_date_short())+ # automatic label formatting
scale_y_continuous(
expand = c(0,0))+ # remova o excesso de espaço abaixo do eixo x, faça o nivelamento
labs(
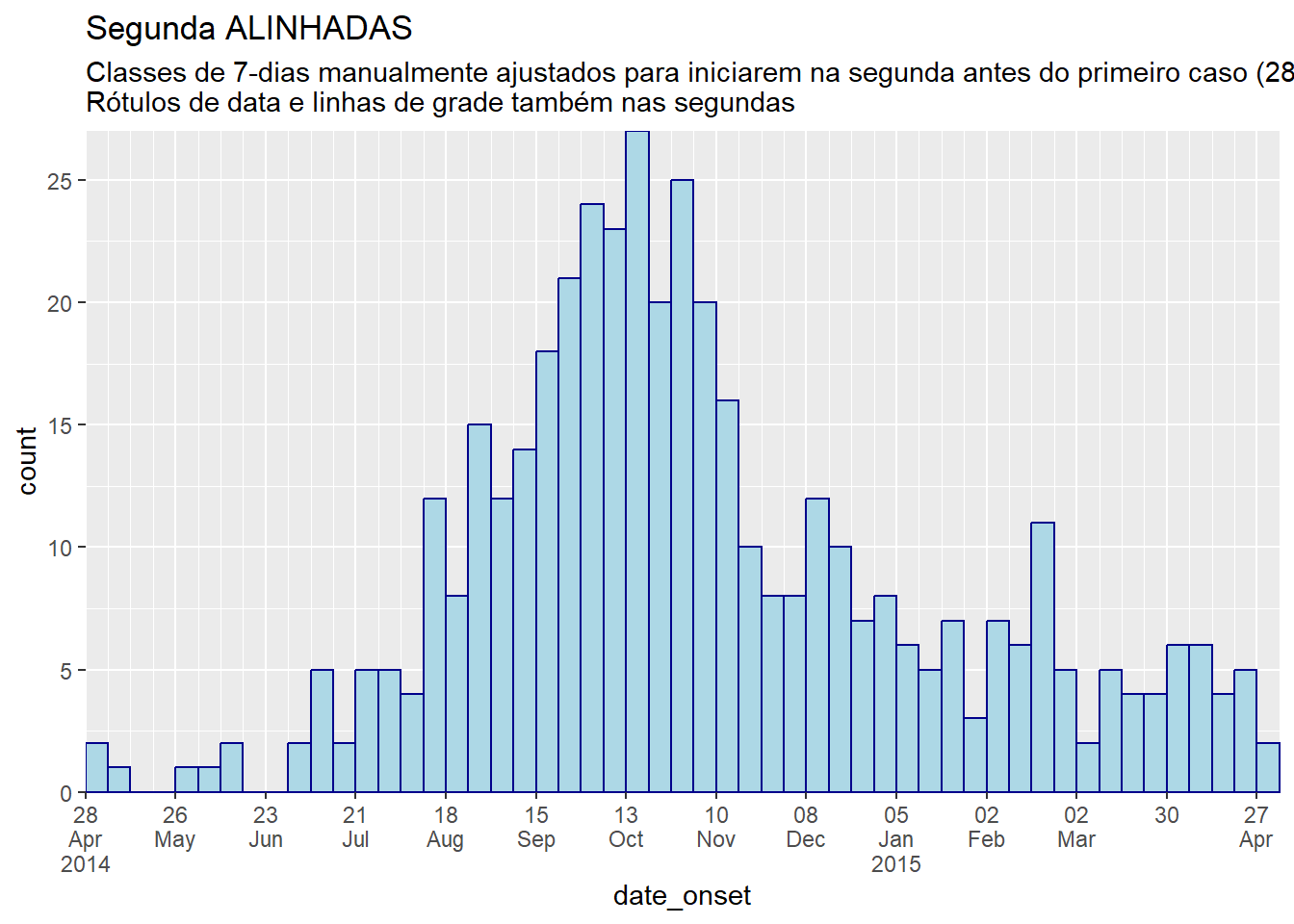
title = "Segunda ALINHADAS",
subtitle = "Classes de 7-dias manualmente ajustados para iniciarem na segunda antes do primeiro caso (28 de abril)\nRótulos de data e linhas de grade também nas segundas")
# ALINHAMENTO TOTAL NA SEGUNDA COM RÓTULOS DE MESES:
############################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# quebras do histograma ajustaddas para 7 dias iniciando na segunda antes do primeiro caso
breaks = weekly_breaks_central, # definido anteriormente nesta página
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # remova o excesso de espaço no eixo x abaixo e após as barras de casos
date_breaks = "months", # Segunda a cada 4 semanas
date_minor_breaks = "week", # Semanas iniciadas nas segundas
label = scales::label_date_short())+ # label formatting
scale_y_continuous(
expand = c(0,0))+ # remova o excesso de espaço abaixo do eixo x, faça um nivelamento
theme(panel.grid.major = element_blank())+ # Remove as linhas de grade principais )caem no primeiro dia do mês)
labs(
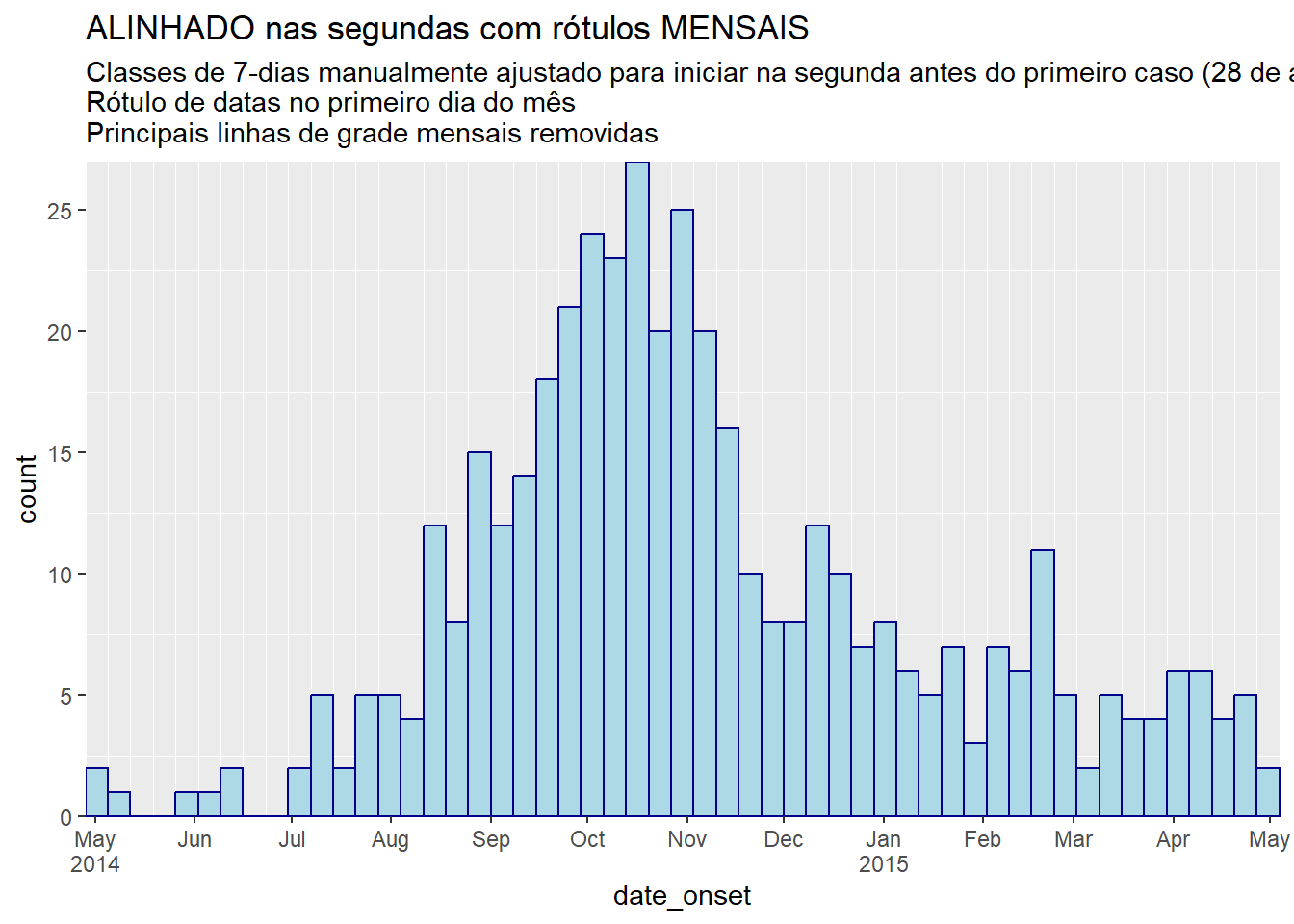
title = "ALINHADO nas segundas com rótulos MENSAIS",
subtitle = "Classes de 7-dias manualmente ajustado para iniciar na segunda antes do primeiro caso (28 de abril)\nRótulo de datas no primeiro dia do mês\nPrincipais linhas de grade mensais removidas")
# ALINHAMENTO TOTAL NO DOMINGO: especifique manualmente as quebras das classes E rótulos para serem nos domingos
############################################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# quebra do histograma ajustadas para serem de 7 dias, iniciando no Domingo antes do primeiro caso
breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "7 days"),
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0),
# quebras dos rótulos de datas e principais linhas de grade ajustadas para ocorrerem a cada 3 semanas iniciando no domingo antes do primeiro caso
breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "3 weeks"),
# linhas de grade secundárias ajustadas para iniciarem semanalmente no domingo antes do primeiro caso
minor_breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "7 days"),
label = scales::label_date_short())+ # automatic label formatting
scale_y_continuous(
expand = c(0,0))+ # remova o espaço em excesso abaixo do eixo x, faça um nivelamento
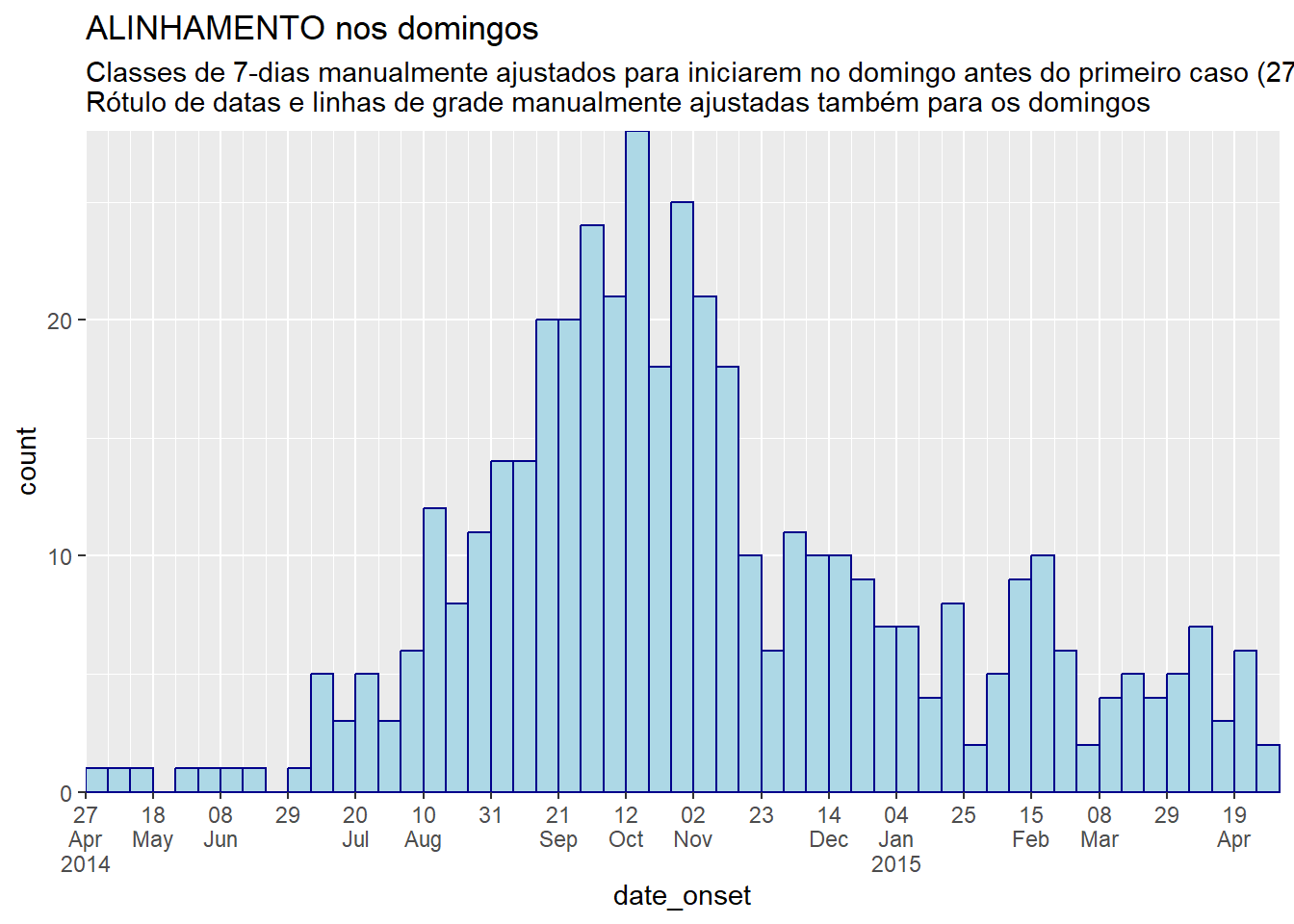
labs(title = "ALINHAMENTO nos domingos",
subtitle = "Classes de 7-dias manualmente ajustados para iniciarem no domingo antes do primeiro caso (27 de abril)\nRótulo de datas e linhas de grade manualmente ajustadas também para os domingos")Dados agregados
Frequentemente, ao invés de uma linelist, você inicia com contagens agregadas de unidades, distritos, etc. Você pode criar uma epicurva com o ggplot(), mas o código será levemente diferente. Esta seção irá utilizar o conjunto de dados do count_data que foi importado anteriormente, na seção de preparação dos dados. Este conjunto de dados é o linelist agregado para contagens diárias por hospital. As primeiras 50 linhas são mostradas abaixo.
Criando um gráfico de contagens diárias
Nós podemos criar um gráfico de uma epicurva diária destas contagens diárias. Aqui estão as diferenças no código:
- Dentro do mapeamento estético
aes(), especifiquey =como a coluna de contagem (neste caso, o nome da coluna én_cases) - Adicione o argumento
stat = "identity"dentro degeom_histogram(), que especifica que a altura da barra deve ser o valory =, e não o número de linhas, como é o padrão - Adicione o argumento
width =para evitar linhas verticais brancas entre as barras. Para contagens diárias o ajuste é 1. Para contagens semanais o ajuste é 7. Para contagens mensais, linhas brancas são um problema (cada M~es possui diferente número de dias) - considere transformar seu eixo x para um factor ordenado categoricamente (meses) e utilizandogeom_col().
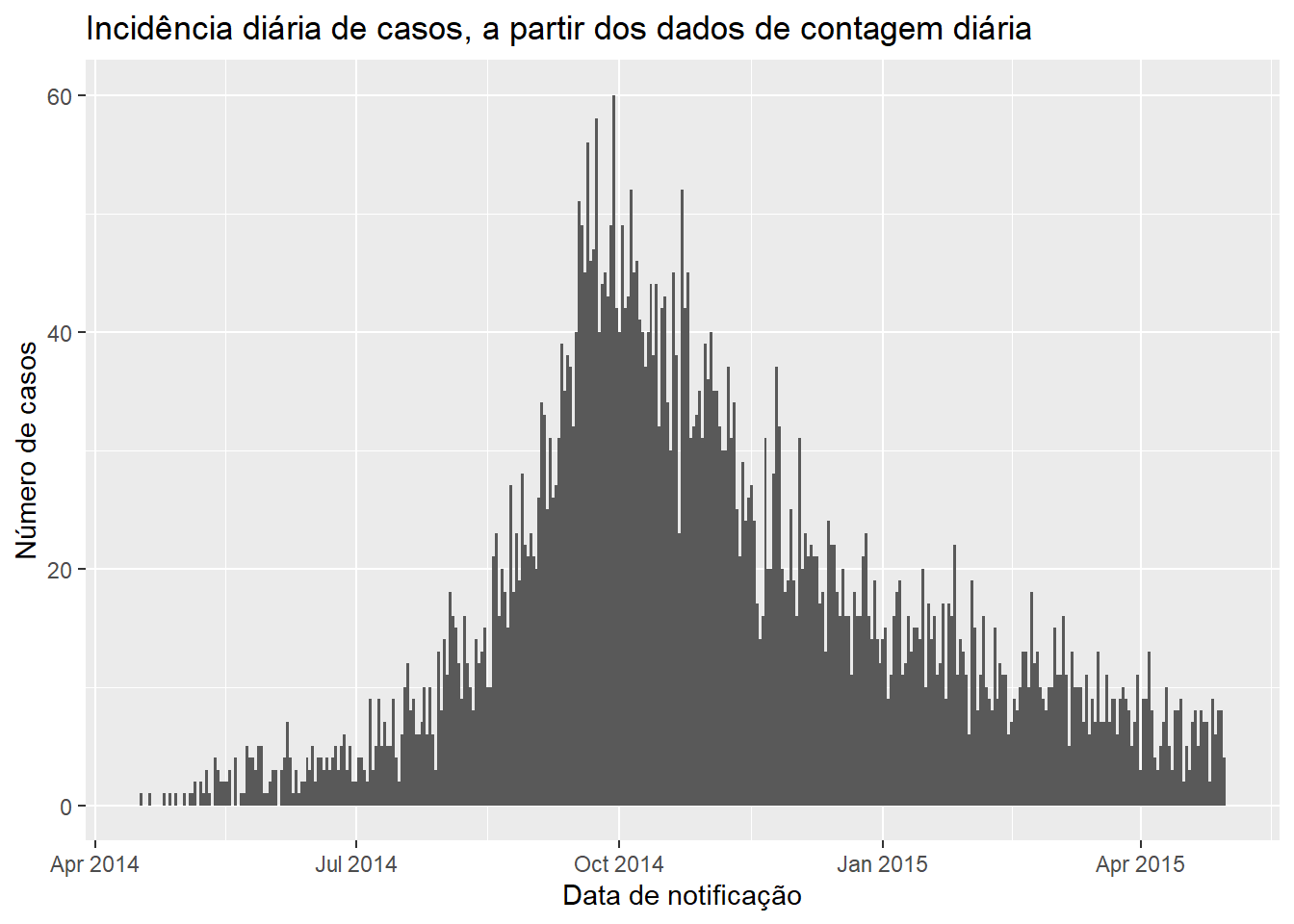
ggplot(data = count_data)+
geom_histogram(
mapping = aes(x = date_hospitalisation, y = n_cases),
stat = "identity",
width = 1)+ # para contagens diárias, ajuste width = 1 para evitar o espaço braco entre as barras
labs(
x = "Data de notificação",
y = "Número de casos",
title = "Incidência diária de casos, a partir dos dados de contagem diária")Criando um gráfico de contagens semanais
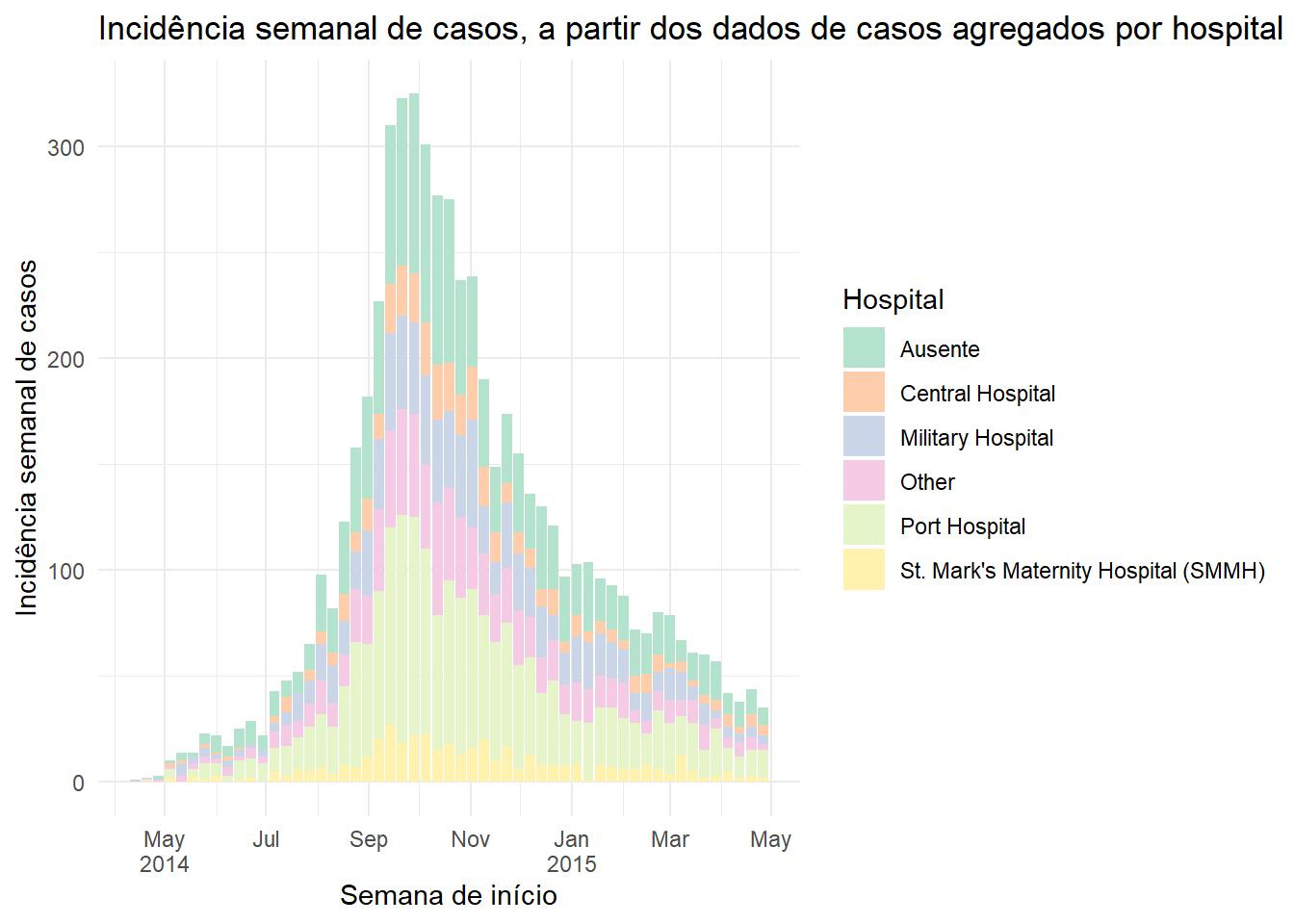
Se os seus casos já estão contados por semana, eles podem parecer como o seguinte conjunto de dados (chamado count_data_weekly):
As primeiras 50 linhas de count_data_weekly são mostradas abaixo. Você pode ver que as contagens foram agregadas por semanas. Cada semana é mostrada pelo primeiro dia da semana (segunda-feira, por padrão).
Agora crie o gráfico de forma que x = a coluna da semana epidemiológica. Lembre de adicionar y = a coluna de contagens para o mapeamento estético, e adicione stat = "identity", como explicado acima.
ggplot(data = count_data_weekly)+
geom_histogram(
mapping = aes(
x = epiweek, # eixo x é a semana epidemiológica (variável da classe Data)
y = n_cases_weekly, # altura do eixo y nas contagens de casos semanais
group = hospital, # nós estamos agrupando as barras e colorindo por hospital
fill = hospital),
stat = "identity")+ # isto também é requerido quando criar um gráfico dos dados de contagem
# rótulos para o eixo x
scale_x_date(
date_breaks = "2 months", # rótulos a cada 2 meses
date_minor_breaks = "1 month", # linhas de grade a cada mês
label = scales::label_date_short())+ # label formatting
# Escolha a paleta de cor (utiliza o pacote RColorBrewer)
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()+
labs(
x = "Semana de início",
y = "Incidência semanal de casos",
fill = "Hospital",
title = "Incidência semanal de casos, a partir dos dados de casos agregados por hospital")Média móvel
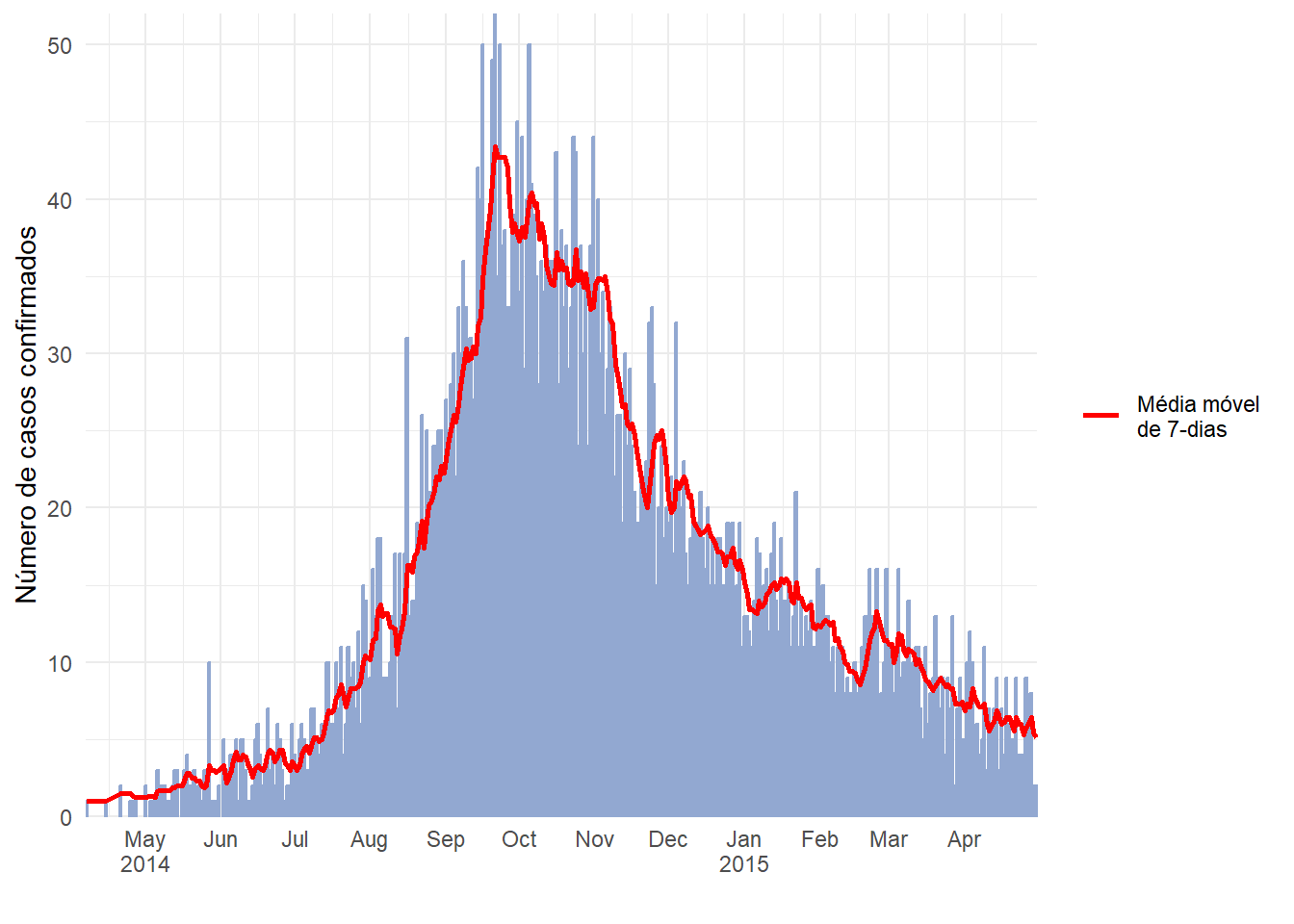
Veja a página sobre médias móveis para uma descrição detalhada e diferentes opções. Abaixo, uma alternativa para calcular as médias móveis com o pacote slider é utilizada. Nesta abordagem, a média móvel é calculada no conjunto de dados antes de criar os gráficos:
- Agregue os dados em contagens conforme necessário (diárias, semanais, etc.) (veja a página Agrupando dados)
- Crie uma nova coluna para salvar a média móvel, criada com
slide_index()do pacote slider
- Crie um gráfico da média móvel como uma
geom_line()acima do (após) o histograma da epicurva
Veja este útil resumo online do pacote slider
# carregue o pacote
pacman::p_load(slider) # slider utilizado para calcular as médias móveis
# crie um conjunto de dados de contagens diárias e média móvel de 7-dias
#######################################################
ll_counts_7day <- linelist %>% # inicie com o objeto linelist
## conte os casos por dia
count(date_onset, name = "new_cases") %>% # crie uma nova coluna com as contagens, chamada "new_cases"
drop_na(date_onset) %>% # remova os casos sem a informação do dia de início dos sintomas (date_onset)
## calcule o número médio de casos em uma janela de 7 dias
mutate(
avg_7day = slider::slide_index( # crie uma nova coluna
new_cases, # calcule baseado nos valores da coluna new_cases
.i = date_onset, # o indexador é a coluna de date_onset, de forma que as contagens sem datas são incluídas na janela de análise
.f = ~mean(.x, na.rm = TRUE), # a função utilizada é mean() com os valores em branco removidos
.before = 6, # a janela de análise é o dia e os 6-dias anteriores
.complete = FALSE), # precisa ser FALSE para unlist() funcionar na próxima etapa
avg_7day = unlist(avg_7day)) # converta da classe "list" para a classe "numeric"
# crie o gráfico
######
ggplot(data = ll_counts_7day) + # inicie com o novo conjunto de dados criado acima
geom_histogram( # crie uma epicurva em histograma
mapping = aes(
x = date_onset, # coluna de datas no eixo x
y = new_cases), # a altura é o número de novos casos diários
stat = "identity", # altura da coluna é o valor de y
fill="#92a8d1", # cor legal para as barras
colour = "#92a8d1", # mesma cor para a borda das barras
)+
geom_line( # crie uma linha para a média móvel
mapping = aes(
x = date_onset, # coluna de data para o eixo x
y = avg_7day, # valor de y ajustado para a coluna de média móvel
lty = "Média móvel \nde 7-dias"), # nome da linha na legenda
color="red", # cor da linha
size = 1) + # espessura da linha
scale_x_date( # escala da data
date_breaks = "1 month",
label = scales::label_date_short(), # label formatting
expand = c(0,0)) +
scale_y_continuous( # escala do eixo y
expand = c(0,0),
limits = c(0, NA)) +
labs(
x="",
y ="Número de casos confirmados",
fill = "Legenda")+
theme_minimal()+
theme(legend.title = element_blank()) # remove o título da legendaFacetas/múltiplos pequenos
Como em outros ggplots, você pode criar gráficos facetados (“múltiplos pequenos”). Como explicado na página dicas do ggplot deste manual, você pode utilizar tanto facet_wrap() quanto facet_grid(). Aqui, nós demonstramos com o facet_wrap(). Para epicurvas, facet_wrap() é tipicamente mais fácil, uma vez que provavelmente você só precisa facetar uma coluna.
A sintaxe geral é facet_wrap(rows ~ cols), em que no lado esquerdo do til (~) é o nome da coluna a ser espalhada através das “linhas” do gráfico facetado, e no lado direito do til é o nome de uma coluna a ser espalhada através das “colunas” do gráfico facetado. De forma mais simples, só utilize um nome de coluna, no lado direito do til: facet_wrap(~age_cat).
Eixos livres
Você precisará decidir se as escalas dos eixos para cada faceta são “fixas” para as mesmas dimensões (padrão), ou “livres” (significando que irão mudar baseado nos dados dentro da faceta). Faça isso com o argumento scales = dentro de facet_wrap() ao especificar “free_x” ou “free_y”, ou “free”.
Número de colunas e linhas das facetas
Isto pode ser especificado com ncol = e nrow = dentro de facet_wrap().
Ordem dos painéis
Para alterar a ordem de aparecimento, altere a ordem dos níveis da coluna de factor utilizada para criar as facetas.
Estética
Tamanho da fonte e face, cor da tira, etc. podem ser modificados através de theme() com argumentos como:
-
strip.text = element_text()(tamanho, cor, face, ângulo…) -
strip.background = element_rect()(ex.: element_rect(fill=“grey”))
-
strip.position =(posição da tira “bottom” (embaixo), “top” (em cima), “left” (à esquerda), ou “right” (à direita))
Rótulos das tiras
Rótulos dos gráficos facetados podem ser modificados por meio de “rótulos” da coluna como um factor, ou pelo uso de um “rotulador”.
Crie um rotulador como este, utilizando a função as_labeller() do ggplot2. Então, forneça o rotulador para o argumento labeller = de facet_wrap(), como mostrado abaixo.
my_labels <- as_labeller(c(
"0-4" = "Ages 0-4",
"5-9" = "Ages 5-9",
"10-14" = "Ages 10-14",
"15-19" = "Ages 15-19",
"20-29" = "Ages 20-29",
"30-49" = "Ages 30-49",
"50-69" = "Ages 50-69",
"70+" = "Over age 70"))Um exemplo de gráfico facetado - facetado pela coluna age_cat.
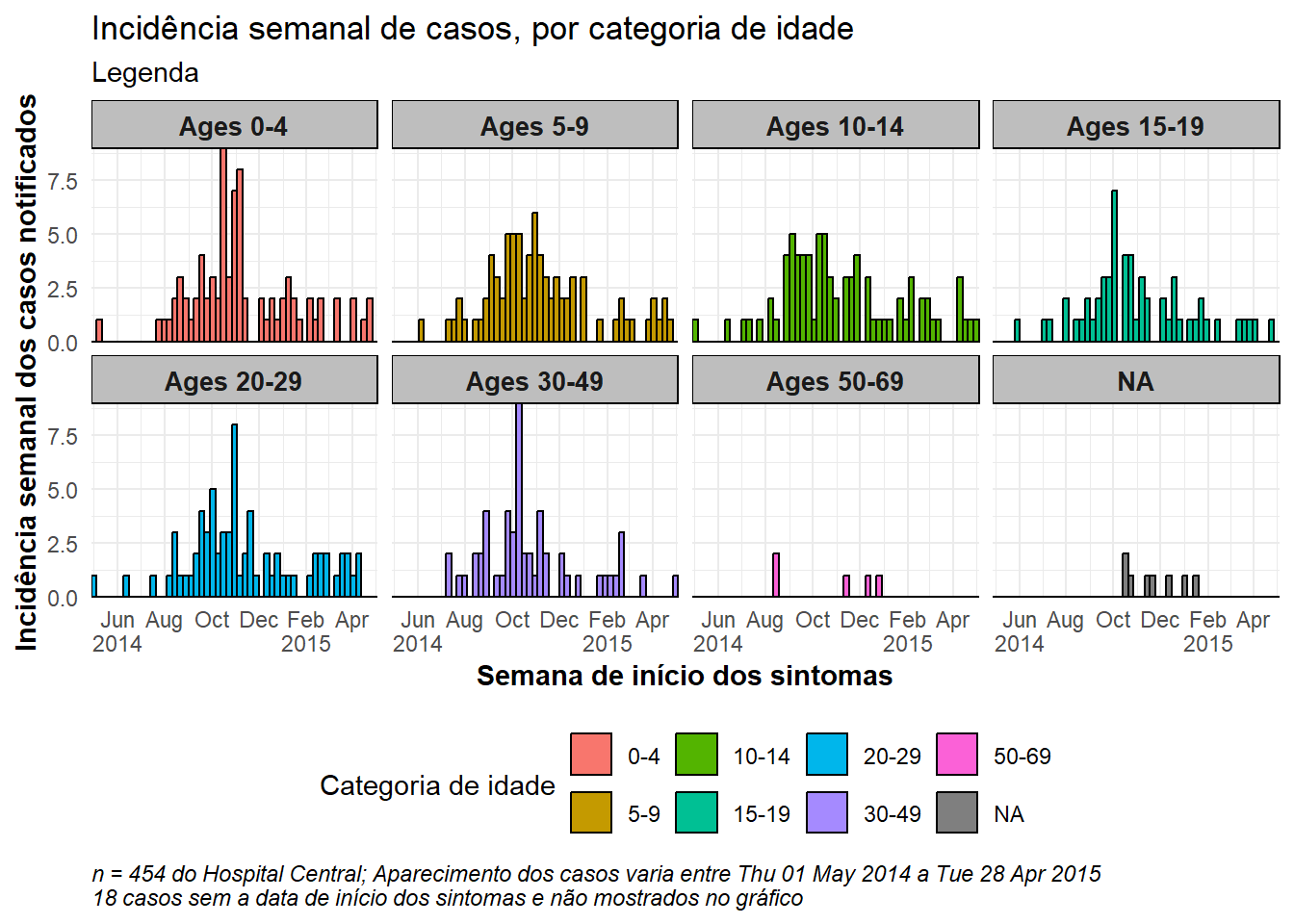
# crie o gráfico
###########
ggplot(central_data) +
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # argumentos dentro de aes() aplicam-se ao grupo
color = "black", # argumentos fora de aes() aplicam-se a todos os dados
# quebras do histograma
breaks = weekly_breaks_central)+ # vetor pré-definido de datas (veja mais acima nesta página)
# Os rótulos no eixo x
scale_x_date(
expand = c(0,0), # remove o espaço em excesso do eixo x abaixo e após as barras de casos
date_breaks = "2 months", # rótulos aparecem a cada 2 meses
date_minor_breaks = "1 month", # linhas verticais aparecem a cada mês
label = scales::label_date_short())+ # label formatting
# eixo y
scale_y_continuous(expand = c(0,0))+ # remove o espaço em excesso do eixo y entre os fundos das barras e os rótulos
# temas estéticos
theme_minimal()+ # um conjunto de temas para simplificar a plotagem
theme(
plot.caption = element_text(face = "italic", hjust = 0), # título no lado esquerdo em itálico
axis.title = element_text(face = "bold"),
legend.position = "bottom",
strip.text = element_text(face = "bold", size = 10),
strip.background = element_rect(fill = "grey"))+ # títulos dos eixos em negrito
# crie as facetas
facet_wrap(
~age_cat,
ncol = 4,
strip.position = "top",
labeller = my_labels)+
# rótulos
labs(
title = "Incidência semanal de casos, por categoria de idade",
subtitle = "Legenda",
fill = "Categoria de idade", # forneça um novo título para a legenda
x = "Semana de início dos sintomas",
y = "Incidência semanal dos casos notificados",
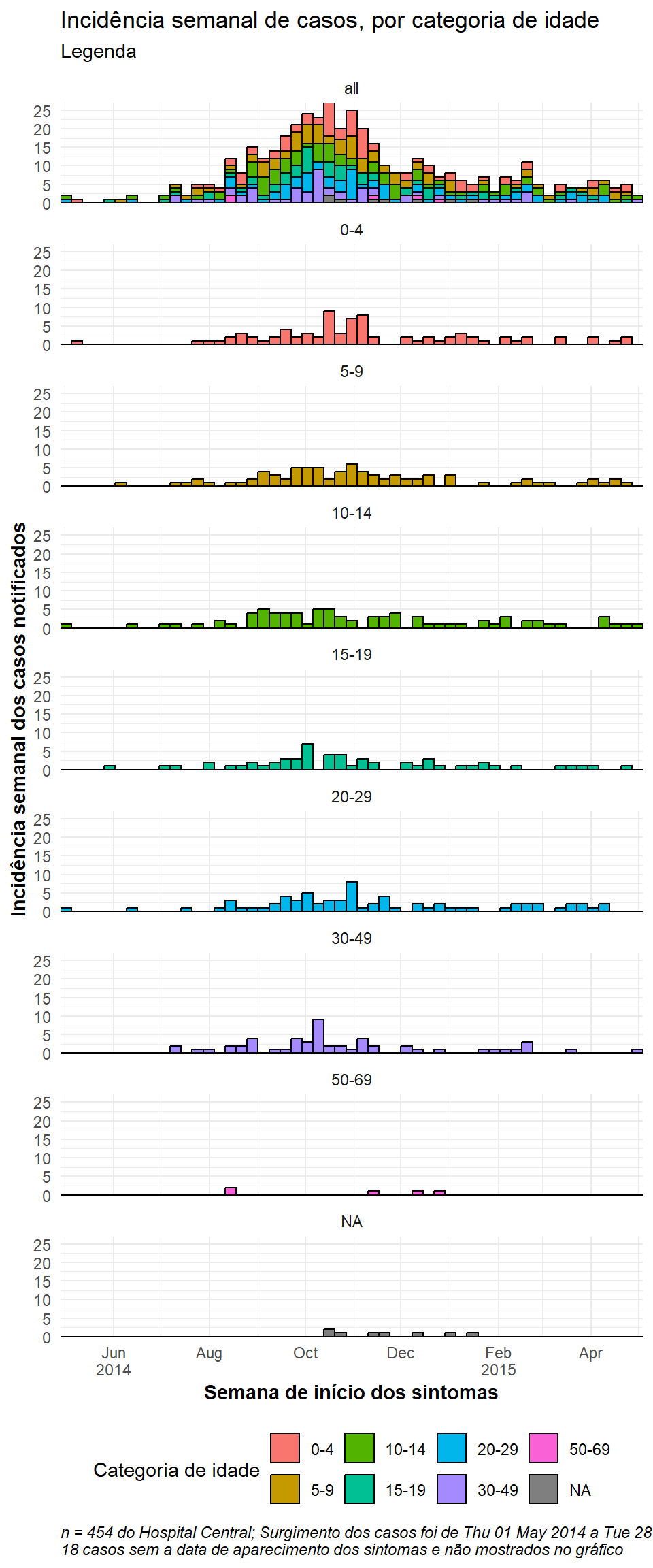
caption = stringr::str_glue("n = {nrow(central_data)} do Hospital Central; Aparecimento dos casos varia entre {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} a {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} casos sem a data de início dos sintomas e não mostrados no gráfico"))Veja este link para mais informações sobre os rotuladores.
Epidemia total no fundo da faceta
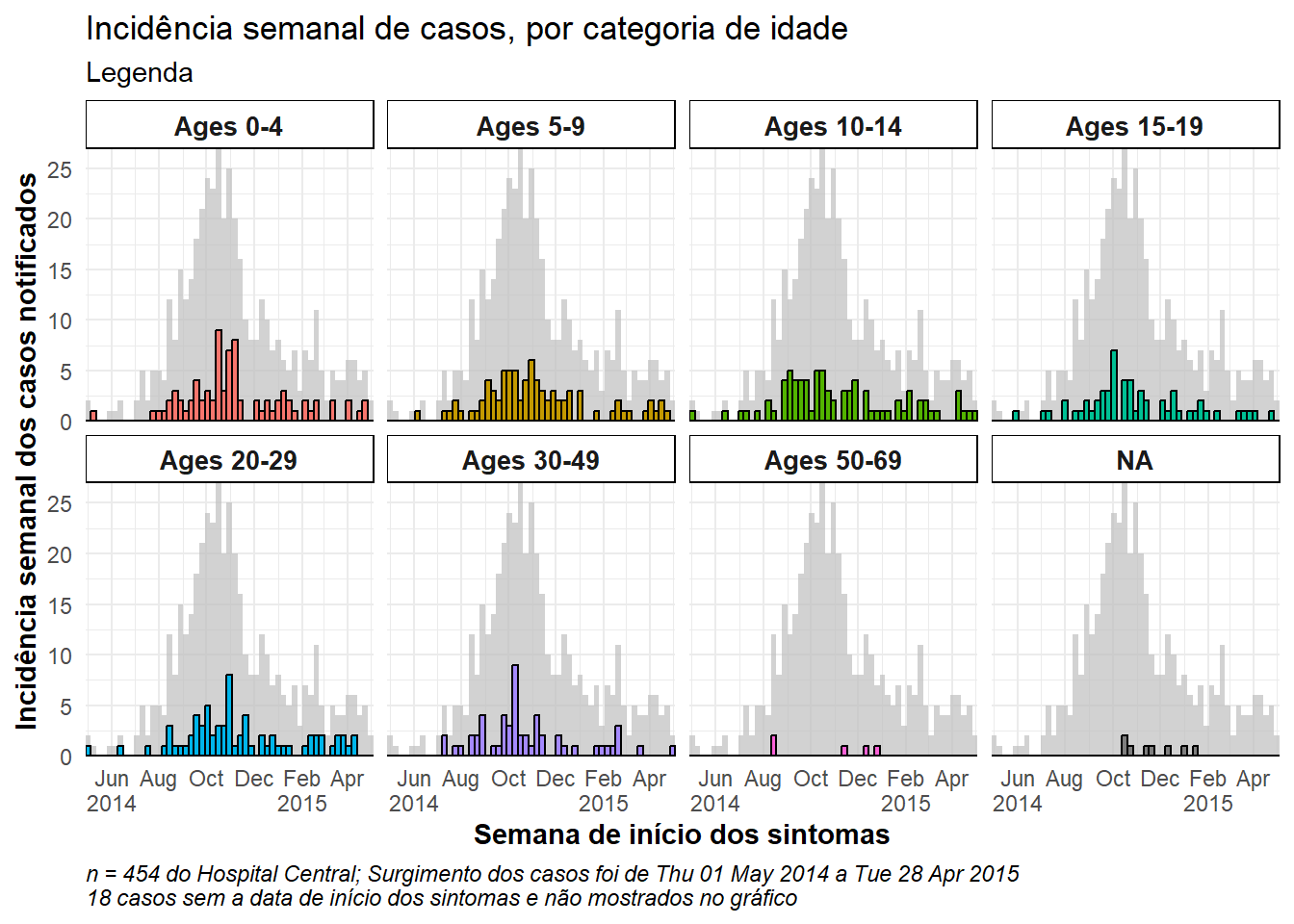
Para mostrar o total da epidemia no fundo de cada faceta, adicione a função gghighlight() com parênteses vazios ao ggplot. Isto é do pacote gghighlight. Observe que o máximo do eixo y em todas as facetas é agora baseado no pico da epidemia inteira. Existem mais exemplos deste pacote na página sobre dicas do ggplot.
ggplot(central_data) +
# epicurvas por grupo
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # argumentos dentro de aes() são aplicados por grupo
color = "black", # argumentos fora de aes() são aplicados a todos os dados
# quebras do histograma
breaks = weekly_breaks_central)+ # vetor de datas pré-definidas (veja o topo da seção sobre ggplot)
# adicione a epidemia total em cinza em cada faceta
gghighlight::gghighlight()+
# rótulos no eixo x
scale_x_date(
expand = c(0,0), # remova o espaço em excesso do eixo x abaixo e após as barras de casos
date_breaks = "2 months", # rótulos aparecem a cada 2 meses
date_minor_breaks = "1 month", # linhas verticais aparecem a cada 1 mês
label = scales::label_date_short())+ # label formatting
# eixo y
scale_y_continuous(expand = c(0,0))+ # remove o excesso de espaço do eixo y abaixo de 0
# temas estéticos
theme_minimal()+ # um conjunto de temas para simplificar o gráfico
theme(
plot.caption = element_text(face = "italic", hjust = 0), # título no lado esquerdo em itálico
axis.title = element_text(face = "bold"),
legend.position = "bottom",
strip.text = element_text(face = "bold", size = 10),
strip.background = element_rect(fill = "white"))+ # títulos dos eixos em negrito
# crie as facetas
facet_wrap(
~age_cat, # cada gráfico é um valor de age_cat
ncol = 4, # número de colunas
strip.position = "top", # posição do título/tira da faceta
labeller = my_labels)+ # rotulador definido acima
# rótulos
labs(
title = "Incidência semanal de casos, por categoria de idade",
subtitle = "Legenda",
fill = "Categoria de idade", # forneça um novo título para a legenda
x = "Semana de início dos sintomas",
y = "Incidência semanal dos casos notificados",
caption = stringr::str_glue("n = {nrow(central_data)} do Hospital Central; Surgimento dos casos foi de {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} a {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} casos sem a data de início dos sintomas e não mostrados no gráfico"))Uma faceta com dados
Se você quer ter uma caixa de faceta que contem todos os dados, duplique o conjunto de dados inteiro e trate as duplicatas como um valor de facetas. Uma função “auxiliar”, CreateAllFacet(), abaixo pode auxiliar nisso (agradecimento a esse post). Quando é executado, o número de linhas duplica, e então terá uma nova coluna chamada facet, em que as linhas duplicadas terão o valor “all”, e as linhas originais terão o valor original da coluna de facetas. Agora você só precisa facetear com a coluna facet.
Aqui está a função auxiliar. Execute ela de forma que esteja disponível para você.
# Defina uma função auxiliar
CreateAllFacet <- function(df, col){
df$facet <- df[[col]]
temp <- df
temp$facet <- "all"
merged <-rbind(temp, df)
# garanta que o valor da faceta é um fator
merged[[col]] <- as.factor(merged[[col]])
return(merged)
}Agora aplique a função auxiliar para o conjunto de dados, na coluna age_cat:
# Crie um conjunto de dados que é duplicado e com a nova coluna "facet" para mostrar "all" (todas) as categorias de idade como outro nível da faceta
central_data2 <- CreateAllFacet(central_data, col = "age_cat") %>%
# ajuste os níveis do fator
mutate(facet = fct_relevel(facet, "all", "0-4", "5-9",
"10-14", "15-19", "20-29",
"30-49", "50-69", "70+"))Warning: There was 1 warning in `mutate()`.
ℹ In argument: `facet = fct_relevel(...)`.
Caused by warning:
! 1 unknown level in `f`: 70+# verifique os níveis
table(central_data2$facet, useNA = "always")
all 0-4 5-9 10-14 15-19 20-29 30-49 50-69 <NA>
454 84 84 82 58 73 57 7 9 Alterações notáveis no comando ggplot() são:
- Os dados utilizados agora são central_data2 (duplique as linhas, com a nova coluna “facet”)
- Rotulador precisará ser atualizado, caso usado
- Opcional: para obter facetas empilhadas verticalmente: a coluna facetada é movida para o lado das linhas na equação e no lado esquerdo é substituído por “.” (
facet_wrap(facet~.)), encol = 1. Você também pode precisar ajustar o comprimento e altura da imagem do gráfico salvo (vejaggsave()no dicas do ggplot).
ggplot(central_data2) +
# epicurvas atuais por grupo
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # argumentos dentro de aes() são aplicados por grupo
color = "black", # argumentos fora de aes() são aplicados a todos os dados
# quebras do histograma
breaks = weekly_breaks_central)+ # vetor de datas pré-definidos (veja o topo da seção sobre o ggplot)
# Rótulos no eixo x
scale_x_date(
expand = c(0,0), # remova o excesso de espaço no eixo x abaixo e após as barras de casos
date_breaks = "2 months", # rótulos aparecem a cada 2 meses
date_minor_breaks = "1 month", # linhas verticais aparecem a cada mês
label = scales::label_date_short())+ # label formatting
# eixo y
scale_y_continuous(expand = c(0,0))+ # remove o espaço em excesso do eixo y entre a base das barras e os rótulos
# temas estéticos
theme_minimal()+ # um conjunto de temas para simplificar o gráfico
theme(
plot.caption = element_text(face = "italic", hjust = 0), # título no lado esquerdo em itálico
axis.title = element_text(face = "bold"),
legend.position = "bottom")+
# crie as facetas
facet_wrap(facet~. , # cada gráfico é um valor da faceta
ncol = 1)+
# rótulos
labs(title = "Incidência semanal de casos, por categoria de idade",
subtitle = "Legenda",
fill = "Categoria de idade", # forneça um novo título para a legenda
x = "Semana de início dos sintomas",
y = "Incidência semanal dos casos notificados",
caption = stringr::str_glue("n = {nrow(central_data)} do Hospital Central; Surgimento dos casos foi de {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} a {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} casos sem a data de aparecimento dos sintomas e não mostrados no gráfico"))32.3 Dados preliminares
Os dados mais recentes mostrados em epicurvas precisam, frequentemente, serem marcados como preliminares, ou sujeitos à demoras na notificação. Isto pode ser feito ao adicionar uma linha vertical e/ou retângulo sobre um número específico de dias. Aqui estão duas opções:
- Utilize
annotate():- Para utilizar uma linha, use
annotate(geom = "segment"). Forneçax,xend,y, eyend. Ajuste o tamanho, tipo de linha (lty), e cor. - Para utilizar um retângulo, use
annotate(geom = "rect"). Forneça xmin/xmax/ymin/ymax. Ajuste a cor e o alfa.
- Para utilizar uma linha, use
- Agrupe os dados por status da tentativa e utilize cores diferentes para essas barras
CUIDADO: Você pode querer experimentar geom_rect() para desenhar um retângulo, mas ajustar a transparência não funciona no contexto de uma linelist. Esta função sobrepõe um retângulo para cada observação/linha!. Utilize ou um alfa muito baixo (ex.: 0.01), ou outra abordagem.
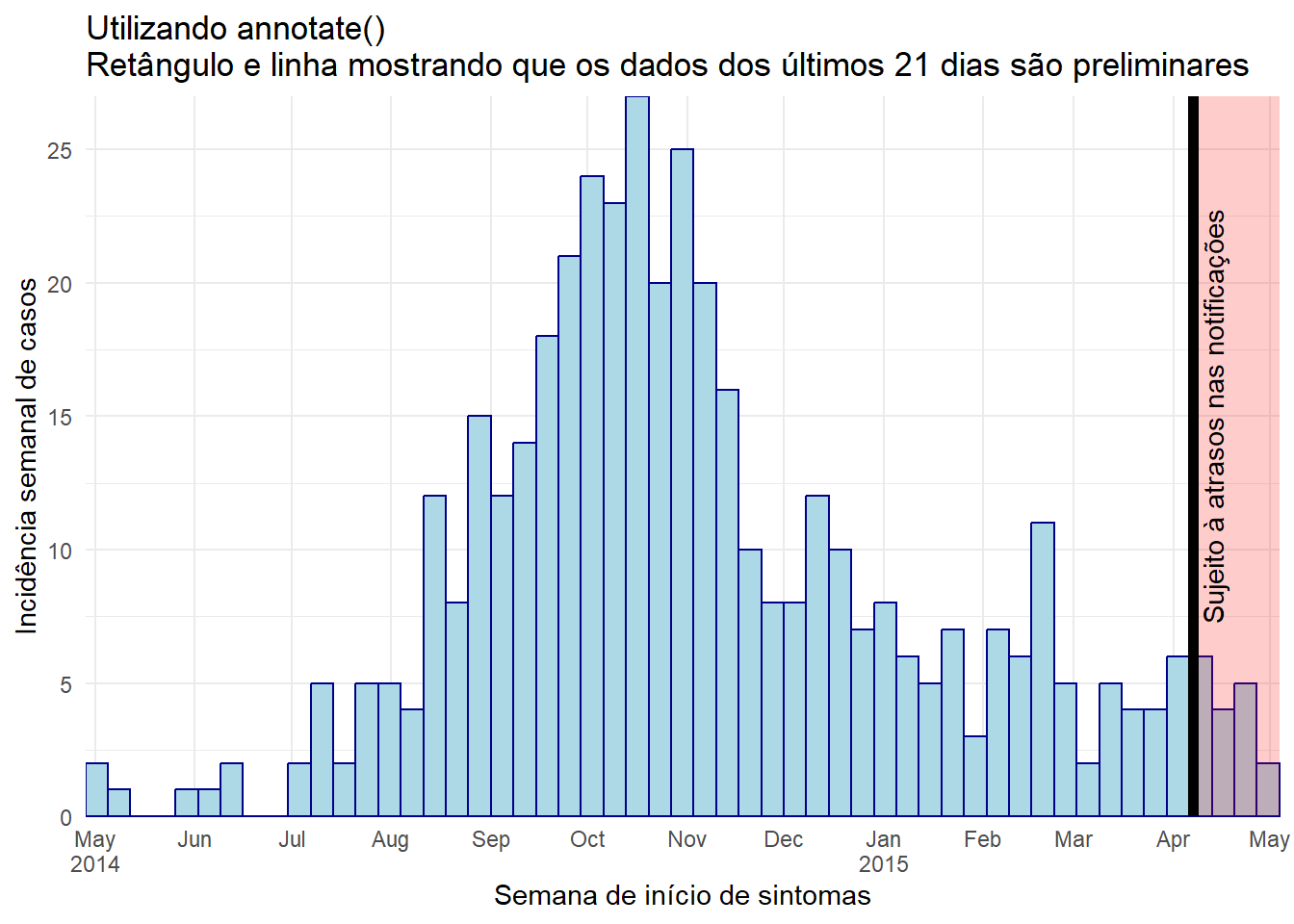
Utilizando annotate()
- Dentro de
annotate(geom = "rect"), os argumentosxminexmaxprecisam ser dados como valores da classe Date (data). - Note que, devido ao fato destes dados serem agregados em barras semanais, e a última barra extender para a segunda após o último caso, a região sombreada pode parecer cobrir 4 semanas
- Aqui é um exemplo online de
annotate()
ggplot(central_data) +
# histograma
geom_histogram(
mapping = aes(x = date_onset),
breaks = weekly_breaks_central, # vetor de data pré-definido - veja o topo da seção sobre o ggplot
color = "darkblue",
fill = "lightblue") +
# escalas
scale_y_continuous(expand = c(0,0))+
scale_x_date(
expand = c(0,0), # remove o excesso de espaço do eixo x abaixo e após as barras de casos
date_breaks = "1 month", # Primeiro do mês
date_minor_breaks = "1 month", # Primeiro do mês
label = scales::label_date_short())+ # label formatting
# rótulos e tema
labs(
title = "Utilizando annotate()\nRetângulo e linha mostrando que os dados dos últimos 21 dias são preliminares",
x = "Semana de início de sintomas",
y = "Incidência semanal de casos")+
theme_minimal()+
# adicione um retângulo vermelho semi-transparente nos dados preliminares
annotate(
"rect",
xmin = as.Date(max(central_data$date_onset, na.rm = T) - 21), # a nota precisa ser englobada dentro de as.Date()
xmax = as.Date(Inf), # a nota precisa ser englobada dentro de as.Date()
ymin = 0,
ymax = Inf,
alpha = 0.2, # alfa fácil e intuitivo para ajustar usando annotate()
fill = "red")+
# adicione uma linha preta vertical no topo das outras camadas
annotate(
"segment",
x = max(central_data$date_onset, na.rm = T) - 21, # 21 dias antes dos últimos dados
xend = max(central_data$date_onset, na.rm = T) - 21,
y = 0, # linha inicia em y = 0
yend = Inf, # linha no topo do gráfico
size = 2, # tamanho da linha
color = "black",
lty = "solid")+ # tipo de linha ex.: "solid", "dashed"
# adicione texto no retângulo
annotate(
"text",
x = max(central_data$date_onset, na.rm = T) - 15,
y = 15,
label = "Sujeito à atrasos nas notificações",
angle = 90)A mesma linha preta vertical pode ser obtida com o código abaixo, mas, utilizando geom_vline(), você perde a capacidade de controlar a altura:
geom_vline(xintercept = max(central_data$date_onset, na.rm = T) - 21,
size = 2,
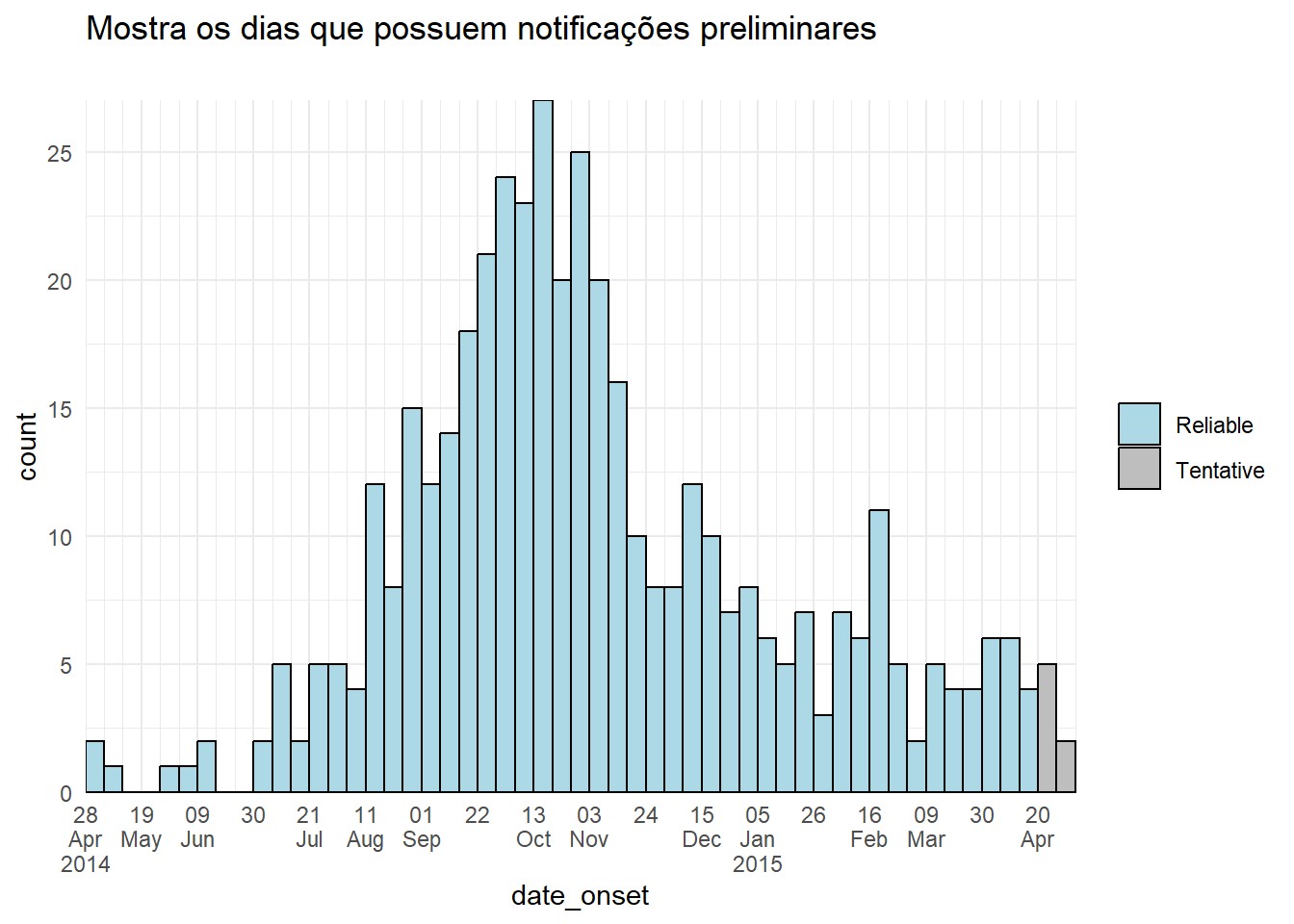
color = "black")Cor das barras
Uma abordagem alternativa pode ser ajustar a cor ou exibição das dos dados preliminares. Você pode criar uma nova coluna no estágio de preparação dos dados, e utiliza-la para agrupar os dados, de forma que o aes(fill = ) dos dados preliminares possa ter uma cor ou alfa diferente das outras barras.
# adiciona uma coluna
############
plot_data <- central_data %>%
mutate(tentative = case_when(
date_onset >= max(date_onset, na.rm=T) - 7 ~ "Tentative", # preliminares e estiverem dentro dos últimos 7 dias
TRUE ~ "Reliable")) # todos os demais são confiáveis
# crie o gráfico
######
ggplot(plot_data, aes(x = date_onset, fill = tentative)) +
# histograma
geom_histogram(
breaks = weekly_breaks_central, # vetor de datas pré-definido, veja o topo da página sobre o ggplot
color = "black") +
# escalas
scale_y_continuous(expand = c(0,0))+
scale_fill_manual(values = c("lightblue", "grey"))+
scale_x_date(
expand = c(0,0), # remove o espaço em excesso do eixo x, abaixo e após as barras de casos
date_breaks = "3 weeks", # segundas a cada 3 semanas
date_minor_breaks = "week", # semanas iniciadas nas segundas
label = scales::label_date_short())+ # label formatting
# rótulos e temas
labs(title = "Mostra os dias que possuem notificações preliminares",
subtitle = "")+
theme_minimal()+
theme(legend.title = element_blank()) # remove o título da legenda32.4 Etiquetas multiníveis de data
Se você quiser etiquetas multiníveis de datas (ex.: mês e ano) sem duplicar as etiquetas de níveis mais baixos, utilize uma das abordagens abaixo:
Lembre - você pode utilizar ferramentas como \n dentro dos argumentos de date_labels ou labels para colocar partes de cada etiqueta em uma nova linha abaixo da atual. Entretanto, o código abaixo o ajuda a colocar anos ou meses (por exemplo) em uma linha abaixo e apenas uma vez. Algumas notas sobre o código abaixo:
- Contagem de casos são agregados em semanas por razões estéticas. Veja a página sobre epicurvas (parte sobre a separação dos dados agregados) para mais detalhes.
- Uma linha
geom_area()é utilizada no lugar de um histograma, uma vez que a abordagem de faceteamento abaixo não funciona bem com histogramas.
Agregue para contagens semanais
# Crie o conjunto de dados de contagens de casos por semana
#######################################
central_weekly <- linelist %>%
filter(hospital == "Central Hospital") %>% # filtre a linelist
mutate(week = lubridate::floor_date(date_onset, unit = "weeks")) %>%
count(week) %>% # faça um resumo das contagens de casos semanais
drop_na(week) %>% # remova os casos sem a informação de onset_date
complete( # preencha todas as semanas com nenhum caso notificado
week = seq.Date(
from = min(week),
to = max(week),
by = "week"),
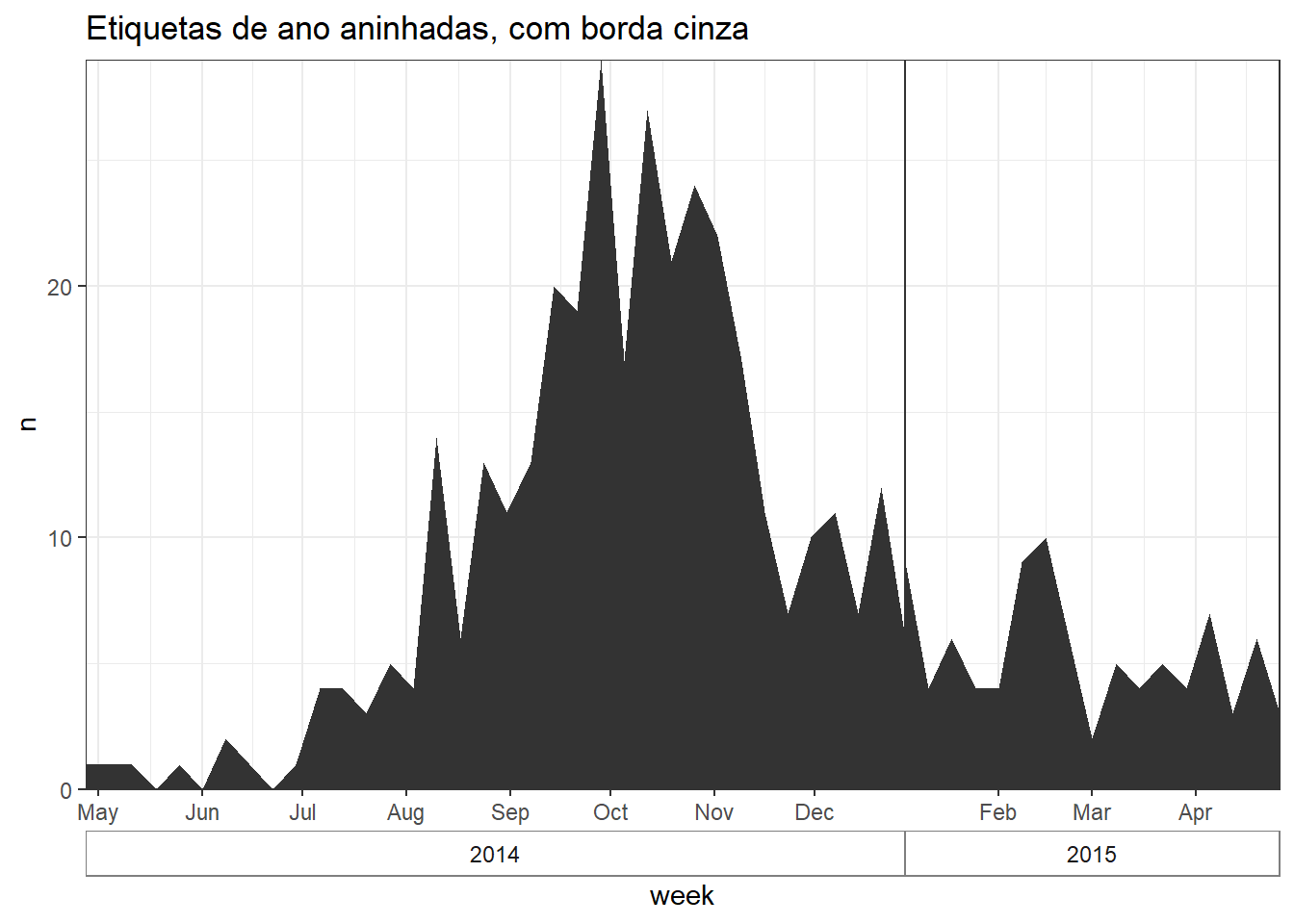
fill = list(n = 0)) # converta novos campos NA (faltantes) para contagens de 0Crie os gráficos
# crie um gráfico com a borda da caixa no ano
##############################
ggplot(central_weekly) +
geom_area(aes(x = week, y = n), # crie uma linha, especificando x e y
stat = "identity") + # a altura da linha é o número da contagem
scale_x_date(date_labels="%b", # formato do rótulo de data mostra o mês
date_breaks="month", # rótulos de data no primeiro dia de cada mês
expand=c(0,0)) + # remove o espaço em excesso em cada extremidade
scale_y_continuous(
expand = c(0,0))+ # remove o espaço em excesso abaixo do eixo x
facet_grid(~lubridate::year(week), # crie uma faceta com o ano (da coluna da classe Date (Data))
space="free_x",
scales="free_x", # eixo x se adapta para o intervalo dos dados (não é fixo)
switch="x") + # etiquetas de faceta (ano) na base
theme_bw() +
theme(strip.placement = "outside", # posicionamento das etiquetas das facetas
strip.background = element_rect(fill = NA, # rótulos das facetas sem preenchimento e com bordas cinzas
colour = "grey50"),
panel.spacing = unit(0, "cm"))+ # sem espaço entre os painéis das facetas
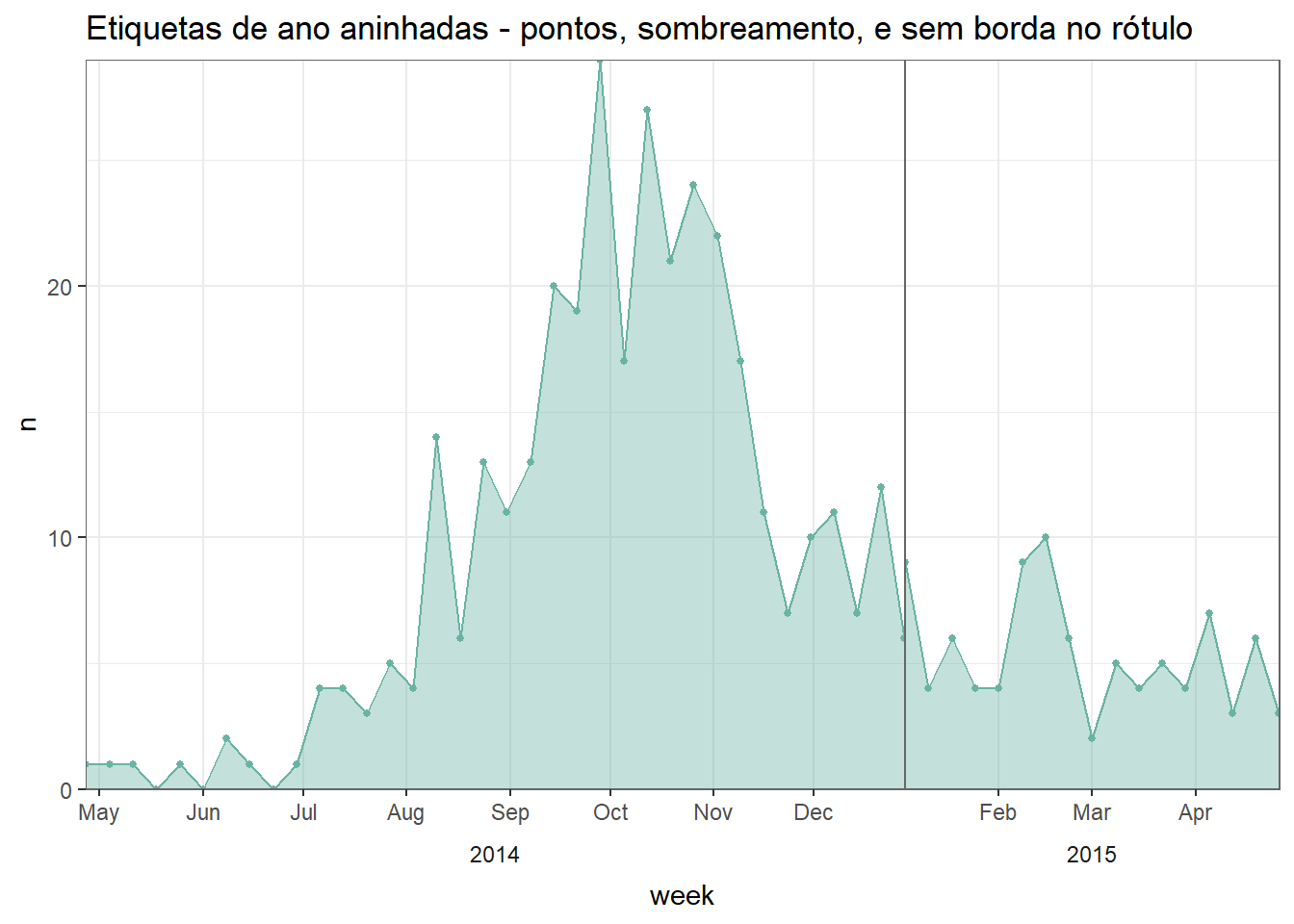
labs(title = "Etiquetas de ano aninhadas, com borda cinza")# crie o gráfico sem a borda de caixa no ano
#################################
ggplot(central_weekly,
aes(x = week, y = n)) + # estabeleça o x e y para o gráfico inteiro
geom_line(stat = "identity", # crie a linha, em que a altura da linha é o número da contagem
color = "#69b3a2") + # cor da linha
geom_point(size=1, color="#69b3a2") + # crie pontos nos pontos de data semanais
geom_area(fill = "#69b3a2", # preencha a área abaixo da linha
alpha = 0.4)+ # preencha a transparência
scale_x_date(date_labels="%b", # formato do rótulo de data mostra o mês
date_breaks="month", # rótulos de data no primeiro dia de cada mês
expand=c(0,0)) + # remova o espaço em excesso
scale_y_continuous(
expand = c(0,0))+ # remova o espaço em excesso abaixo do eixo x
facet_grid(~lubridate::year(week), # faça a faceta no ano (da coluna de classe Date (data))
space="free_x",
scales="free_x", # eixo x se adapta ao intervalo de dados (não é fixo)
switch="x") + # rótulos da faceta (ano) na base
theme_bw() +
theme(strip.placement = "outside", # posicionamento do rótulo da faceta
strip.background = element_blank(), # rótulo da faceta sem fundo
panel.grid.minor.x = element_blank(),
panel.border = element_rect(colour="grey40"), # borda cinza para o painel da faceta
panel.spacing=unit(0,"cm"))+ # sem espaço entre os painéis da faceta
labs(title = "Etiquetas de ano aninhadas - pontos, sombreamento, e sem borda no rótulo")As técnicas acima foram adaptadas desta e desta postagem no stackoverflow.com.
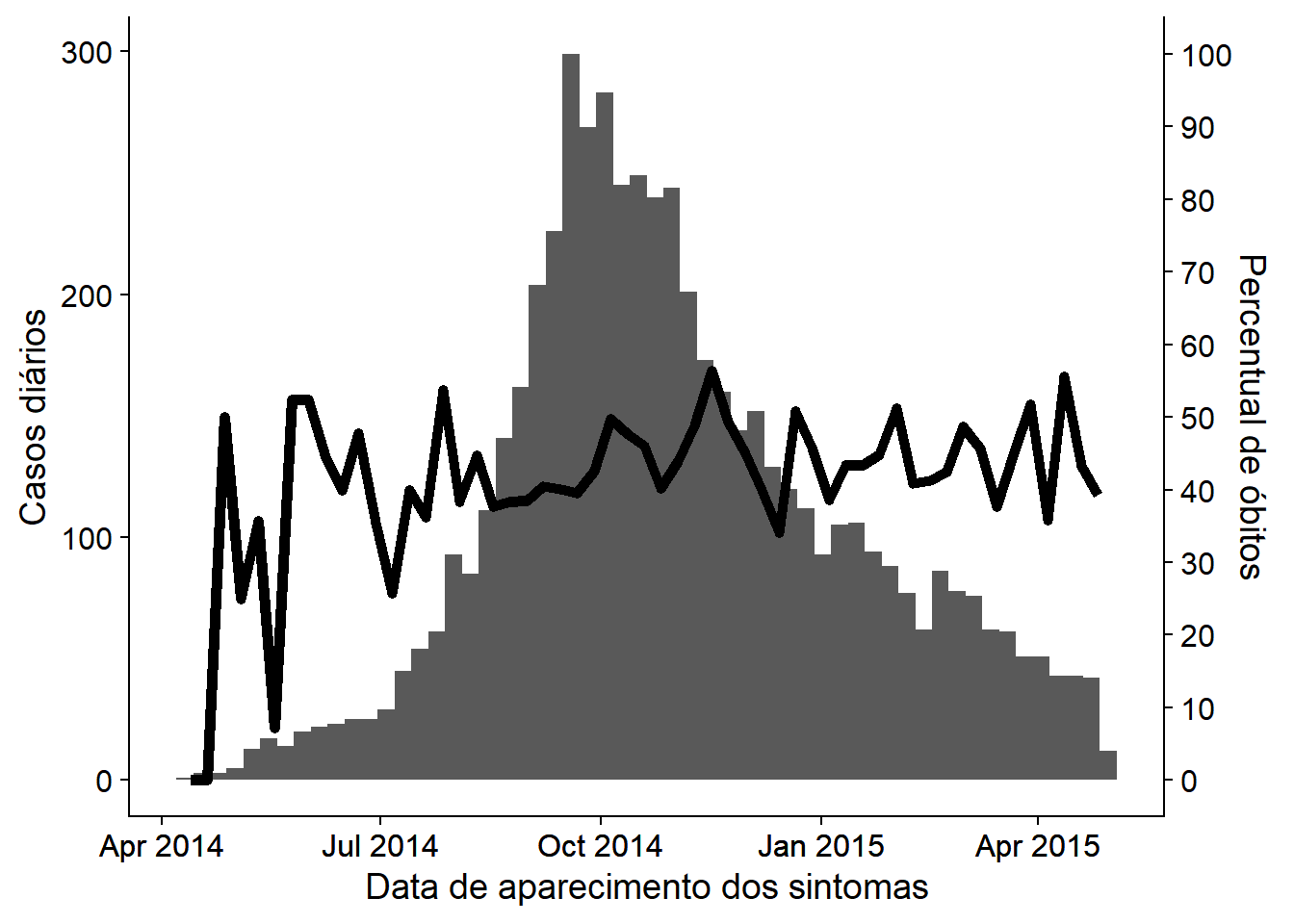
32.5 Eixo duplo
Embora existam discussões intensas sobre a validade de eixos duplos na comunidade de visualização de dados, muitos supervisores de epidemiologia ainda querem ver uma epicurva ou gráfico similar com um percentual sobreposto com um segundo eixo. Isto é discutido mais extensivamente na página dicas do ggplot, mas um exemplo utilizando o método de cowplot é mostrado abaixo:
- Dois gráficos distintos são feitos, e então combinados com o pacote cowplot.
- Os gráficos precisam ter exatamente o mesmo eixo x (ajuste os limites). Do contrário, os dados e rótulos não serão alinhados
- Cada um utiliza
theme_cowplot()e um deles tem o eixo y movido para o lado direito do gráfico
# carregue o pacote
pacman::p_load(cowplot)
# Crie o primeiro gráfico de histograma de uma epicurva
#######################################
plot_cases <- linelist %>%
# crie o gráfico com casos por semana
ggplot()+
# crie o histograma
geom_histogram(
mapping = aes(x = date_onset),
# quebras das classes a cada semana, iniciando na segunda antes do primeiro caso, e indo até a segunda após o último caso
breaks = weekly_breaks_all)+ # vetor pré-definido de datas semanais (veja o topo da seção sobre o ggplot)
# especifique o início e o fim do eixo da data para alinhar com o outro gráfico
scale_x_date(
limits = c(min(weekly_breaks_all), max(weekly_breaks_all)))+ # mín/máx das quebras semanais pré-definidas do histograma
# rótulos
labs(
y = "Casos diários",
x = "Data de aparecimento dos sintomas"
)+
theme_cowplot()
# crie um segundo gráfico com o percentual de óbitos por semana
###########################################
plot_deaths <- linelist %>% # inicie com a linelist
group_by(week = floor_date(date_onset, "week")) %>% # crie a coluna week (semana)
# utilize summarise para obter o percentual semanal de casos que morreram
summarise(n_cases = n(),
died = sum(outcome == "Death", na.rm=T),
pct_died = 100*died/n_cases) %>%
# inicie o gráfico
ggplot()+
# linha com o percentual semanal de óbitos
geom_line( # crie um linha com o percentual de mortos
mapping = aes(x = week, y = pct_died), # especifique o y-altura como sendo da coluna pct_died
stat = "identity", # ajuste a altura da linha para o valor na coluna pct_death, e não a quantidade de linhas (que é o padrão)
size = 2,
color = "black")+
# Mesmos limites no eixo de datas como no outro gráfico - alinhamento perfeito
scale_x_date(
limits = c(min(weekly_breaks_all), max(weekly_breaks_all)))+ # mín/máx das quebras semanais pré-definidas do histograma
# ajustes no eixo y
scale_y_continuous( # ajuste o eixo y
breaks = seq(0,100, 10), # ajuste os intervalos de quebra do eixo da porcentagem
limits = c(0, 100), # ajuste a extensão do eixo de porcentagem
position = "right")+ # mova o eixo de porcentagem para a direita
# rótulo do eixo y, sem rótulo no eixo x
labs(x = "",
y = "Percentual de óbitos")+ # rótulo do eixo de porcentagem
theme_cowplot() # adicione isto para unir os dois gráficosAgora, use o cowplot para sobrepor os dois gráficos. A atenção precisa ser dada ao alinhamento do eixo x, ao lado do eixo y, e ao uso de theme_cowplot().
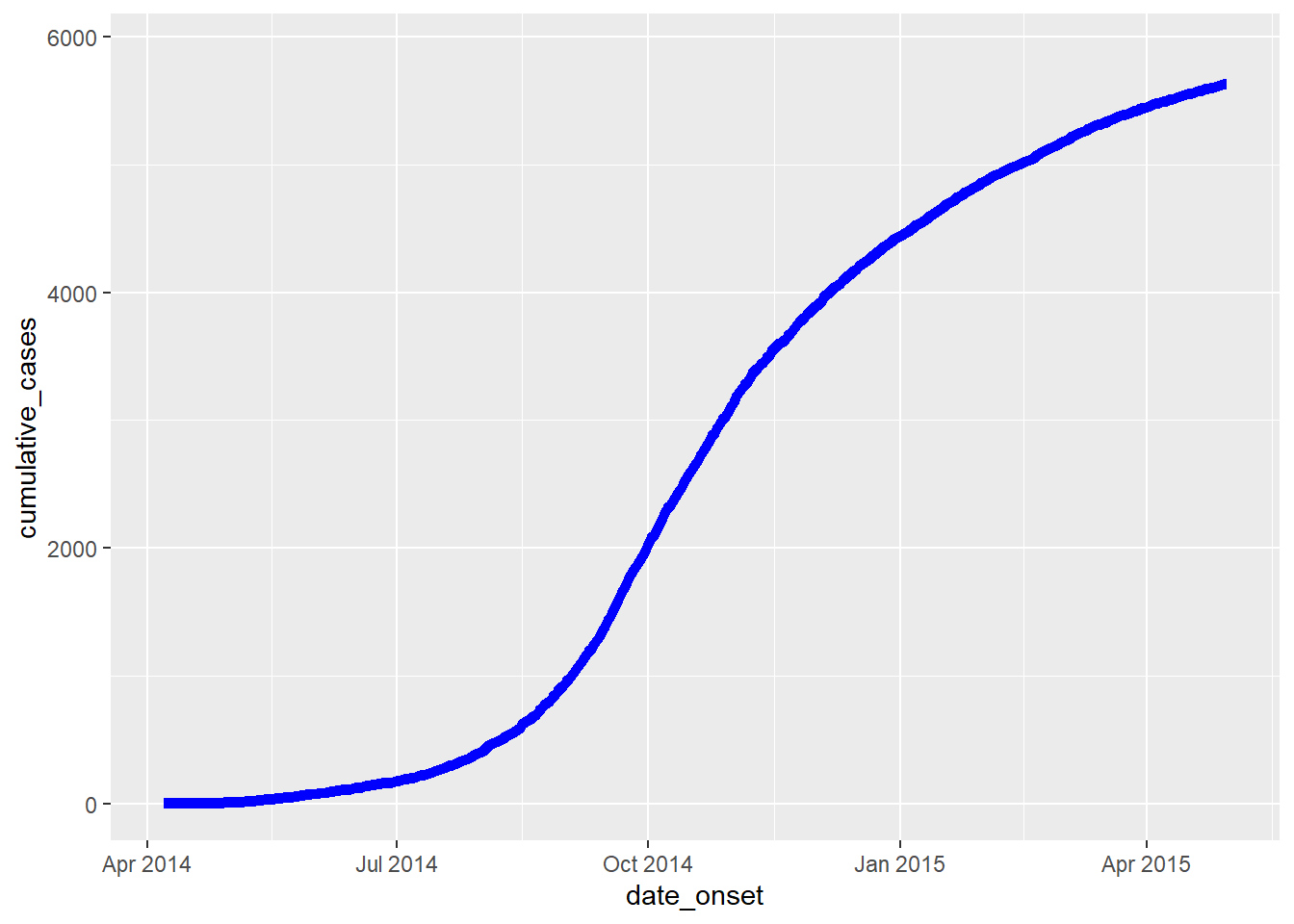
32.6 Incidência acumulada
Nota: Caso esteja utilizando o pacote incidence2, veja a seção sobre como você pode produzir incidência acumulada com uma simples função. Esta página irá ensinar como calcular a incidência acumulada e fazer um gráfico dela utilizando ggplot().
Se estiver iniciando com os casos em uma linelist, crie uma nova coluna contendo o número acumulado de casos por dia em um surto utilizando a função cumsum() do R base:
cumulative_case_counts <- linelist %>%
count(date_onset) %>% # contagem de linhas por dia (salvo na coluna "n")
mutate(
cumulative_cases = cumsum(n) # nova coluna com a quantidade de linhas acumuladas em cada data
)As primeiras 10 linhas são mostradas abaixo:
Esta coluna de casos acumulados pode então ser utilizada para fazer um gráfico com a data de início dos sintomas, date_onset, utilizando geom_line():
plot_cumulative <- ggplot()+
geom_line(
data = cumulative_case_counts,
aes(x = date_onset, y = cumulative_cases),
size = 2,
color = "blue")
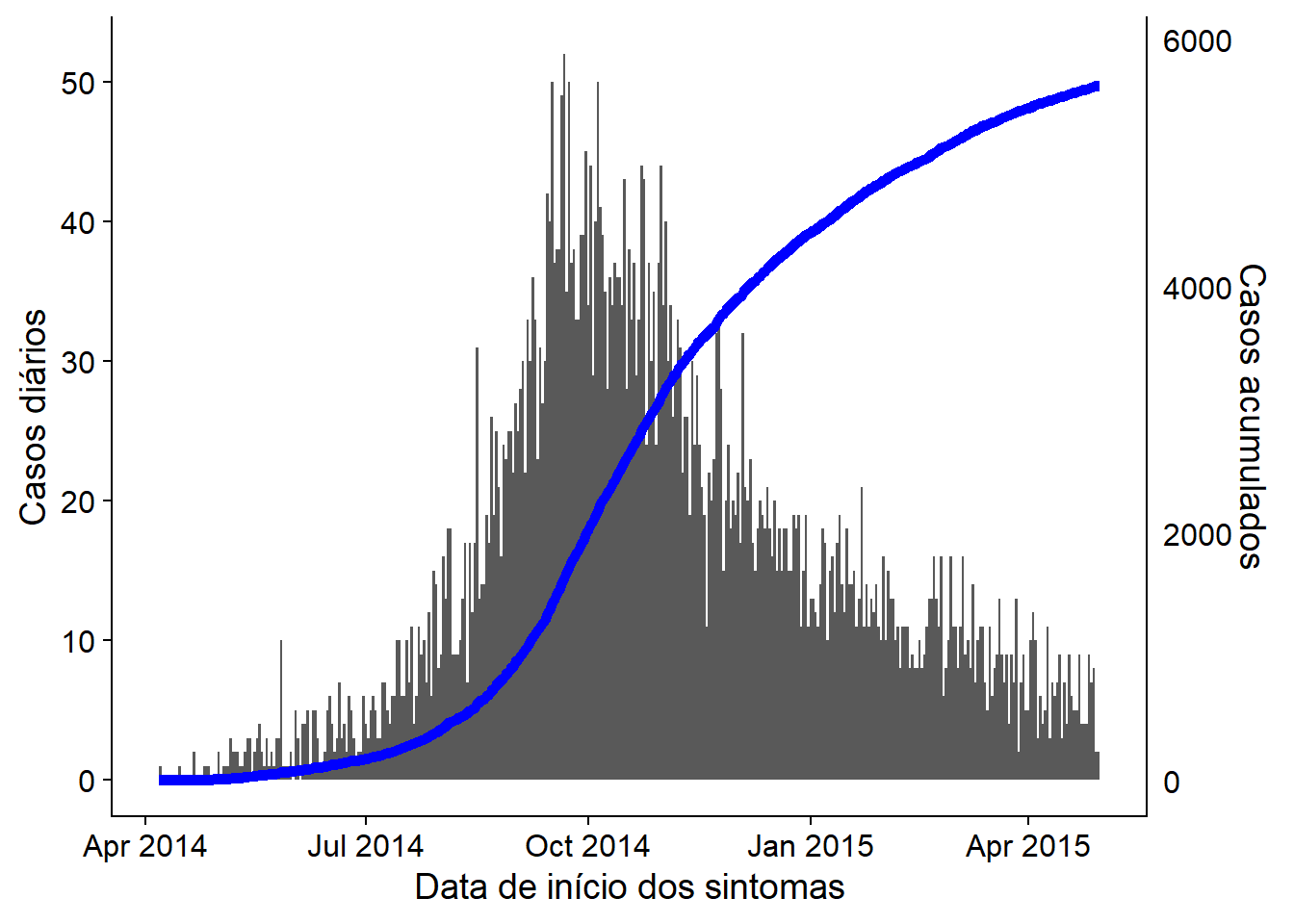
plot_cumulativeEla também pode ser sobreposta em uma epicurva, com dois eixos, utilizando o método cowplot descrito acima e na página dicas do ggplot:
# carregue o pacote
pacman::p_load(cowplot)
# crie primeiro o histograma de uma epicurva
plot_cases <- ggplot()+
geom_histogram(
data = linelist,
aes(x = date_onset),
binwidth = 1)+
labs(
y = "Casos diários",
x = "Data de início dos sintomas"
)+
theme_cowplot()
# crie um segundo gráfico com uma linha de casos acumulados
plot_cumulative <- ggplot()+
geom_line(
data = cumulative_case_counts,
aes(x = date_onset, y = cumulative_cases),
size = 2,
color = "blue")+
scale_y_continuous(
position = "right")+
labs(x = "",
y = "Casos acumulados")+
theme_cowplot()+
theme(
axis.line.x = element_blank(),
axis.text.x = element_blank(),
axis.title.x = element_blank(),
axis.ticks = element_blank())Agora, use cowplot para sobrepor os dois gráficos. Atenção ao alinhamento do eixo x, o lado do eixo y, e o uso de theme_cowplot().