24 Modelagem de epidemias
24.1 Visão geral do tópico
Existe um conjunto crescente de ferramentas para modelagem em epidemiologia que nos permite desenvolver análises complexas com esforço mínimo. Este capítulo apresenta uma síntese sobre como usar essas ferramentas para:
estimar o número efetivo de reprodução Rt e estatísticas relacionadas como tempo de duplicação
produzir projeções de curto prazo da incidência futura
Tenha em mente que esta página não é uma revisão das metodologias e métodos estatísticos empregados por estas ferramentas. Para tanto, utilize os links disponíveis no subtópico Recursos extras para encontrar artigos detalhando essas metodologias. Antes de utilizar as ferramentas a seguir, garanta que você compreenda os métodos subjacentes empregados; isto garantirá que você possa interpretar adequadamente os resultados.
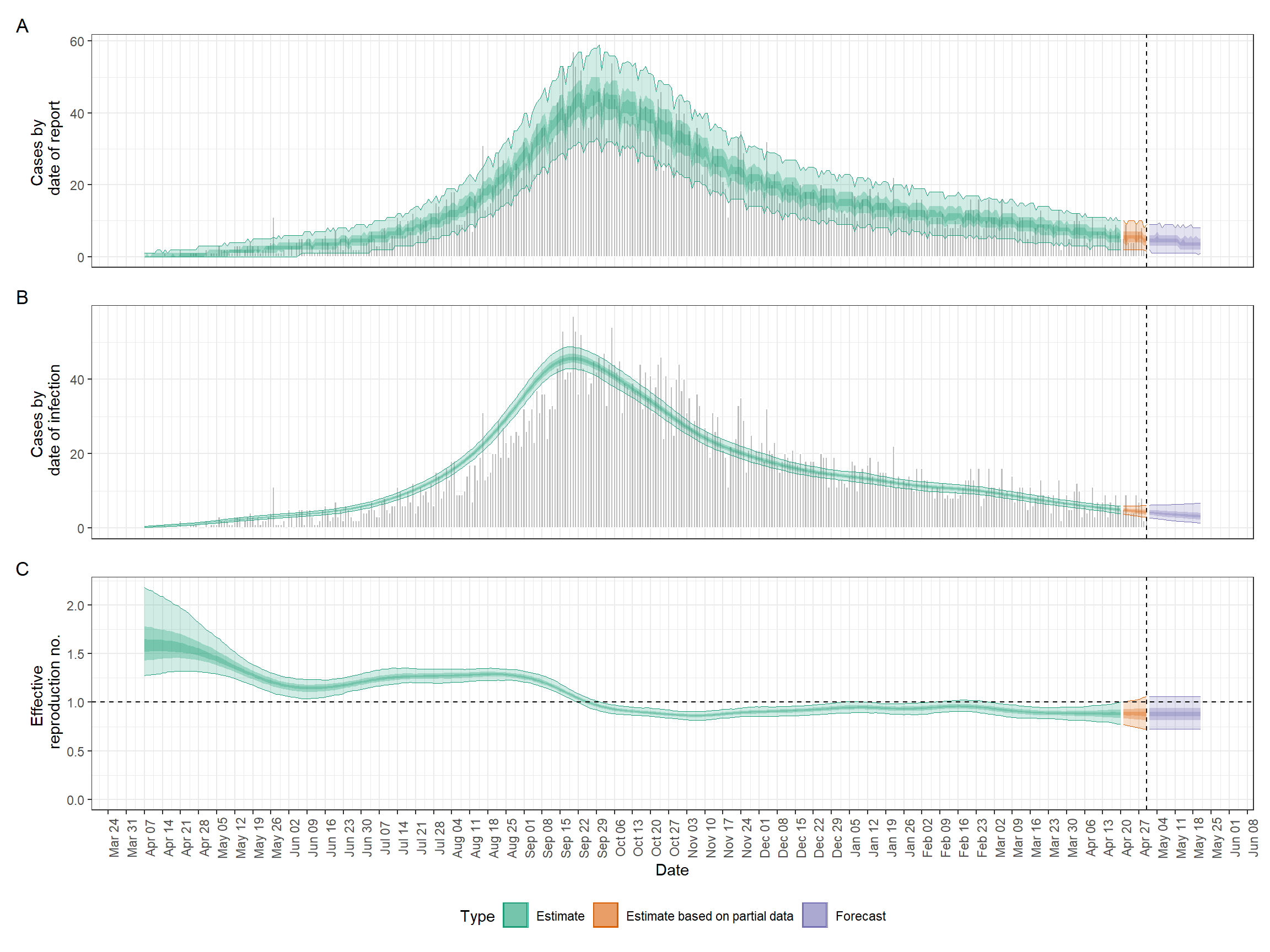
Abaixo está um exemplo de uma das análises que construíremos neste capítulo.
24.2 Preparação
Iremos utilizar dois métodos e pacotes diferentes para estimar o Rt, chamados de EpiNow e EpiEstim, assim como o pacote projections para fazer previsões da incidência de casos.
Este pedaço de código mostra o carregamento dos pacotes necessários para as análises. Neste manual, enfatizamos o uso de p_load(), do pacote pacman, que instala o pacote, caso necessário, e o inicia para uso. Você também pode carregar pacotes instalados com library(), do R base. Veja a página sobre Introdução ao R para mais informações sobre pacotes no R.
pacman::p_load(
rio, # Importar arquivos
here, # Localizar arquivos
tidyverse, # Gerenciamento dos dados + gráficos ggplot2
epicontacts, # Analisar as redes de transmissão
EpiNow2, # Estimar o Rt
EpiEstim, # Estimar Rt
projections, # Projeções da incidência
incidence2, # Trabalhando com dados de incidência
epitrix, # Funções uteis de epi
distcrete # Distribuições discretas .;
)Nesta seção, iremos utilizar a linelist dos casos limpa para todas as análises. Se você quiser acomapnhar, clique para baixar a linelist “limpa” (como arquivo .rds). Veja a página Download do manual e dados para baixar todos os dados utilizados como exemplo neste manual.
# importe a linelist limpa
linelist <- import("linelist_cleaned.rds")24.3 Estimando o Rt
EpiNow2 vs. EpiEstim
O número de reprodução R é uma medida da capacidade de transmissão de uma doença e é definido como a quantidade esperada de casos secundários para cada caso infectado. Em uma população totalmente susceptível, este valor representa o número básico de reprodução R0. Entretanto, conforme o número de indivíduos susceptíveis em uma população muda no decorrer de um surto ou pandemia, e conforme várias medidas de resposta e controle são implementadas, a medida mais comumente utilizada de transmissibilidade é o número efetivo de reprodução Rt; este é definido como a quantidade de casos secundários por cada caso infectado em um determinando ponto no tempo t.
O pacote EpiNow2 fornece a estrutura mais sofisticada para estimar o Rt. Ele tem duas vantagens chave sobre o outro pacote comumente utilizado, EpiEstim:
- Ele leva em consideração as demoras nas notificações ao estimar o Rt, mesmo quando dados recentes são incompletos.
- Ele estima o Rt a partir das datas de infecção, em vez das datas de início das notificações, o que significa que o efeito de uma intervenção irá imediatamente refletir em mudanças no Rt, em vez de demorar para alterar.
Entretanto, ele também possuí duas desvantagens chave:
- Ele necessita de conhecimento da distribuição do tempo de geração (ex.: distribuição dos intervalos de infecção entre casos primários e secundários), distribuição do tempo de incubação (ex.: distribuição dos intervalos entre a infecção e o início dos sintomas) e qualquer outra distribuição de intervalos relevante para os seus dados (ex.: se você tiver datas de notificação, você precisa da distribuição dos intervalos entre início dos sintomas e notificação dos casos). Enquanto isto irá permitir estimativas mais acuradas do Rt, EpiEstim apenas requer a distribuição seriada dos intervalos (ex.: a distribuição de intervalos entre o início dos sintomas de casos primários e secundários), que pode ser a única distribuição disponível para você.
- EpiNow2 é significamente mais devagar do que EpiEstim, por fatores entre 100-1000 mais lento! Por exemplo, estimar o Rt para a amostra do surto trabalhada nesta seção levou por volta de quatro horas (esta estimativa rodou por um elevado número de iterações para garantir elevada acurácia, e provavelmente poderia ser reduzida caso necessário. Entretanto, o ponto é que este algoritmo é mais devagar, no geral). Logo, este pacote pode ser inviável caso você esteja atualizando regularmente suas estimativas do Rt.
Qual pacote você irá escolher irá depender dos seus dados, tempo e recursos computacionais disponíveis.
EpiNow2
Estimando a distribuição dos intervalos
As distribuições dos intervalos necessárias para utilizar o EpiNow2 variam de acordo com os seus dados. Essencialmente, você precisar ser capaz de descrever o intervalo entre a data de infecção e a data do evento que você quer utilizar para estimar o Rt. Caso você esteha utilizando as datas de início dos sintomas, isto seria simplesmente a distribuição do período de incubação. Se você estiver utilizando as datas de notificação, você utiliza o intervalo entre infecção à notificação. Como é improvável que esta distribuição seja conhecida diretamente, EpiNow2 permite conectar múltiplas distribuições de intervalo; neste caso, os intervalos entre a infecção e o aparecimento dos sintomas (ex.: o período de incubação, que provavelmente é conhecido) e entre o início dos sintomas e a notificação (que você pode frequentemente estimar a partir dos seus dados).
Como temos as datas de início dos sintomas para todos os nossos casos em nossa linelist de exemplo, nós apenas precisamos da distribuição do período de incubação para conectar os nossos dados (ex.: datas de início dos sintomas) para a data de infecção. Nós podemos ou estimar esta distribuição a partir dos dados, ou utilizar valores da literatura.
Uma estimativa do período de incubação da Ebola encontrada na literatura (obtida deste artigo) possuí uma média de 9.1, desvio padrão de 7.3, e o valor máximo de 30. Isto pode ser especificado no R como mostrado a seguir:
incubation_period_lit <- list(
mean = log(9.1),
mean_sd = log(0.1),
sd = log(7.3),
sd_sd = log(0.1),
max = 30
)Observe que o EpiNow2 pede que a distribuição destes intervalos seja fornecida em uma escala log (logarítmica), por isso chamamos log ao redor de cada valor (exceto o parâmetro max que, de forma confusa, precisa ser fornecido em uma escala natural). Os mean_sd e sd_sd definem o desvio padrão da média e as estimativas do desvio padrão. Como neste caso estes valores não são conhecidos, nós escolhemos um valor bastante arbitrário, 0.1.
Nesta análise, nós estimamos a distribuição do período de incubação a partir da própria linelist utilizando a função bootstrapped_dist_fit, que irá ajustar uma distribuição log-normal para os intervalos observados entre a infecção e o aparecimento dos sintomas na linelist.
## estime o período de incubação
incubation_period <- bootstrapped_dist_fit(
linelist$date_onset - linelist$date_infection,
dist = "lognormal",
max_value = 100,
bootstraps = 1
)A outra distribuição que precisamos é o tempo de geração. Como temos dados sober os tempos de infecção and os links de transmissão, nós podemos estimar esta distribuição a partir da linelist ao calcular o intervalo entre o tempo de infecção de pares infectores-infectados. Para fazer isto, nós utilizamos a função get_pairwise do pacote epicontacts, que nos permite calcular diferenças entre os pares a partir das propriedades da linelist sobre os pares de transmissão. Nós primeiro criamos um objeto epicontact (veja a página Cadeias de transmissão para mais detalhes):
## gere o objeto contatos
contacts <- linelist %>%
transmute(
from = infector,
to = case_id
) %>%
drop_na()
## gere o objeto epicontact
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
directed = TRUE
)Então, ajustamos a diferença no tempo de incubação entre os pares da transmissão, calculado com get_pairwise, em uma distribuição gamma:
## estime o tempo de geração gamma
generation_time <- bootstrapped_dist_fit(
get_pairwise(epic, "date_infection"),
dist = "gamma",
max_value = 20,
bootstraps = 1
)Executando o EpiNow2
Agora nós só precisamos calcular a incidência diária a partir da linelist, que podemos fazer facilmente com as funções group_by() e n(), do dplyr. Note que o EpiNow2 requer que os nomes das colunas sejam date e confirm.
## obtenha a incidência a partir da data de início dos sintomas
cases <- linelist %>%
group_by(date = date_onset) %>%
summarise(confirm = n())Podemos, então, estimar o Rt utilizando a função epinow. Algumas notas sobre os dados usados:
- Nós podemos fornecer qualquer quantidade de distribuições de intervalos ‘encadeados’ para o argumento
delays; simplesmente iríamos inserí-los junto com o objetoincubation_perioddentro da funçãodelay_opts. -
return_outputgarante que o resultado da análise é obtido dentro do R, e não apenas salvo em um arquivo. -
verboseespecifica que queremos uma leitura/update do progresso. -
horizonindica em quantos dias queremos estimar a incidência futura. - Nós adicionamos outras opções no argumento
stanpara especificar por quanto tempo queremos executar a inferência. Aumentar osamplesechainsirá te dar uma estimativa mais precisa que melhor caracteriza a incerteza, entretanto irá demorar mais para ser calculada.
## execute o epinow
epinow_res <- epinow(
reported_cases = cases,
generation_time = generation_time,
delays = delay_opts(incubation_period),
return_output = TRUE,
verbose = TRUE,
horizon = 21,
stan = stan_opts(samples = 750, chains = 4)
)Analisando o resultado da análise
Assim que o código terminar de ser executado, nós podemos criar um resumo da análise facilmente, da seguinte maneira. Role a imagem para ver a sua real extensão.
Nós também podemos olher diferentes resumos estatísticos:
## tabela resumo
epinow_res$summary measure estimate
<char> <char>
1: New confirmed cases by infection date 4 (2 -- 6)
2: Expected change in daily cases Unsure
3: Effective reproduction no. 0.88 (0.73 -- 1.1)
4: Rate of growth -0.012 (-0.028 -- 0.0052)
5: Doubling/halving time (days) -60 (130 -- -25)
numeric_estimate
<list>
1: <data.table[1x9]>
2: 0.56
3: <data.table[1x9]>
4: <data.table[1x9]>
5: <data.table[1x9]>Para mais análise e customização do gráfico, você pode acessar as estimativas diárias resumidas através de $estimates$summarised. Nós iremos converter isto do padrão data.table para um tibble, facilitando o uso com dplyr.
## extraia o resumo e converta para formato tibble
estimates <- as_tibble(epinow_res$estimates$summarised)
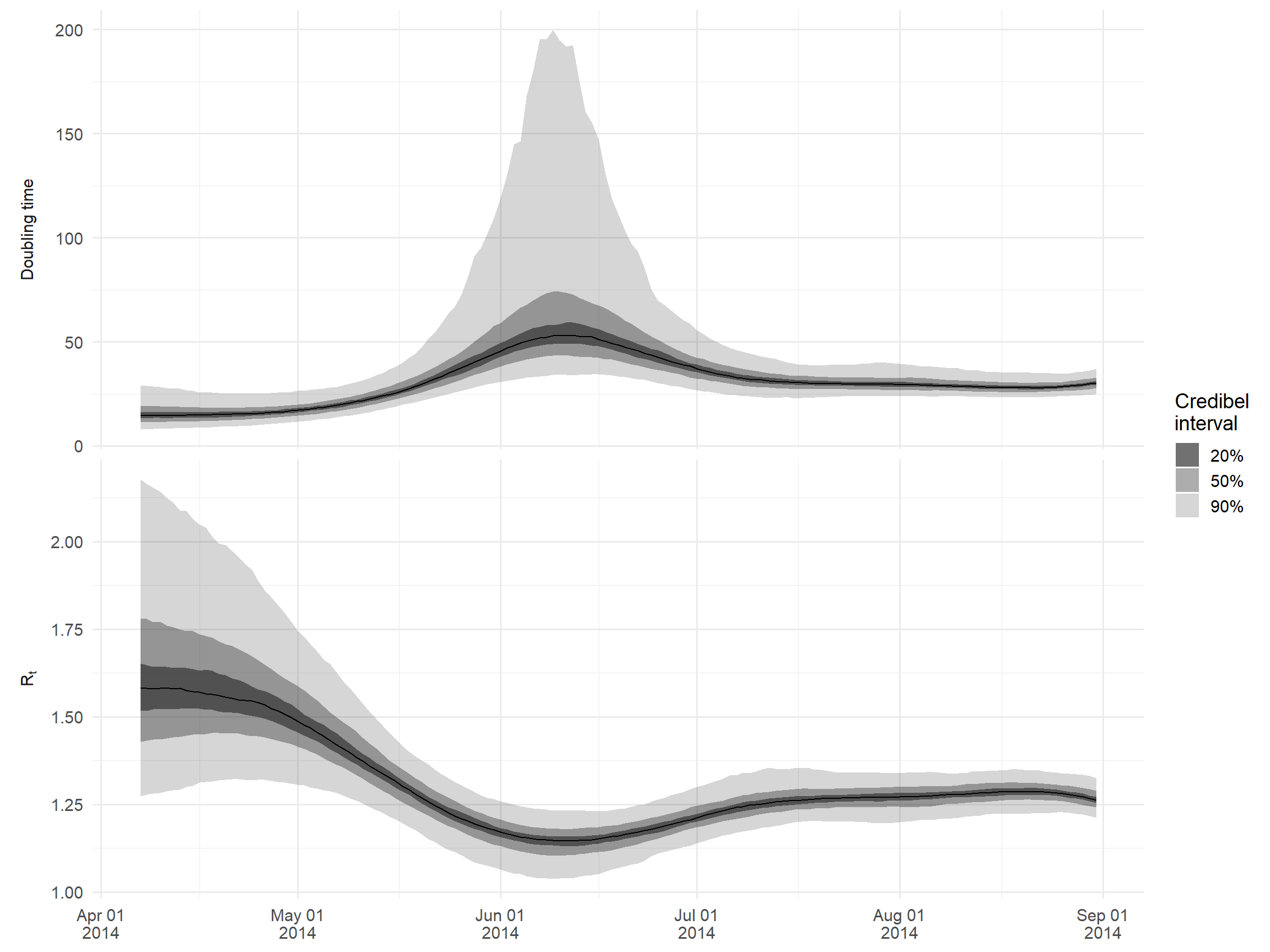
estimatesComo um exemplo, vamos criar um gráfico do tempo de duplicação e do Rt. Nós iremos apenas olhar os primeiros meses do surto, quando o Rt está bem acima de um, para evitar traçar tempos de duplicação extremamente elevados.
Nós utilizamos a fórmula log(2)/growth_rate para calcular o tempo de duplicação a partir da taxa de crescimento estimada.
## crie amplas df para um gráfico mediano
df_wide <- estimates %>%
filter(
variable %in% c("growth_rate", "R"),
date < as.Date("2014-09-01")
) %>%
## converta as taxas de crescimento para o tempo de duplicação
mutate(
across(
c(median, lower_90:upper_90),
~ case_when(
variable == "growth_rate" ~ log(2)/.x,
TRUE ~ .x
)
),
## renomeie a variável para refletir na transformação de taxa de crescimento para tempo de duplicação
variable = replace(variable, variable == "growth_rate", "doubling_time")
)
## crie um data frame longo para criar gráfico de quantis
df_long <- df_wide %>%
## aqui nós combinamos quantis correspondentes (ex.: lower_90 para upper_90)
pivot_longer(
lower_90:upper_90,
names_to = c(".value", "quantile"),
names_pattern = "(.+)_(.+)"
)
## crie um gráfico
ggplot() +
geom_ribbon(
data = df_long,
aes(x = date, ymin = lower, ymax = upper, alpha = quantile),
color = NA
) +
geom_line(
data = df_wide,
aes(x = date, y = median)
) +
## utilize label_parsed para conseguir subscrever o rótulo
facet_wrap(
~ variable,
ncol = 1,
scales = "free_y",
labeller = as_labeller(c(R = "R[t]", doubling_time = "Doubling~time"), label_parsed),
strip.position = 'left'
) +
## defina manualmente a transparência do quantil
scale_alpha_manual(
values = c(`20` = 0.7, `50` = 0.4, `90` = 0.2),
labels = function(x) paste0(x, "%")
) +
labs(
x = NULL,
y = NULL,
alpha = "Credibel\ninterval"
) +
scale_x_date(
date_breaks = "1 month",
date_labels = "%b %d\n%Y"
) +
theme_minimal(base_size = 14) +
theme(
strip.background = element_blank(),
strip.placement = 'outside'
)EpiEstim
Para executar EpiEstim, nós precisamos fornecer dados de incidência diária, e especificar o intervalo seriado (i.e.: a distribuição dos intervalos entre o início dos sintomas dos casos primários e secundários).
Dados de incidência podem ser fornecidos para o EpiEstim como um vetor, um quadro de dados, ou um objeto incidence do pacote original incidence. Você consegue até distinguir entre importados e infecções adquiridas localmente; veja a documentação em ?estimate_R para mais detalhes.
Nós iremos criar a entrada de dados usando incidence2. Veja a página sobre Curvas epidêmicas para mais exemplos com o pacote incidence2. Já que existem updates no pacote incidence2 que não fornecem a entrada necessária do estimateR(), existem algumas pequenas etapas adicionais necessárias. O objeto incidence consiste de uma tabela tibble com as datas e as respectivas contagens. Nós usamos complete(), do pacote tidyr, para garantir que todas as datas sejam incluídas (até as datas sem casos), e então rename() as colunas para gerarem o que é esperado pela função estimate_R() em uma etapa posterior.
## obtenha a incidência a partir da data de início dos sintomas
cases <- incidence2::incidence(linelist, date_index = "date_onset") %>% # obtenha a quantidade de casos por dia
tidyr::complete(date_index = seq.Date( # garanta que todas as datas estão presentes
from = min(date_index, na.rm = T),
to = max(date_index, na.rm=T),
by = "day"),
fill = list(count = 0)) %>% # converta contagens NA para 0
rename(I = count, # renomeie para os nomes utilizados no estimateR
dates = date_index)O pacote fornece diferentes opções para especificar os intervalos seriados, os detalhes são fornecidos na documentação em ?estimate_R. Nós iremos cobrir duas das opções aqui.
Utilizando estimativas de intervalos seriados da literatura
Ao usar a opção method = "parametric_si", podemos especificar manualmente a média e desvio padrão do intervalo seriado em um objeto config criado usando a função make_config. Nós usamos uma média e um desvio padrão de 12.0 e 5.2, respectivamente, definidos neste artigo:
## crie o config
config_lit <- make_config(
mean_si = 12.0,
std_si = 5.2
)Então, nós podemos estimar o Rt com a função estimate_R:
cases <- cases %>%
filter(!is.na(date))
#create a dataframe for the function estimate_R()
cases_incidence <- data.frame(dates = seq.Date(from = min(cases$dates),
to = max(cases$dates),
by = 1))
cases_incidence <- left_join(cases_incidence, cases) %>%
select(dates, I) %>%
mutate(I = ifelse(is.na(I), 0, I))Joining with `by = join_by(dates)`epiestim_res_lit <- estimate_R(
incid = cases_incidence,
method = "parametric_si",
config = config_lit
)Default config will estimate R on weekly sliding windows.
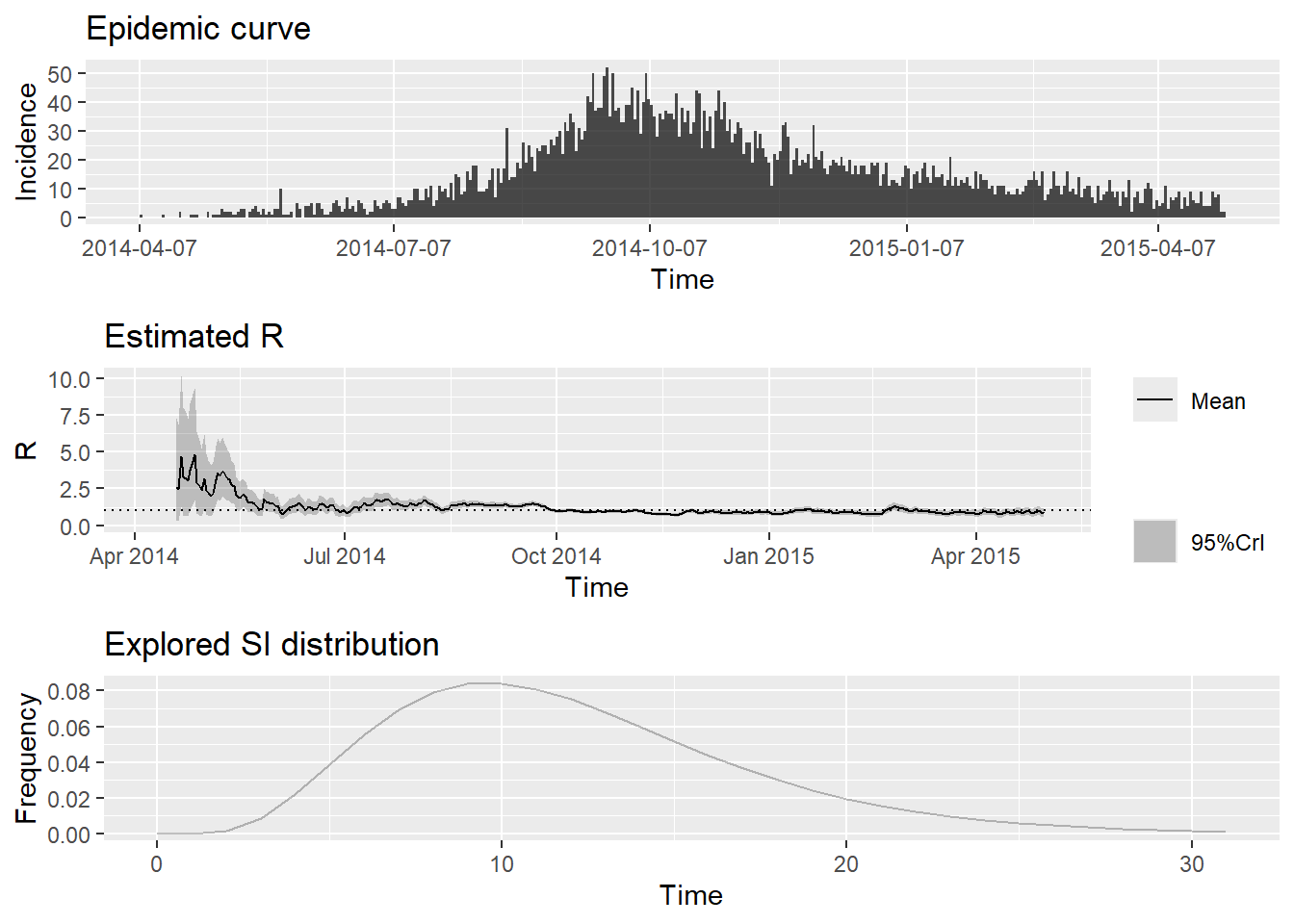
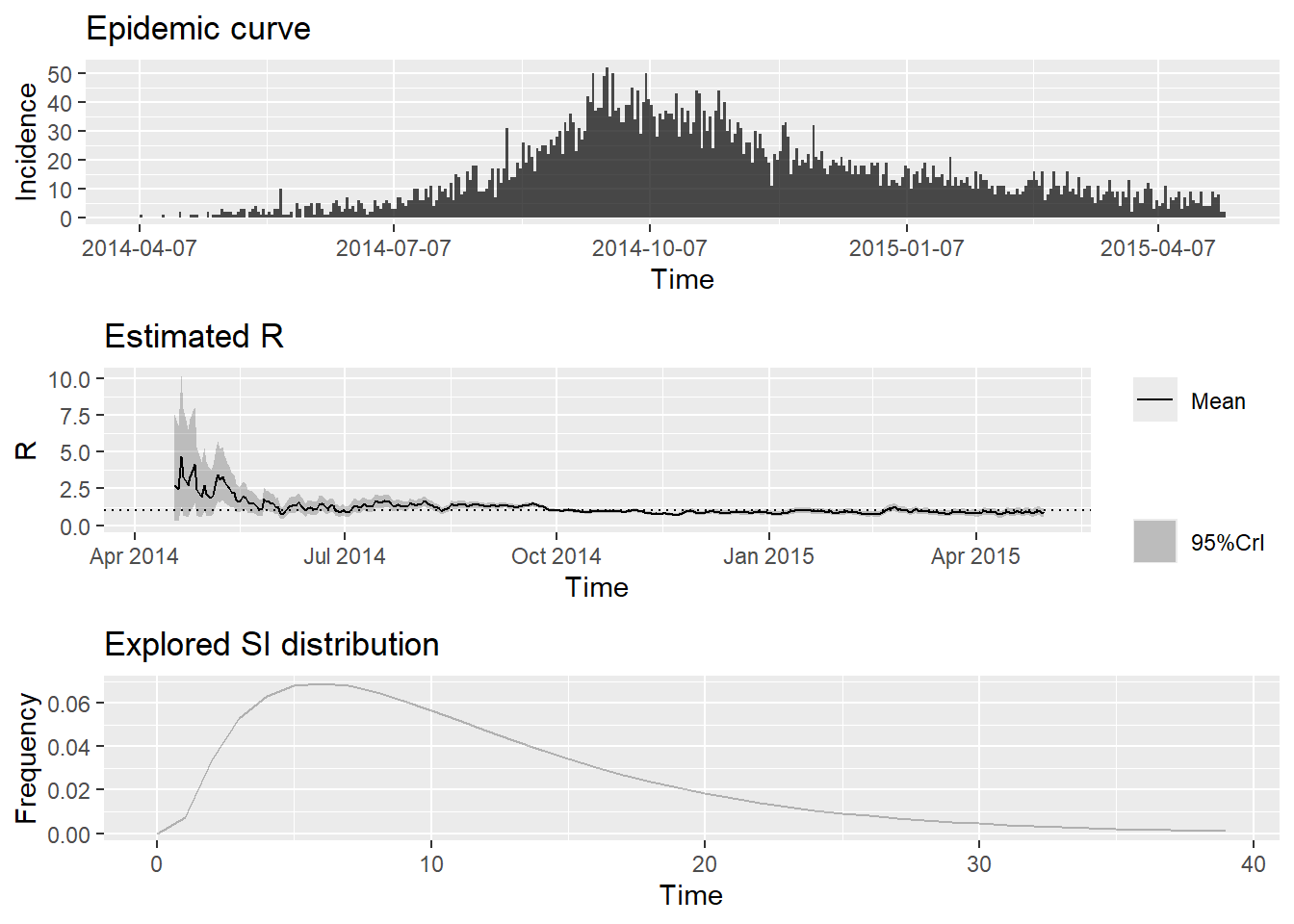
To change this change the t_start and t_end arguments. e criar um gráfico resumindo os resultados da análise:
Utilizando estimativas de intervalos seriados dos dados
Conforme obtemos dados sobre as datas de início dos sintomas and e links de transmissão, nós podemos também estimar o intervalo seriado a partir da linelist ao calcular o intervalo entre as datas de início dos sintomas dos pares infectante-infectado.Como fizemos na seção do EpiNow2, nós iremos agora utilizar a função get_pairwise, do pacote epicontacts, que nos permite calcular as diferenças entre os pares de transmissão nas características na linelsit. Primeiro, criamos um objeto epicontact (veja a página Cadeias de transmissão para mais detalhes):
## gere os contatos
contacts <- linelist %>%
transmute(
from = infector,
to = case_id
) %>%
drop_na()
## gere um objeto epicontact
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
directed = TRUE
)Então ajustamos a diferença entre as datas de início dos sintomas dos pares de transmissão, calculado usando get_pairwise, para uma distribuição gamma. Utilizamos a função fit_disc_gamma, do pacote epitrix, para fazer este procedimento de ajuste, uma vez que precisamos de uma distribuição discreta.
## estime o intervalo seriado gamma
serial_interval <- fit_disc_gamma(get_pairwise(epic, "date_onset"))Então aplicamos esta informação no objeto config, executamos o EpiEstim novamente, e criamos um gráfico dos resultados:
## crie o config
config_emp <- make_config(
mean_si = serial_interval$mu,
std_si = serial_interval$sd
)
## execute o epiestim
epiestim_res_emp <- estimate_R(
incid = cases_incidence,
method = "parametric_si",
config = config_emp
)Default config will estimate R on weekly sliding windows.
To change this change the t_start and t_end arguments. ## crie um gráfico dos resultados
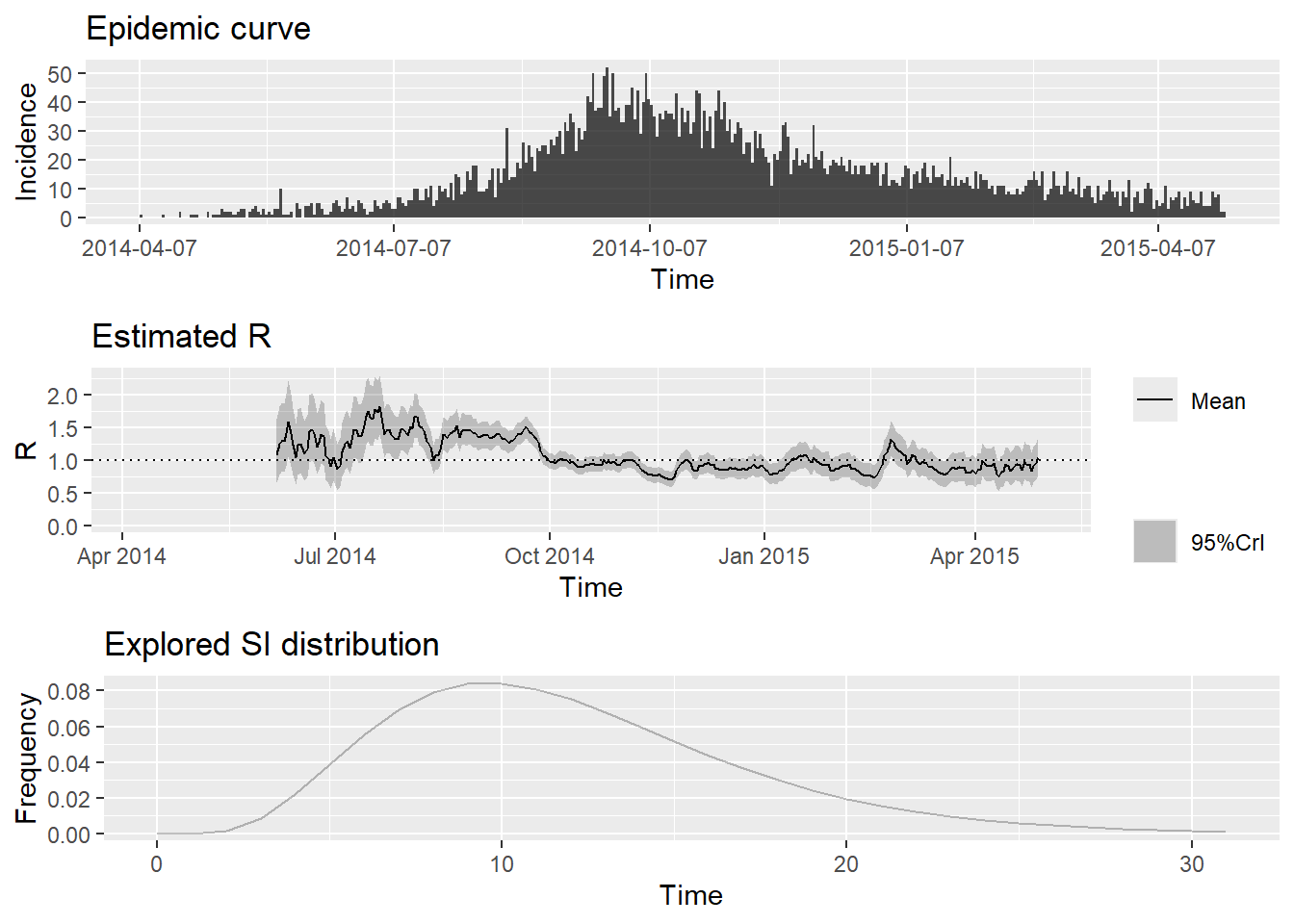
plot(epiestim_res_emp)Especificando as janelas de estimação do tempo
Estas opções padrão irão fornecer uma estimativa semanal móvel, e podem atuar como um aviso que você está estimando o Rt mutio precocemente no surto, para uma estimativa precisa. Você pode mudar isto ao ajustar uma data de início posterior para a estimativa, como mostrado abaixo. Infelizmente, o EpiEstim apenas fornece um método bem desajeitado de especificar estes tempos de estimativas, em que você precisa fornecer um vetor de números inteiros referentes as datas de início e fim para cada intervalo de tempo.
## defina um vetor de datas iniciando em 1o de junho
start_dates <- seq.Date(
as.Date("2014-06-01"),
max(cases$dates) - 7,
by = 1
) %>%
## substraia a data de início para converto para numérico
`-`(min(cases$dates)) %>%
## converta para número inteiro
as.integer()
## adicione seis dias para um intervalo móvel de uma semana
end_dates <- start_dates + 6
## crie o config
config_partial <- make_config(
mean_si = 12.0,
std_si = 5.2,
t_start = start_dates,
t_end = end_dates
)Agora re-executamos o EpiEstim, e podemos ver as estimativas apenas a partir de junho:
Analisando os resultados
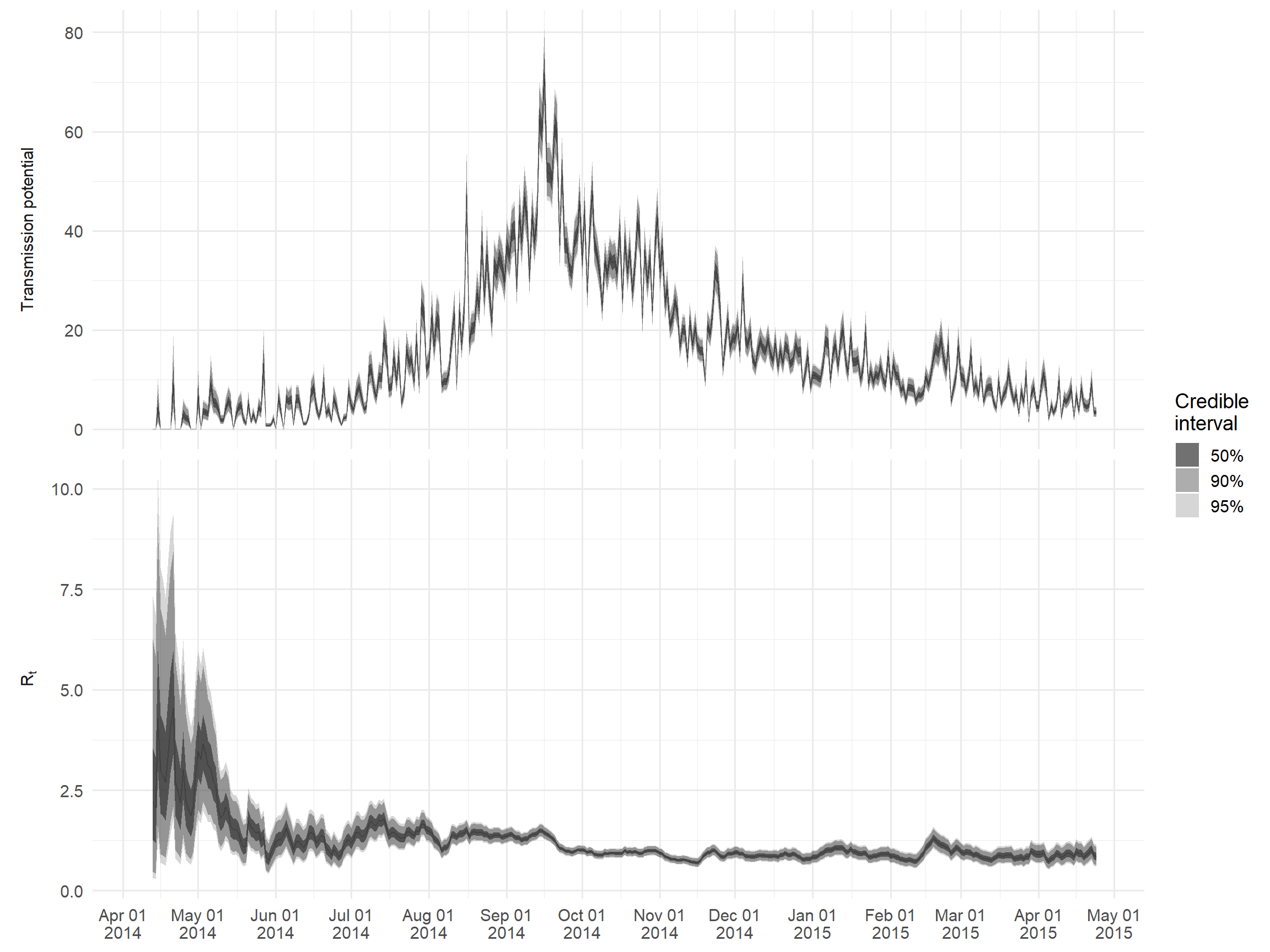
Os principais resultados podem ser acessados através de $R. Como um exemplo, nós iremos criar um gráfico do Rt e uma medida de “potencial de transmissão”, dada pelo produto de Rt e o número de casos notificados naquele dia; isto representa o número esperado de casos na próxima geração da infecção.
## crie um quadro de dados amplo para a mediana
df_wide <- epiestim_res_lit$R %>%
rename_all(clean_labels) %>%
rename(
lower_95_r = quantile_0_025_r,
lower_90_r = quantile_0_05_r,
lower_50_r = quantile_0_25_r,
upper_50_r = quantile_0_75_r,
upper_90_r = quantile_0_95_r,
upper_95_r = quantile_0_975_r,
) %>%
mutate(
## extraia a data média de t_start e t_end
dates = epiestim_res_emp$dates[round(map2_dbl(t_start, t_end, median))],
var = "R[t]"
) %>%
## una com os dados de incidência diária
left_join(cases, "dates") %>%
## calcule o risco através de todas as estimativas de r
mutate(
across(
lower_95_r:upper_95_r,
~ .x*I,
.names = "{str_replace(.col, '_r', '_risk')}"
)
) %>%
## separe as estimativas de r e as estimativas de risco
pivot_longer(
contains("median"),
names_to = c(".value", "variable"),
names_pattern = "(.+)_(.+)"
) %>%
## atribua os níveis do fator
mutate(variable = factor(variable, c("risk", "r")))
## crie um data frame longo a partir dos quantis
df_long <- df_wide %>%
select(-variable, -median) %>%
## separe o r/estimativas de risco e níveis de quantis
pivot_longer(
contains(c("lower", "upper")),
names_to = c(".value", "quantile", "variable"),
names_pattern = "(.+)_(.+)_(.+)"
) %>%
mutate(variable = factor(variable, c("risk", "r")))
## crie o gráfico
ggplot() +
geom_ribbon(
data = df_long,
aes(x = dates, ymin = lower, ymax = upper, alpha = quantile),
color = NA
) +
geom_line(
data = df_wide,
aes(x = dates, y = median),
alpha = 0.2
) +
## use label_parsed para permitir rótulos subescritos
facet_wrap(
~ variable,
ncol = 1,
scales = "free_y",
labeller = as_labeller(c(r = "R[t]", risk = "Transmission~potential"), label_parsed),
strip.position = 'left'
) +
## defina manualmente a transparência do quantil
scale_alpha_manual(
values = c(`50` = 0.7, `90` = 0.4, `95` = 0.2),
labels = function(x) paste0(x, "%")
) +
labs(
x = NULL,
y = NULL,
alpha = "Credible\ninterval"
) +
scale_x_date(
date_breaks = "1 month",
date_labels = "%b %d\n%Y"
) +
theme_minimal(base_size = 14) +
theme(
strip.background = element_blank(),
strip.placement = 'outside'
)24.4 Projeções da incidência
EpiNow2
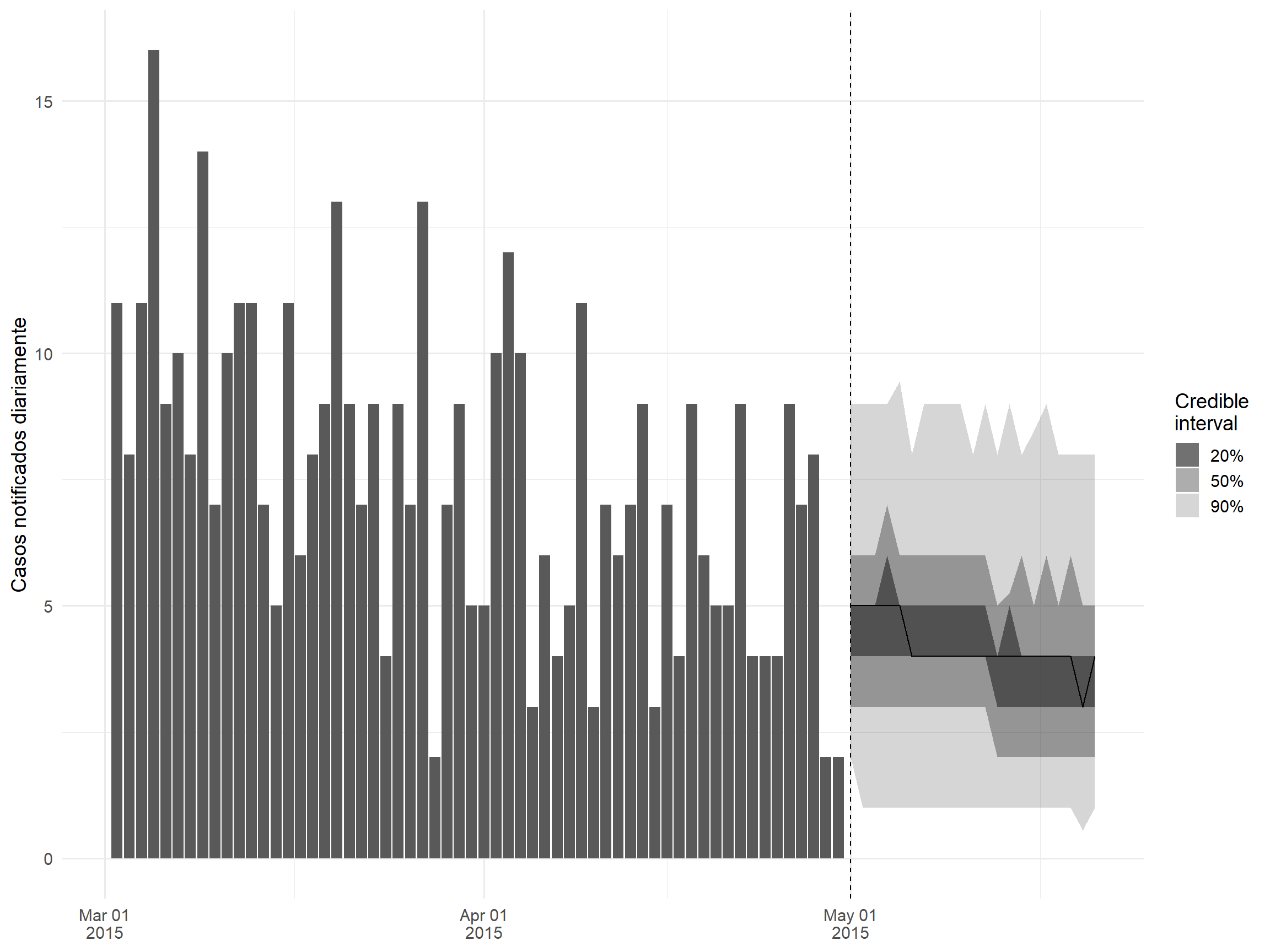
Além de estimar o Rt, EpiNow2 também é capaz de prever o Rt e projetar o número de casos ao ser integrado com o pacote EpiSoon. Tudo o que você precisa fazer é especificar o argumento horizon ao usar a função epinow, indicando quantos dias você quer projetar no futuro; veja a seção do EpiNow2 em “Estimando o Rt” para detalhes sobre como configurar e executar o EpiNow2. Nesta seção, nós iremos apenas fazer o gráfico dos resultados desta análise, salvos no objeto epinow_res.
## defina a data mínima do gráfico
min_date <- as.Date("2015-03-01")
## extraia as estimativas resumidas
estimates <- as_tibble(epinow_res$estimates$summarised)
## extraia os dados brutos da incidência de casos
observations <- as_tibble(epinow_res$estimates$observations) %>%
filter(date > min_date)
## extraia as estimativas previstas do número de caso
df_wide <- estimates %>%
filter(
variable == "reported_cases",
type == "forecast",
date > min_date
)
## converta para o formato longo para criar o gráfico de quantil
df_long <- df_wide %>%
## aqui combinamos os quantis correspondentes (ex.: lower_90 to upper_90)
pivot_longer(
lower_90:upper_90,
names_to = c(".value", "quantile"),
names_pattern = "(.+)_(.+)"
)
## crie o gráfico
ggplot() +
geom_histogram(
data = observations,
aes(x = date, y = confirm),
stat = 'identity',
binwidth = 1
) +
geom_ribbon(
data = df_long,
aes(x = date, ymin = lower, ymax = upper, alpha = quantile),
color = NA
) +
geom_line(
data = df_wide,
aes(x = date, y = median)
) +
geom_vline(xintercept = min(df_long$date), linetype = 2) +
## defina manualmente a transparência do quantil
scale_alpha_manual(
values = c(`20` = 0.7, `50` = 0.4, `90` = 0.2),
labels = function(x) paste0(x, "%")
) +
labs(
x = NULL,
y = "Casos notificados diariamente",
alpha = "Credible\ninterval"
) +
scale_x_date(
date_breaks = "1 month",
date_labels = "%b %d\n%Y"
) +
theme_minimal(base_size = 14)Pacote projections
O pacote projections, desenvolvido pela RECON, torna bem fácil o ato de prever incidências no curto prazo, requerindo apenas conhecimento do número efetivo de reprodução Rt, e o intervalo seriado. Aqui, nós iremos abordar como usar estimativas seriadas de intervalo da literatura e como usar nossas próprias estimativas baseadas na linelist.
Utilizando estimativas de intervalo seriado da literatura
O pacote projections precise de uma distribuição seriada discreta de intervalos da classe distcrete, do pacote distcrete. Nós iremos utilizar uma distribuição gamma com uma média de 12.0 e desvio padrão de 5.2 definido neste artigo. Para converter estes valores para os parâmetros de formato e escala requiridos para a distribuição gamma, iremos utilizar a função gamma_mucv2shapescale do pacote epitrix.
## obtenha os parâmetros de formato e escala da média mu e o coeficiente de
## variação (ex.: a razão do desvio padrão para a média)
shapescale <- epitrix::gamma_mucv2shapescale(mu = 12.0, cv = 5.2/12)
## crie um objeto do tipo *distcrete*
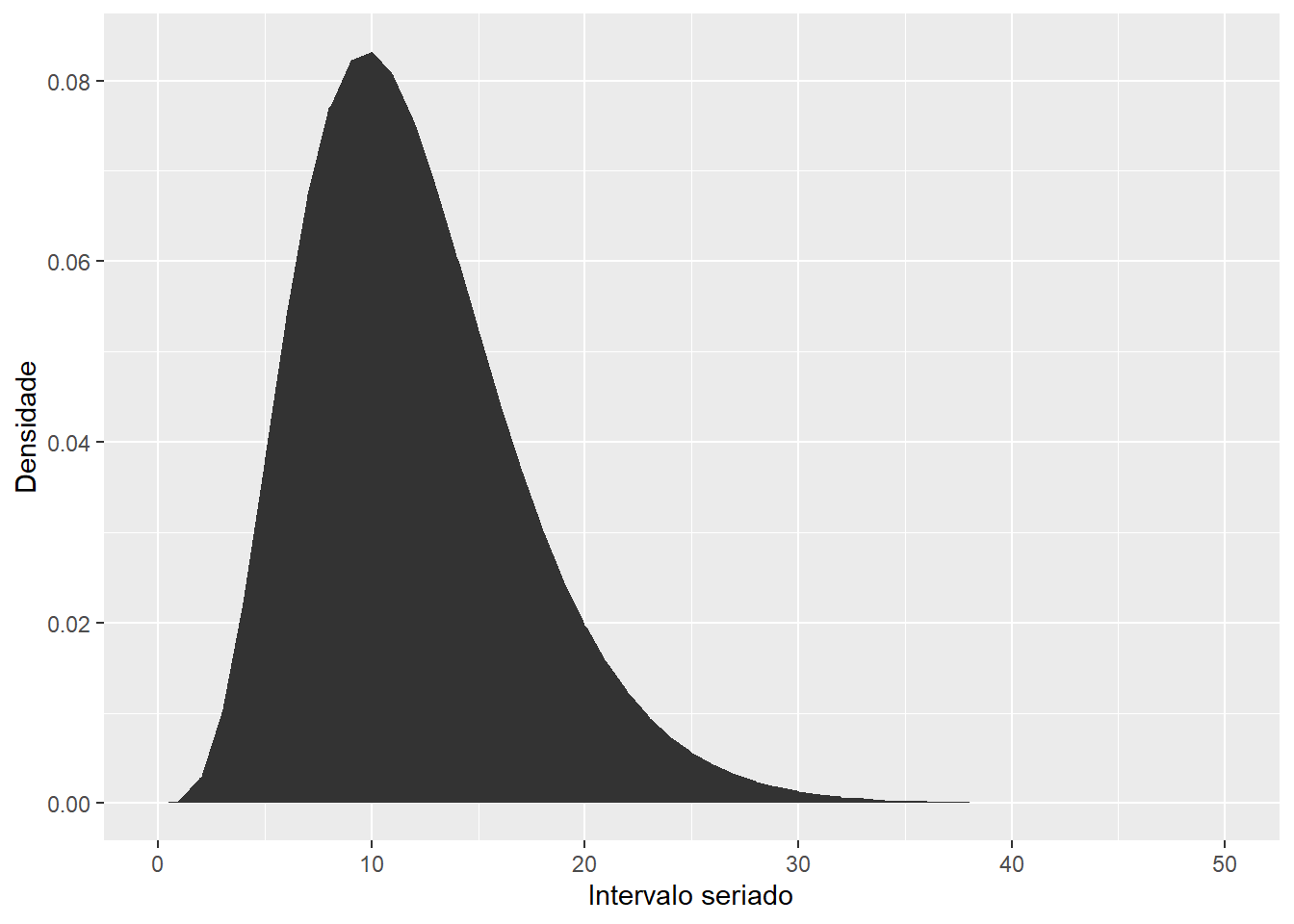
serial_interval_lit <- distcrete::distcrete(
name = "gamma",
interval = 1,
shape = shapescale$shape,
scale = shapescale$scale
)Aqui está uma checagem rápida para garantir que o intervalo seriado está correto. Nós acessamos a densidade da distribuição gamma que acabamos de definir com $d, que é equivalente a chamar dgamma:
Utilizando estivamitvas de intervalo seriadas a partir dos dados
Como temos dados com as datas de início dos sintomas e links de transmissão, nós podemos também estimar o intervalo seriado a partir da linelist ao calcular o intervalo entre as datas de início dos sintomas dos pares infectante-infectado. Como fizemos na seção do EpiNow2, nós iremos utilizar a função get_pairwise do pacote epicontacts, que nos permite calcular diferenças em pares das propriedades da linelist nos pares de transmissão. Primeiro, criamos um objeto epicontact (veja a página Cadeias de transmissão para mais detalhes):
## crie os contacts
contacts <- linelist %>%
transmute(
from = infector,
to = case_id
) %>%
drop_na()
## crie o objeto epicontacts
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
directed = TRUE
)Então ajustamos a diferença no início de sintomas entre os pares de transmissão, calculando usando get_pairwise, para uma distriuição gamma. Nós usamos a função fit_disc_gamma, do pacote epitrix, para realizar este procedimento de ajuste, uma vez que precisamos de uma distribuição discreta.
## estime o intervalo seriado gamma
serial_interval <- fit_disc_gamma(get_pairwise(epic, "date_onset"))
## inspecione a estimativa
serial_interval[c("mu", "sd")]$mu
[1] 11.51047
$sd
[1] 7.696056Projeções da incidência
Para prever a incidência futura, nós ainda precisamos fornecer a incidência histórica na forma de um objeto incidence, assim como uma amostra de valores plausíveis de Rt. Nós iremos gerar estes valores utilizando as estimativas de Rt geradas pelo EpiEstim na seção anterior (na subseção “Estimando Rt”) e salvo no objeto epiestim_res_emp. No código abaixo, nós extraímos a média e as estimativas de desvio padrão do Rt para a última janela de tempo do surto (usando a função tail para acessar o último elemento em um vetor), e simulamos 1000 valores de uma distribuição gamma utilizando rgamma. Você também pode fornecer seu própria vetor de valores Rt que você quer usar para projeções futuras.
## crie um objeto incidence a partir das datas de início dos sintomas
inc <- incidence::incidence(linelist$date_onset)256 missing observations were removed.## extraia valores plausíveis de r para maior parte das estimativas recentes
mean_r <- tail(epiestim_res_emp$R$`Mean(R)`, 1)
sd_r <- tail(epiestim_res_emp$R$`Std(R)`, 1)
shapescale <- gamma_mucv2shapescale(mu = mean_r, cv = sd_r/mean_r)
plausible_r <- rgamma(1000, shape = shapescale$shape, scale = shapescale$scale)
## cheque a distribuição
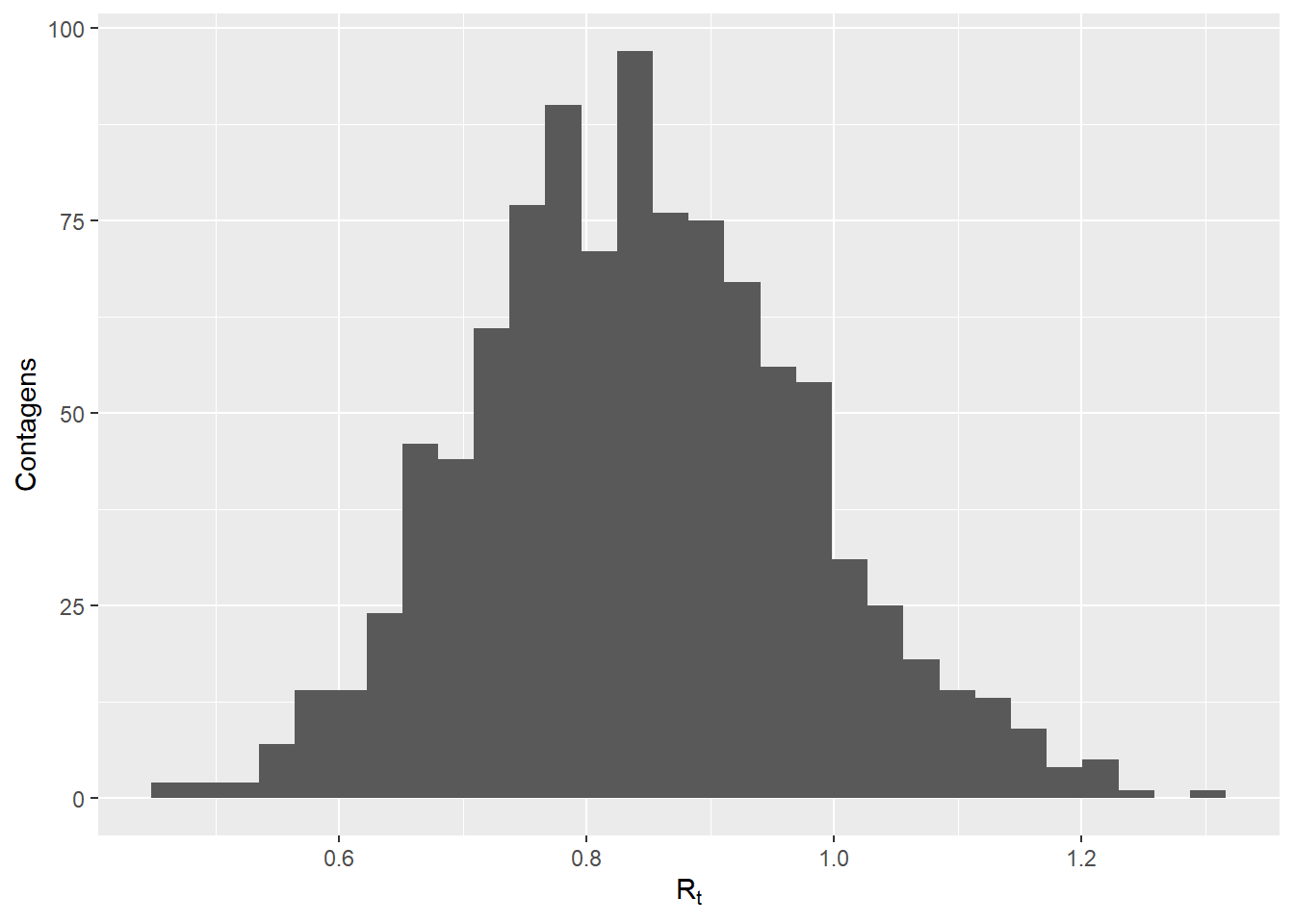
qplot(x = plausible_r, geom = "histogram", xlab = expression(R[t]), ylab = "Contagens")`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Nós então usamos a função project() para criar a previsão atual. Nós especificamos quantos dias queremos prever através dos argumentos n_days, e especificamos o número de simulações usando o argumento n_sim.
## crie a projeção
proj <- project(
x = inc,
R = plausible_r,
si = serial_interval$distribution,
n_days = 21,
n_sim = 1000
)Nós podemos, então, criar um gráfico da incidência e projeções usando as funções plot() e add_projections(). Nós podemos facilmente criar subconjuntos do objeto incidence para apenas mostrar os casos mais recentes ao utilizar o operador de colchetes retos.
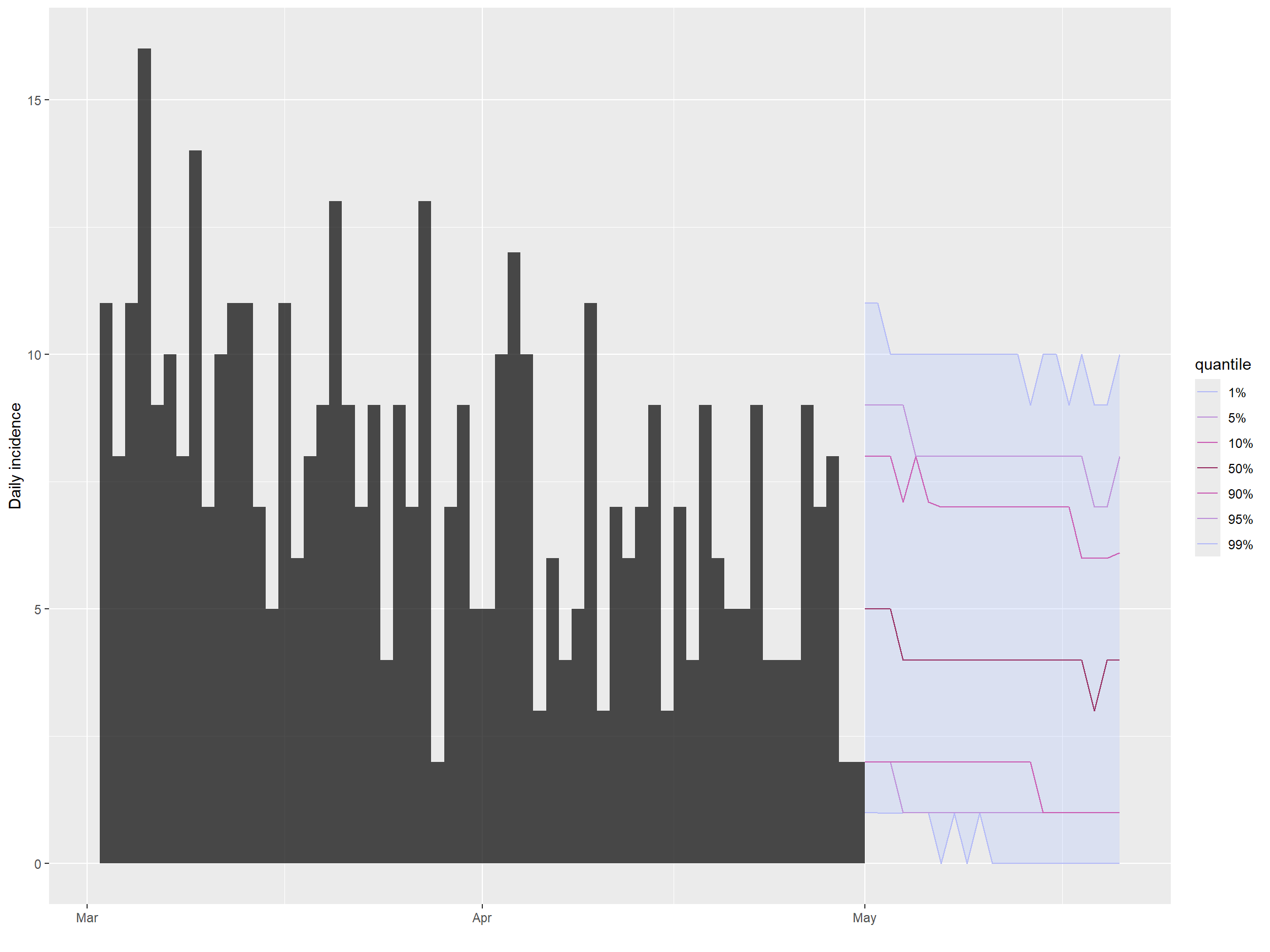
## crie um gráfico da incidência e projeções
plot(inc[inc$dates > as.Date("2015-03-01")]) %>%
add_projections(proj)Você pode também facilmente extrair as novas estimativas brutas do número diário de casos ao converter o resultado da análise para um quadro de dados.
## converta para um quadro de dados os dados brutos
proj_df <- as.data.frame(proj)
proj_df24.5 Recursos extras
- Aqui está o artigo descrevendo a metodologia empregada no EpiEstim.
- Aqui está o artigo descrevendo a metodologia implementada no EpiNow2.
- Aqui está um artigo descrevendo diferentes considerações dos metodológicas e práticas para estimar o Rt.